Применение радиомики для выявления остеопороза — текущие возможности и перспективы (научный обзор)
- Авторы: Чугаев А.И.1,2, Васильев Ю.А.1, Петряйкин А.В.1, Блохин И.А.1, Владзимирский А.В.1, Омелянская О.В.1
-
Учреждения:
- Научно-практический клинический центр диагностики и телемедицинских технологий
- «МРТ24»
- Выпуск: Том 6, № 1 (2025)
- Страницы: 63-77
- Раздел: Обзоры
- URL: https://ogarev-online.ru/DD/article/view/310053
- DOI: https://doi.org/10.17816/DD635014
- ID: 310053
Цитировать
Аннотация
Распространённость остеопоротических переломов продолжает увеличиваться по мере старения населения, происходящего по причине демографического перехода. Данная проблема актуальна для развитых стран, включая Российскую Федерацию. Радиомика в перспективе может стать хорошим инструментом для выявления остеопороза.
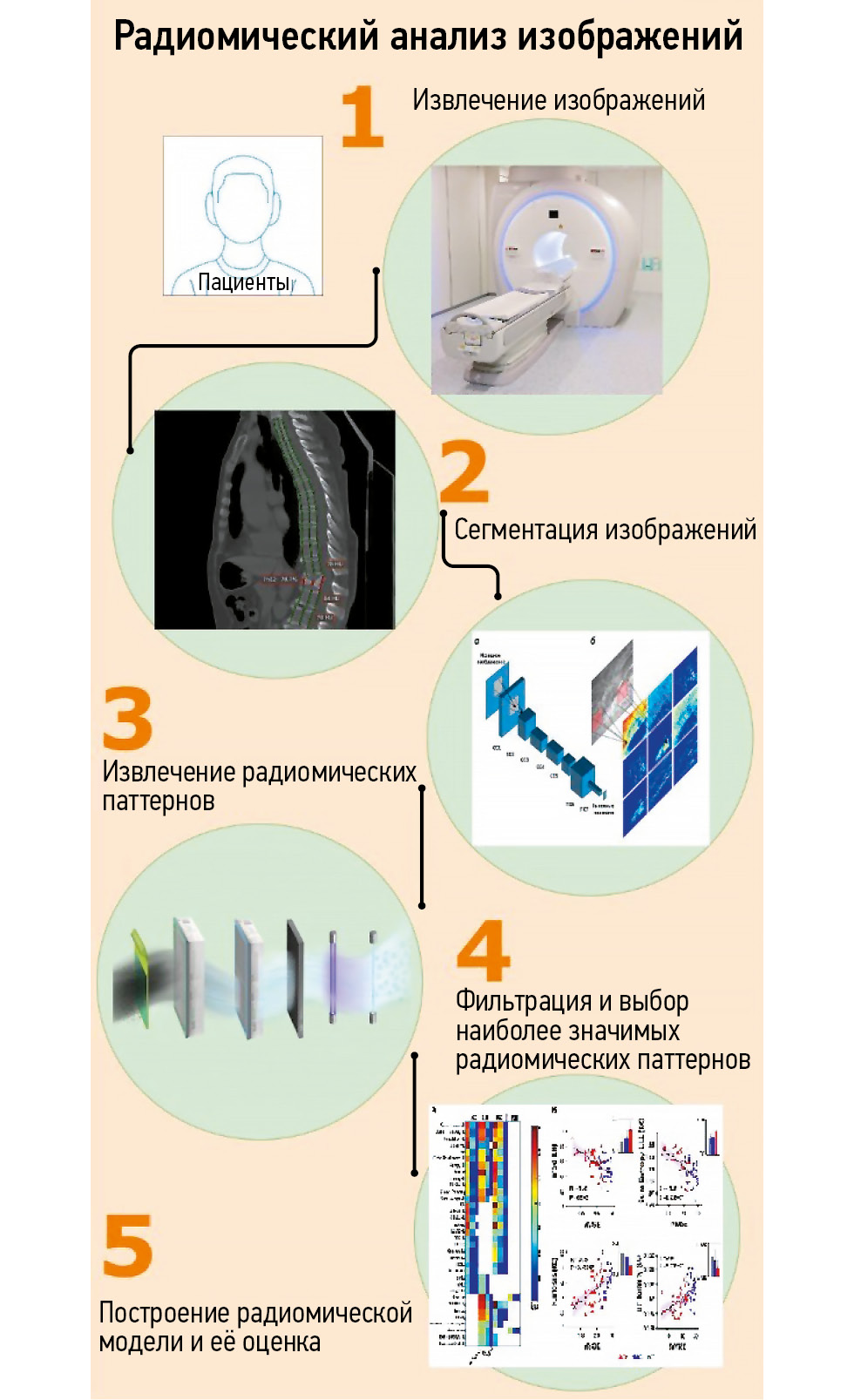
В обзоре продемонстрировано развитие и применение радиомического анализа в диагностике онкологических и неонкологических заболеваний, в частности — остеопороза.
Поиск литературы, соответствующий теме обзора, осуществляли с использованием поисковых систем, таких как PubMed, Google Schholar и eLibrary, за последние пять лет. Данные о распространённости и эпидемиологии остеопороза взяты из публикаций за последние пятнадцать лет. Поиск выполняли с использованием ключевых слов: «radiomic», «osteoporosis», «texture», «magnetic resonance imaging», «computed tomography», «non-oncological radiomics», «магнитно-резонансная томография», «компьютерная томография», «радиомика», «остеопороз», «текстурный анализ», «радиомический анализ». В обзор включены данные оригинальных клинических исследований. В результате найдено 247 статей, из которых в обзор после анализа публикаций отобрано 59 исследований.
Отмечено ограниченное количество работ, изучающих возможности радиомического анализа в отношении выявления остеопороза. Необходимо дальнейшее проведение исследований в области изучения потенциала радиомического анализа с использованием изображений компьютерной и магнитно-резонансной томографии в выявлении остеопороза в сравнении с признанными методиками — двухэнергетической рентгеновской абсорбциометрией и алгоритмом FRAX (Fracture Risk Assessment Tool).
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Антон Иванович Чугаев
Научно-практический клинический центр диагностики и телемедицинских технологий; «МРТ24»
Автор, ответственный за переписку.
Email: chugaev020379@yandex.ru
ORCID iD: 0009-0006-8930-9320
Россия, Москва; Москва
Юрий Александрович Васильев
Научно-практический клинический центр диагностики и телемедицинских технологий
Email: VasilevYA1@zdrav.mos.ru
ORCID iD: 0000-0002-5283-5961
SPIN-код: 4458-5608
канд. мед. наук
Россия, МоскваАлексей Владимирович Петряйкин
Научно-практический клинический центр диагностики и телемедицинских технологий
Email: alexeypetraikin@gmail.com
ORCID iD: 0000-0003-1694-4682
SPIN-код: 6193-1656
д-р мед. наук
Россия, МоскваИван Андреевич Блохин
Научно-практический клинический центр диагностики и телемедицинских технологий
Email: i.blokhin@npcmr.ru
ORCID iD: 0000-0002-2681-9378
SPIN-код: 3306-1387
канд. мед. наук
Россия, МоскваАнтон Вячеславович Владзимирский
Научно-практический клинический центр диагностики и телемедицинских технологий
Email: VladzimirskijAV@zdrav.mos.ru
ORCID iD: 0000-0002-2990-7736
SPIN-код: 3602-7120
д-р мед. наук
Россия, МоскваОльга Васильевна Омелянская
Научно-практический клинический центр диагностики и телемедицинских технологий
Email: OmelyanskayaOV@zdrav.mos.ru
ORCID iD: 0000-0002-0245-4431
SPIN-код: 8948-6152
Россия, Москва
Список литературы
- Kanis JA, Melton LJ, Christiansen C, et al. The diagnosis of osteoporosis. Journal of Bone and Mineral Research. 1994;9(8):1137–1141. doi: 10.1002/jbmr.5650090802
- Lesnyak OM, Baranova IA, Belova KYu, et al. Osteoporosis in Russian Federation: epidemiology, socio-medical and economical aspects (review). Traumatology and Orthopedics of Russia. 2018;24(1):155–168. doi: 10.21823/2311-2905-2018-24-1-155-168 EDN: YVGNSE
- Lesnyak O, Svedbom A, Belova K, et al. Quality of life after fragility fracture in the Russian Federation: results from the Russian arm of the International Cost and Utility Related to Osteoporotic Fractures Study (ICUROS). Archives of Osteoporosis. 2020;15(1):37. doi: 10.1007/s11657-020-0699-6 EDN: ZGZODH
- LeBoff MS, Greenspan SL, Insogna KL, et al. The clinician’s guide to prevention and treatment of osteoporosis. Osteoporos Int. 2022;33(10):2049–102. doi: 10.1007/s00198-021-05900-y Corrected and republished from: Osteoporos Int. 2022;33(10):2243. doi: 10.1007/s00198-022-06479-8
- van Staa TP, Dennison EM, Leufkens HGM, Cooper C. Epidemiology of fractures in England and Wales. Bone. 2001;29(6):517–522. doi: 10.1016/s8756-3282(01)00614-7 EDN: EJUXUV
- Wainwright SA, Marshall LM, Ensrud KE, et al. Hip fracture in women without osteoporosis. J Clin Endocrinol Metab. 2005;90(5):2787–2793. doi: 10.1210/jc.2004-1568
- Schuit SCE, van der Klift M, Weel AE, et al. Fracture incidence and association with bone mineral density in elderly men and women: the Rotterdam Study. Bone. 2004;34(1):195–202. doi: 10.1016/j.bone.2003.10.001 EDN: MDSZRJ Corrected and republished from: Bone. 2006;38(4):603.
- Siris ES, Chen YT, Abbott TA, et al. Bone mineral density thresholds for pharmacological intervention to prevent fractures. Arch Intern Med. 2004;164(10):1108–1112. doi: 10.1001/archinte.164.10.1108 EDN: XTVWZS
- Fink HA, Milavetz DL, Palermo L, et al. What proportion of incident radiographic vertebral deformities is clinically diagnosed and vice versa? J Bone Miner Res. 2005;20(7):1216–1222. doi: 10.1359/JBMR.050314
- Viswanathan M, Reddy S, Berkman N, et al. Screening to prevent osteoporotic fractures: updated evidence report and systematic review for the US preventive services task force. JAMA. 2018;319(24):2532–2551. doi: 10.1001/jama.2018.6537
- Greenspan SL, Singer A, Vujevich K, et al. Implementing a fracture liaison service open model of care utilizing a cloud-based tool. Osteoporos Int. 2018;29(4):953–960. doi: 10.1007/s00198-017-4371-y EDN: PBVQEQ
- Gillies RJ, Kinahan PE, Hricak H. Radiomics: images are more than pictures, they are data. Radiology. 2016;278(2):563–577. doi: 10.1148/radiol.2015151169
- Belaya ZhE, Belova KYu, Biryukova EV, et al. Federal clinical guidelines for diagnosis, treatment and prevention of osteoporosis. Osteoporosis and Bone Diseases. 2021;24(2):4–47. doi: 10.14341/osteo12930 EDN: TUONYE
- Genant HK, Wu CY, van Kuijk C, Nevitt MC. Vertebral fracture assessment using a semiquantitative technique. J Bone Miner Res. 1993;8(9):1137–1148. doi: 10.1002/jbmr.5650080915
- Lesnyak O, Bilezikian JP, Zakroyeva A, et al. Report on the audit on burden of osteoporosis in Eight Countries of the Eurasian Region: Armenia, Belarus, Georgia, Moldova, Kazakhstan, the Kyrgyz Republic, the Russian Federation, and Uzbekistan. Archives of Osteoporosis. 2020;15(1):175. doi: 10.1007/s11657-020-00836-y EDN: UNBZCB
- Wang X, Sanyal A, Cawthon PM, et al. Prediction of new clinical vertebral fractures in elderly men using finite element analysis of CT scans. J Bone Miner Res. 2012;27(4):808–816. doi: 10.1002/jbmr.1539
- Samelson EJ, Broe KE, Xu H, et al. Cortical and trabecular bone microarchitecture as an independent predictor of incident fracture risk in older women and men in the Bone Microarchitecture International Consortium (BoMIC): a prospective study. Lancet Diabetes Endocrinol. 2019;7(1):34–43. doi: 10.1016/S2213-8587(18)30308-5 Corrected and republished from: Lancet Diabetes Endocrinol. 2019;7(1):e1. doi: 10.1016/S2213-8587(18)30345-0 Corrected and republished from: Lancet Diabetes Endocrinol. 2019;7(6):e18. doi: 10.1016/S2213-8587(19)30140-8
- Vasilev YuA, Vladzymyrskyy AV, Artyukova ZR, et al. Diagnostics and screening of osteoporosis based on the results of computed tomography of the abdominal organs: guidelines. Moscow: Moscow Center for Diagnostics and Telemedicine; 2023. (In Russ.) EDN: DXUJZD
- Lin W, He C, Xie F, et al. Quantitative CT screening improved lumbar BMD evaluation in older patients compared to dual-energy X-ray absorptiometry. BMC Geriatr. 2023;23(1):231. doi: 10.1186/s12877-023-03963-6 EDN: IMPCNF
- Liu ZJ, Zhang C, Ma C, et al. Automatic phantom-less QCT system with high precision of BMD measurement for osteoporosis screening: technique optimisation and clinical validation. J Orthop Translat. 2022;33:24–30. doi: 10.1016/j.jot.2021.11.008 EDN: ZEKPFP
- Hossain SD, Petraikin AV, Muraev AA, et al. Bone mineral density radiopaque templates for cone beam computed tomography and multidetector computed tomography. Digital Diagnostics. 2023;4(3):292–305. doi: 10.17816/DD501771 EDN: KWYJXH
- Manhard MK, Nyman JS, Does MD. Advances in imaging approaches to fracture risk evaluation. Transl Res. 2017;181:1–14. doi: 10.1016/j.trsl.2016.09.006 EDN: YWDLBD
- Kanis JA, Hans D, Cooper C, et al. Interpretation and use of FRAX in clinical practice. Osteoporos Int. 2011;22(9):2395–2411. doi: 10.1007/s00198-011-1713-z EDN: YAEETY
- Adami G, Biffi A, Porcu G, et al. A systematic review on the performance of fracture risk assessment tools: FRAX, DeFRA, FRA-HS. J Endocrinol Invest. 2023;46(11):2287–2297. doi: 10.1007/s40618-023-02082-8 EDN: KGDPNO
- Nikitinskaya OA, Toroptsova NV. Assessment of fractures risk using the FRAX® tool (a ten-year retrospective study). Almanac of Clinical Medicine. 2016;(32):50–55. doi: 10.18786/2072-0505-2014-32-50-55 EDN: SXYJPF
- McCague C, Ramlee S, Reinius M, et al. Introduction to radiomics for a clinical audience. Clinical Radiology. 2023;78(2):83–98. doi: 10.1016/j.crad.2022.08.149 EDN: FTQEEU
- Haralick RM, Shanmugam K, Dinstein I. Textural Features for Image Classification. IEEE Trans Syst Man Cybern. 1973;SMC-3(6):610–621. doi: 10.1109/TSMC.1973.4309314
- Sutton RN, Hall EL. Texture measures for automatic classification of pulmonary disease. IEEE Transactions on Computers. 1972;C-21(7):667–676. doi: 10.1109/T-C.1972.223572
- Park H, Lim Y, Ko ES, et al. Radiomics signature on magnetic resonance imaging: association with disease-free survival in patients with invasive breast cancer. Clin Cancer Res. 2018;24(19):4705–14. doi: 10.1158/1078-0432.CCR-17-3783
- Yao Q, Liu M, Yuan K, et al. Radiomics nomogram based on dual-energy spectral CT imaging to diagnose low bone mineral density. BMC Musculoskeletal Disorders. 2022;23(1):424. doi: 10.1186/s12891-022-05389-4 EDN: ZJCBIU
- Rizzo S, Botta F, Raimondi S, et al. Radiomics: the facts and the challenges of image analysis. Eur Radiol Exp. 2018;2(1):36. doi: 10.1186/s41747-018-0068-z EDN: FCYFNJ
- Zwanenburg A, Vallières M, Abdalah MA, et al. The image biomarker standardization initiative: standardized quantitative radiomics for high-throughput image-based phenotyping. Radiology. 2020;295(2):328–38. doi: 10.1148/radiol.2020191145 EDN: HZVKJN
- Corrias G, Micheletti G, Barberini L, et al. Texture analysis imaging «what a clinical radiologist needs to know». Eur J Radiol. 2022;146(2):110055. doi: 10.1016/j.ejrad.2021.110055
- Court LE, Fave X, Mackin D, et al. Computational resources for radiomics. Transl Cancer Res. 2016;5(4):340–348. doi: 10.21037/tcr.2016.06.17 EDN: POXYYB
- van Timmeren JE, Cester D, Tanadini-Lang S, et al. Radiomics in medical imaging — «how-to» guide and critical reflection. Insights Imaging. 2020;11(1):91. doi: 10.1186/s13244-020-00887-2
- Wagner MW, Namdar K, Biswas A, et al. Radiomics, machine learning, and artificial intelligence — what the neuroradiologist needs to know. Neuroradiology. 2021;63(12):1957–1967. doi: 10.1007/s00234-021-02813-9 EDN: HJQKML
- Vial A, Stirling D, Field M, et al. The role of deep learning and radiomic feature extraction in cancer-specific predictive modelling: a review. Transl Cancer Res. 2018;7(3):803–816. doi: 10.21037/tcr.2018.05.02
- García Santos JM, Plasencia Martínez JM, Fabuel Ortega P, et al. Radiology departments as COVID-19 entry-door might improve healthcare efficacy and efficiency, and emergency department safety. Insights Imaging. 2021;12(1):1. doi: 10.1186/s13244-020-00954-8 EDN: TALMZP
- Ramspek CL, Jager KJ, Dekker FW, et al. External validation of prognostic models: what, why, how, when and where? Clin Kidney J. 2021;14(1):49–58. doi: 10.1093/ckj/sfaa188 EDN: LAPIBN
- Limkin EJ, Sun R, Dercle L, et al. Promises and challenges for the implementation of computational medical imaging (radiomics) in oncology. Ann Oncol. 2017;28(6):1191–206. doi: 10.1093/annonc/mdx034
- Santos AG, da Rocha GO, de Andrade JB. Occurrence of the potent mutagens 2- nitrobenzanthrone and 3-nitrobenzanthrone in fine airborne particles. Sci Rep. 2019;9(1):1. doi: 10.1038/s41598-018-37186-2
- Gooden MJ, de Bock GH, Leffers N, et al. The prognostic influence of tumour-infiltrating lymphocytes in cancer: a systematic review with meta-analysis. Br J Cancer. 2011;105(1):93–103. doi: 10.1038/bjc.2011.189
- Ren J, Tian J, Yuan Y, et al. Magnetic resonance imaging based radiomics signature for the preoperative discrimination of stage I-II and III-IV head and neck squamous cell carcinoma. Eur J Radiol. 201;106:1–6. doi: 10.1016/j.ejrad.2018.07.002
- Aerts HJ, Velazquez ER, Leijenaar RTH, et al. Decoding tumour phenotype by noninvasive imaging using a quantitative radiomics approach. Nat Commun. 2014;5(1):4006. doi: 10.1038/ncomms5006 Corrected and republished from: Nat Commun. 2014;5:4644.
- Granzier RWY, van Nijnatten TJA, Woodruff HC, et al. Exploring breast cancer response prediction to neoadjuvant systemic therapy using MRI-based radiomics: a systematic review. Eur J Radiol. 2019;121:108736. doi: 10.1016/j.ejrad.2019.108736 EDN: VXYGTU
- Shaish H, Aukerman A, Vanguri R, et al. Radiomics of MRI for pretreatment prediction of pathologic complete response, tumor regression grade, and neoadjuvant rectal score in patients with locally advanced rectal cancer undergoing neoadjuvant chemoradiation: an international multicenter study. Eur Radiol. 2020;30(11):6263–6273. doi: 10.1007/s00330-020-06968-6 EDN: UOKLLV
- Sun C, Tian X, Liu Z, et al. Radiomic analysis for pretreatment prediction of response to neoadjuvant chemotherapy in locally advanced cervical cancer: a multicentre study. EBioMedicine. 2019;46:160–169. doi: 10.1016/j.ebiom.2019.07.049
- Zhao L, Gong J, Xi Y, et al. MRI-based radiomics nomogram may predict the response to induction chemotherapy and survival in locally advanced nasopharyngeal carcinoma. Eur Radiol. 2020;30(1):537–546. doi: 10.1007/s00330-019-06211-x EDN: EIRSSZ
- Nardone V, Reginelli A, Grassi R, et al. Delta radiomics: a systematic review. Radiol Med. 2021;126(12):1571–1583. doi: 10.1007/s11547-021-01436-7 EDN: PWBARF
- Burian E, Subburaj K, Mookiah MRK, et al. Texture analysis of vertebral bone marrow using chemical shift encoding–based water-fat MRI: a feasibility study. Osteoporos Int. 2019;30(6):1265–1274. doi: 10.1007/s00198-019-04924-9 EDN: IVFDCX
- Kawashima Y, Fujita A, Buch K, et al. Using texture analysis of head CT images to differentiate osteoporosis from normal bone density. Eur J Radiol. 2019;116:212–218. doi: 10.1016/j.ejrad.2019.05.009
- Valentinitsch A, Trebeschi S, Kaesmacher J, et al. Opportunistic osteoporosis screening in multi-detector CT images via local classification of textures. Osteoporos Int. 2019;30(6):1275–1285. doi: 10.1007/s00198-019-04910-1 EDN: USTGTS
- White R, Krueger D, De Guio F, et al. An exploratory study of the texture research investigational platform (TRIP) to evaluate bone texture score of distal femur DXA scans – A TBS-based approach. J Clin Densitom. 2021;24(1):112–117. doi: 10.1016/j.jocd.2019.06.004 EDN: DQGTII
- Rastegar S, Vaziri M, Qasempour Y, et al. Radiomics for classification of bone mineral loss: a machine learning study. Diagnostic and Interventional Imaging. 2020;101(9):599–610. doi: 10.1016/j.diii.2020.01.008 EDN: MXTTFU
- Jiang YW, Xu XJ, Wang R, Chen CM. Radiomics analysis based on lumbar spine CT to detect osteoporosis. European Radiology. 2022;32(11):8019–8026. doi: 10.1007/s00330-022-08805-4 EDN: FFIEJM
- van Griethuysen JJM, Fedorov A, Parmar C, et al. Computational radiomics system to decode the radiographic phenotype. Cancer Res. 2017;77(21):e104–e107. doi: 10.1158/0008-5472.CAN-17-0339
- Steinhauer V, Sergeev NI. Radiomics in Breast Cancer: In-Depth Machine Analysis of MR Images of Metastatic Spine Lesion. Sovremennye tehnologii v medicine. 2022;14(2):16–25. doi: 10.17691/stm2022.14.2.02 EDN: XFVITL
- Kim S, Kim BR, Chae HD, et al. Deep radiomics–based approach to the diagnosis of osteoporosis using hip radiographs. Radiology: Artificial Intelligence. 2022;4(4):e210212. doi: 10.1148/ryai.210212 EDN: QKWOZX
- Wang J, Zhou S, Chen S, et al. Prediction of osteoporosis using radiomics analysis derived from single source dual energy CT. BMC Musculoskeletal Disorders. 2023;24(1):100. doi: 10.1186/s12891-022-06096-w EDN: CSYYUO
- Xie Q, Chen Y, Hu Y, et al. Development and validation of a machine learning-derived radiomics model for diagnosis of osteoporosis and osteopenia using quantitative computed tomography. BMC Medical Imaging. 2022;22(1):140. doi: 10.1186/s12880-022-00868-5 EDN: TBGMNV
- Martel D, Monga A, Chang G. Radiomic analysis of the proximal femur in osteoporosis women using 3T MRI. Front Radiol. 2023;3:1293865. doi: 10.3389/fradi.2023.1293865 EDN: CMHBMX
- Zhen T, Fang J, Hu D, et al. Comparative evaluation of multiparametric lumbar MRI radiomic models for detecting osteoporosis. BMC Musculoskelet Disord. 2024;25(1):185. doi: 10.1186/s12891-024-07309-0 EDN: JXNNRY
- Vasilev YA, Bobrovskaya TM, Arzamasov KM, et al. Medical datasets for machine learning: fundamental principles of standartization and systematization. Manager Zdravookhranenia. 2023;(4):28–41. doi: 10.21045/1811-0185-2023-4-28-41 EDN: EPGAMD
Дополнительные файлы