Диагностическая точность искусственного интеллекта для скрининга рака предстательной железы при бипараметрической магнитно-резонансной томографии: систематический обзор
- Авторы: Крючкова О.В.1, Щепкина Е.В.2,3,4, Рубцова Н.А.5, Алексеев Б.Я.5, Кузнецов А.И.6, Епифанова С.В.1,3, Заря Е.В.1, Талышинский А.Э.7
-
Учреждения:
- Центральная клиническая больница с поликлиникой Управления делами Президента Российской Федерации
- Российская академия народного хозяйства и государственной службы при Президенте Российской Федерации
- Научно-практический клинический центр диагностики и телемедицинских технологий
- Редакция журнала «Педиатрия» имени Г.Н. Сперанского
- Московский научно-исследовательский онкологический институт имени П.А. Герцена — филиал ФГБУ Национального медицинского исследовательского центра Радиологии
- Московский авиационный институт
- Санкт-Петербургский государственный университет
- Выпуск: Том 5, № 3 (2024)
- Страницы: 534-550
- Раздел: Систематические обзоры
- URL: https://ogarev-online.ru/DD/article/view/310036
- DOI: https://doi.org/10.17816/DD626643
- ID: 310036
Цитировать
Полный текст
Аннотация
Обоснование. По последним опубликованным данным, в 2021 г. в России зарегистрировано 40 137 новых случаев рака предстательной железы, что ставит его на второе место среди онкологических заболеваний в мужской популяции после рака лёгкого.
Таким образом, рак предстательной железы является одним из наиболее распространённых злокачественных новообразований у мужчин. В этих условиях точное и своевременное выявление рака предстательной железы приобретает особую важность.
Цель настоящего систематического обзора с метаанализом — оценка качества прогностических моделей, построенных для выявления рака предстательной железы при первичном обращении.
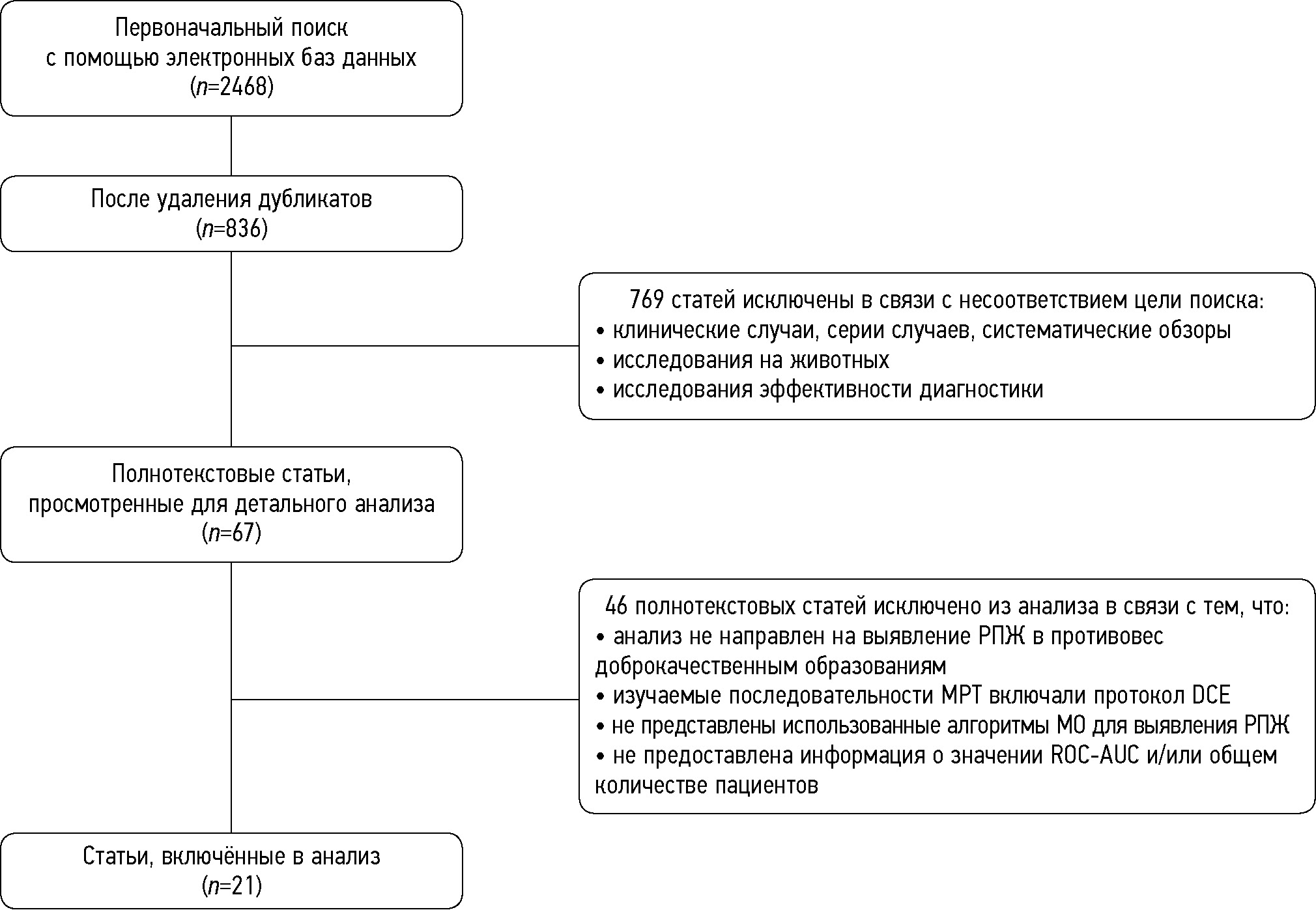
Материалы и методы. Систематический поиск публикаций проводили в специализированных поисковых системах научной информации eLibrary.ru, PubMed, Google Scholar, Web of Science и ResearchGate с использованием установленных методов в соответствии с протоколом PRISMA. Для анализа отбирали статьи, опубликованные в период с января 2019 г. по сентябрь 2023 г. Два автора независимо отбирали публикации в соответствии с критериями включения и исключения.
Результаты. Этот систематический обзор с метаанализом включает 21 исследование. В общей сложности в них участвовало 3630 пациентов, из них 47% пациентов с раком предстательной железы и 53% с доброкачественными образованиями предстательной железы. Возраст пациентов в среднем составил 67,1 года (в диапазоне от 36 до 90 лет). Из всех исследований, в 81% случаев использовался протокол Т2-взвешенных изображений, в 57% — диффузионно-взвешенных изображений, и в 76% — измеряемых коэффициентов диффузии. Кроме того, 43% исследований изучали образования в переходной зоне, 33% — в периферической зоне предстательной железы. В 52% случаев авторы проводили исследование в целом по органу, без разделения на зоны. Анализ показал, что наиболее часто исследователи используют следующие алгоритмы машинного обучения: MLR (Multiple Logistic Regression) — 76%, SVM (Support Vector Machine) — 38%, и RF (Random Forest) — 24%. По данным проведённого метаанализа оценки ROC-AUC в 73 прогностических моделях, описанных в изученных нами публикациях, с использованием метода случайных эффектов, было получено итоговое значение ROC-AUC 0,793 (95% CI; 0,768–0,818), I2=86,71%, p <0,001. Модели, построенные на последовательностях Т2-взвешенных изображений + измеряемых коэффициентов диффузии (ROC-AUC 0,860 95%CI 0,813–0,907), а также модели, построенные по принципу «белый ящик» (ROC-AUC 0.834 95%CI 0.806–0.861), оказались наиболее точными, по сравнению с построенными по принципу «чёрный ящик» (ROC-AUC 0,733 95%CI 0,695–0,771). Модели, использующие радиомические и клинические признаки, были несколько точнее, чем построенные исключительно на радиомических признаках: ROC-AUC 0,869 (95%CI 0,844–0,895) против 0,779 (95%CI 0,751–0,807). Точность моделей практически не различалась по зоне исследования (переходная или периферическая).
Заключение. Результаты многообещающие, но клиническая применимость по-прежнему требует более тщательной проверки со стороны экспертов в медицинских учреждениях и оценки эффективности в проспективных исследованиях.
Полный текст
Открыть статью на сайте журналаОб авторах
Оксана Валентиновна Крючкова
Центральная клиническая больница с поликлиникой Управления делами Президента Российской Федерации
Email: ovk16@bk.ru
ORCID iD: 0000-0001-6483-2074
SPIN-код: 2445-3370
канд. мед. наук
Россия, МоскваЕлена Викторовна Щепкина
Российская академия народного хозяйства и государственной службы при Президенте Российской Федерации; Научно-практический клинический центр диагностики и телемедицинских технологий; Редакция журнала «Педиатрия» имени Г.Н. Сперанского
Автор, ответственный за переписку.
Email: elenaschepkina@gmail.com
ORCID iD: 0000-0002-2079-1482
SPIN-код: 2347-9436
Scopus Author ID: 57211515165
ResearcherId: IAR-4060-2023
канд. социол. наук
Россия, Москва; Москва; МоскваНаталья Алефтиновна Рубцова
Московский научно-исследовательский онкологический институт имени П.А. Герцена — филиал ФГБУ Национального медицинского исследовательского центра Радиологии
Email: rna17@ya.ru
ORCID iD: 0000-0001-8378-4338
SPIN-код: 9712-9091
д-р мед. наук
Россия, МоскваБорис Яковлевич Алексеев
Московский научно-исследовательский онкологический институт имени П.А. Герцена — филиал ФГБУ Национального медицинского исследовательского центра Радиологии
Email: byalekseev@mail.ru
ORCID iD: 0000-0002-3398-4128
SPIN-код: 4692-5705
д-р мед. наук
Россия, МоскваАнтон Игоревич Кузнецов
Московский авиационный институт
Email: drednout5786@yandex.ru
ORCID iD: 0000-0003-2182-5792
SPIN-код: 8824-9080
Россия, Москва
Светлана Викторовна Епифанова
Центральная клиническая больница с поликлиникой Управления делами Президента Российской Федерации; Научно-практический клинический центр диагностики и телемедицинских технологий
Email: svepifanova@yandex.ru
ORCID iD: 0000-0002-7591-5120
SPIN-код: 9067-5033
канд. мед. наук
Россия, Москва; МоскваЕлена Владимировна Заря
Центральная клиническая больница с поликлиникой Управления делами Президента Российской Федерации
Email: zaryya@yandex.ru
ORCID iD: 0009-0001-4444-8881
SPIN-код: 9800-8219
Россия, Москва
Али Эльманович Талышинский
Санкт-Петербургский государственный университет
Email: ali-ma@mail.ru
ORCID iD: 0000-0002-3521-8937
SPIN-код: 7747-0117
д-р мед. наук
Россия, Санкт-ПетербургСписок литературы
- Mottet N., van den Bergh R.C.N., Briers E., et al. EAU-EANM-ESTRO-ESUR-SIOG Guidelines on Prostate Cancer–2020 Update. Part 1: Screening, Diagnosis, and Local Treatment with Curative Intent // European Urology. 2021. Vol. 79, N 2. P. 243–262. doi: 10.1016/j.eururo.2020.09.042
- Здравоохранение в России. 2021 : статистический сборник. Москва : Росстат, 2021.
- Verma S., Rajesh A. A Clinically Relevant Approach to Imaging Prostate Cancer: review // American Journal of Roentgenology. 2011. Vol. 196, Suppl. 3. P. S1–10 Quiz S11–4. doi: 10.2214/AJR.09.7196
- Girometti R., Giannarini G., Panebianco V., et al. Comparison of different thresholds of PSA density for risk stratification of PI-RADSv2.1 categories on prostate MRI // The British Journal of Radiology. 2022. Vol. 95, N 1131. P. 20210886. doi: 10.1259/bjr.20210886
- Niaf E., Lartizien C., Bratan F., et al. Prostate Focal Peripheral Zone Lesions: Characterization at Multiparametric MR Imaging–Influence of a Computer-aided Diagnosis System // Radiology. 2014. Vol. 271, N 3. P. 761–769. doi: 10.1148/radiol.14130448
- Drost F.-J.H., Osses D.F., Nieboer D., et al. Prostate MRI, with or without MRI-targeted biopsy, and systematic biopsy for detecting prostate cancer // Cochrane Database of Systematic Reviews. 2019. Vol. 4, N 4. P. CD012663. doi: 10.1002/14651858.CD012663.pub2
- Goldenberg S.L., Nir G., Salcudean S.E. A new era: artificial intelligence and machine learning in prostate cancer // Nature Reviews Urology. 2019. Vol. 16, N 7. P. 391–403. doi: 10.1038/s41585-019-0193-3
- Cuocolo R., Cipullo M.B., Stanzione A., et al. Machine learning applications in prostate cancer magnetic resonance imaging // European Radiology Experimental. 2019. Vol. 3, N 1. P. 35. doi: 10.1186/s41747-019-0109-2
- Ghezzo S., Bezzi C., Presotto L., et al. State of the art of radiomic analysis in the clinical management of prostate cancer: A systematic review // Critical Reviews in Oncology/Hematology. 2022. Vol. 169. P. 103544. doi: 10.1016/j.critrevonc.2021.103544
- Гележе П.Б., Блохин И.А., Семенов С.С., Caruso D. Радиомика магнитно-резонансной томографии при раке предстательной железы: что известно в настоящее время? // Digital Diagnostics. 2021. Т. 2, №4. C. 441–452. doi: 10.17816/DD70170
- Ferro M., de Cobelli O., Vartolomei M.D., et al. Prostate Cancer Radiogenomics–From Imaging to Molecular Characterization // International Journal of Molecular Sciences. 2021. Vol. 22, N 18. P. 9971. doi: 10.3390/ijms22189971
- Steyerberg E.W., Vickers A.J., Cook N.R., et al. Assessing the Performance of Prediction Models // Epidemiology. 2010. Vol. 21, N 1. P. 128–138. doi: 10.1097/EDE.0b013e3181c30fb2
- Higgins J.P.T., Green S., editors. The Cochrane Collaboration. Cochrane Handbook for Systematic Reviews of Interventions [Internet]. England : John Wiley & Sons Ltd. Дата обращения: 19.03.2020. Режим доступа: https://training.cochrane.org/handbook
- Higgins J.P.T., Thompson S.G., Deeks J.J., Altman D.G. Measuring inconsistency in meta-analyses // BMJ. 2003. Vol. 327, N 7414. P. 557–560. doi: 10.1136/bmj.327.7414.557
- DerSimonian R., Laird N. Meta-analysis in clinical trials // Controlled Clinical Trials. 1986. Vol. 7, N 3. P. 177–188. doi: 10.1016/0197-2456(86)90046-2
- Woźnicki P., Westhoff N., Huber T., et al. Multiparametric MRI for Prostate Cancer Characterization: Combined Use of Radiomics Model with PI-RADS and Clinical Parameters // Cancers (Basel). 2020. Vol. 12, N 7. P. 1767. doi: 10.3390/cancers12071767
- Li M., Yang L., Yue Y., et al. Use of Radiomics to Improve Diagnostic Performance of PI-RADS v2.1 in Prostate Cancer // Frontiers in Oncology. 2021. Vol. 10. P. 631831. doi: 10.3389/fonc.2020.631831
- Gui S., Lan M., Wang C., et al. Application Value of Radiomic Nomogram in the Differential Diagnosis of Prostate Cancer and Hyperplasia // Frontiers in Oncology. 2022. Vol. 12. P. 859625. doi: 10.3389/fonc.2022.859625
- Lu Y., Li B., Huang H., et al. Biparametric MRI-based radiomics classifiers for the detection of prostate cancer in patients with PSA serum levels of 4~10 ng/mL // Frontiers in Oncology. 2022. Vol. 12. P. 1020317. doi: 10.3389/fonc.2022.1020317
- Zhou B., Liu X., Gan H., et al. Differentiation of Prostate Cancer and Stromal Hyperplasia in the Transition Zone With Monoexponential, Stretched-Exponential Diffusion-Weighted Imaging and Diffusion Kurtosis Imaging in a Reduced Number of b Values: Correlation With Whole-Mount Pathology // Journal of Computer Assisted Tomography. 2022. Vol. 46, N 4. P. 545–550. doi: 10.1097/RCT.0000000000001314
- Wu M., Krishna S., Thornhill R.E., et al. Transition zone prostate cancer: Logistic regression and machine-learning models of quantitative ADC, shape and texture features are highly accurate for diagnosis // Journal of Magnetic Resonance Imaging. 2019. Vol. 50, N 3. P. 940–950. doi: 10.1002/jmri.26674
- Zhong J.-G., Shi L., Liu J., et al. Predicting prostate cancer in men with PSA levels of 4–10 ng/mL: MRI-based radiomics can help junior radiologists improve the diagnostic performance // Scientific reports. 2023. Vol. 13, N 1. P. 4846. doi: 10.1038/s41598-023-31869-1
- Ou Y.C., Chang K.H., Tung M.C., et al. Building a Nomogram for Prediction of Prostate Cancer in Patients With Preoperatively Suspected Prostate Cancer // Anticancer Research. 2020. Vol. 40, N 5. P. 2995–3002. doi: 10.21873/anticanres.14280
- McGarry S.D., Bukowy J.D., Iczkowski K.A., et al. Gleason Probability Maps: A Radiomics Tool for Mapping Prostate Cancer Likelihood in MRI Space // Tomography. 2019. Vol. 5, N 1. P. 127–134. doi: 10.18383/j.tom.2018.00033
- Hu L. Zhou D.W., Fu C.X., et al. Advanced zoomed diffusion-weighted imaging vs. full-field-of-view diffusion-weighted imaging in prostate cancer detection: a radiomic features study // European radiology. 2021. Vol. 31, N 3. P. 1760–1769. doi: 10.1007/s00330-020-07227-4
- Ji X., Zhang J., Shi W., et al. Bi-parametric magnetic resonance imaging based radiomics for the identification of benign and malignant prostate lesions: cross-vendor validation // Physical and Engineering Sciences in Medicine. 2021. Vol. 44, N 3. P. 745–754. doi: 10.1007/s13246-021-01022-1
- Jin P., Shen J., Yang L., et al. Machine learning-based radiomics model to predict benign and malignant PI-RADS v2.1 category 3 lesions: a retrospective multi-center study // BMC Medical Imaging. 2023. Vol. 23, N 1. P. 47. doi: 10.1186/s12880-023-01002-9
- Li S., Zheng T., Fan Z., et al. A dynamic-static combination model based on radiomics features for prostate cancer using multiparametric MRI // Physics in Medicine & Biology. 2023. Vol. 68, N 1. P. 015008. doi: 10.1088/1361-6560/aca954
- Ayyad S.M., Badawy M.A., Shehata M., et al. A New Framework for Precise Identification of Prostatic Adenocarcinoma // Sensors. 2022. Vol. 22, N 5. P. 1848. doi: 10.3390/s22051848
- Han L., He G., Mei Y., et al. Combining Magnetic Resonance Diffusion-Weighted Imaging with Prostate-Specific Antigen to Differentiate Between Malignant and Benign Prostate Lesions // Medical Science Monitor. 2022. Vol. 28. P. e935307. doi: 10.12659/MSM.935307
- Chen T., Li M., Gu Y., et al. Prostate Cancer Differentiation and Aggressiveness: Assessment With a Radiomic-Based Model vs. PI-RADS v2 // Journal of Magnetic Resonance Imaging. 2019. Vol. 49, N 3. P. 875–884. doi: 10.1002/jmri.26243
- He D., Wang X., Fu C., et al. MRI-based radiomics models to assess prostate cancer, extracapsular extension and positive surgical margins // Cancer Imaging. 2021. Vol. 21, N 1. P. 46. doi: 10.1186/s40644-021-00414-6
- Jamshidi G., Abbasian Ardakani A., Ghafoori M., et al. Radiomics-based machine-learning method to diagnose prostate cancer using mp-MRI: a comparison between conventional and fused models // Magnetic Resonance Materials in Physics, Biology and Medicine. 2022. Vol. 36, N 1. P. 55–64. doi: 10.1007/s10334-022-01037-z
- Aussavavirojekul P., Hoonlor A., Srinualnad S. Optimization of clinical risk-factor interpretation and radiological findings with machine learning for PIRADS category 3 patients // Prostate. 2022. Vol. 82, N 2. P. 235–244. doi: 10.1002/pros.24266
- Giambelluca D., Cannella R., Vernuccio F., et al. PI-RADS 3 Lesions: Role of Prostate MRI Texture Analysis in the Identification of Prostate Cancer // Current Problems in Diagnostic Radiology. 2021. Vol. 50, N 2. P. 175–185. doi: 10.1067/j.cpradiol.2019.10.009
- Viswanath S.E., Chirra P.V., Yim M.C., et al. Comparing radiomic classifiers and classifier ensembles for detection of peripheral zone prostate tumors on T2-weighted MRI: a multi-site study // BMC Medical Imaging. 2019. Vol. 19, N 1. P. 22. doi: 10.1186/s12880-019-0308-6
- Chawla N.V., Bowyer K.W., Hall L.O., Kegelmeyer W.P. SMOTE: Synthetic Minority Over-sampling Technique // Journal of Artificial Intelligence Research. 2002. Vol. 16, N 1. P. 321–357.
- Dai J.C., Morgan T.N., Goueli R., et al. MRI Features Associated with Histology of Benign Prostatic Hyperplasia Nodules: Generation of a Predictive Model // Journal of Endourology. 2022. Vol. 36, N 3. P. 381–386. doi: 10.1089/end.2021.0397
- Liu J., Dong B., Qu W., et al. Using clinical parameters to predict prostate cancer and reduce the unnecessary biopsy among patients with PSA in the gray zone // Scientific reports. 2020. Vol. 10, N 1. P. 5157. doi: 10.1038/s41598-020-62015-w
- Zhang L., Tang M., Chen S., et al. A meta-analysis of use of Prostate Imaging Reporting and Data System Version 2 (PI-RADS V2) with multiparametric MR imaging for the detection of prostate cancer // European radiology. 2017. Vol. 27, N 12. P. 5204–5214. doi: 10.1007/s00330-017-4843-7
- Zhen L., Liu X., Yegang C., et al. Accuracy of multiparametric magnetic resonance imaging for diagnosing prostate Cancer: a systematic review and meta-analysis // BMC Cancer. 2019. Vol. 19, N 1. P. 1244. doi: 10.1186/s12885-019-6434-2
Дополнительные файлы