Сравнительный анализ полногеномных последовательностей изолятов вируса африканской чумы свиней (Asfarviridae: Asfivirus), выделенных на территории левобережья Днепра в 2023 году
- Авторы: Чернышев Р.С.1, Иголкин А.С.1, Зиняков Н.Г.1, Чвала И.А.1
-
Учреждения:
- ФГБУ «Федеральный центр охраны здоровья животных»
- Выпуск: Том 69, № 5 (2024)
- Страницы: 481-494
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://ogarev-online.ru/0507-4088/article/view/269837
- DOI: https://doi.org/10.36233/0507-4088-263
- EDN: https://elibrary.ru/tsiuzd
- ID: 269837
Цитировать
Аннотация
Введение. Отсутствие данных о полногеномных последовательностях возбудителя африканской чумы свиней (АЧС), циркулирующего на территории левобережья Днепра, ограничивает понимание динамики молекулярной эволюции вируса и характера развития текущего эпизоотического процесса в центральной России и Украине. Определение степени генетической дивергентности и филогенетического родства вируса АЧС во многом может скорректировать стратегию общей и специфической профилактики болезни.
Цель работы – поиск и описание уникальных точечных мутаций (делеций/инсерций/замен) у изолятов, выделенных от домашних свиней на территории Донецкого, Луганского и Запорожского регионов в 2023 г.; установление родства и уровня гомологии с референтными штаммами вируса АЧС генотипа II; субгенотипирование на основе маркерных областей генома.
Материалы и методы. В качестве образцов использовали культуральную суспензию костного мозга свиньи, содержащую вирус АЧС. Подготовку геномной ДНК выполняли методом очистки и концентрирования вируса с последующей экстракцией тотальной нуклеиновой кислоты фенол-хлороформным методом. Процесс высокопроизводительного секвенирования осуществляли с помощью технологии MGI. Сборку консенсусных последовательностей проводили методом картирования прочтений на референс-геном штамма Georgia 2007/1.
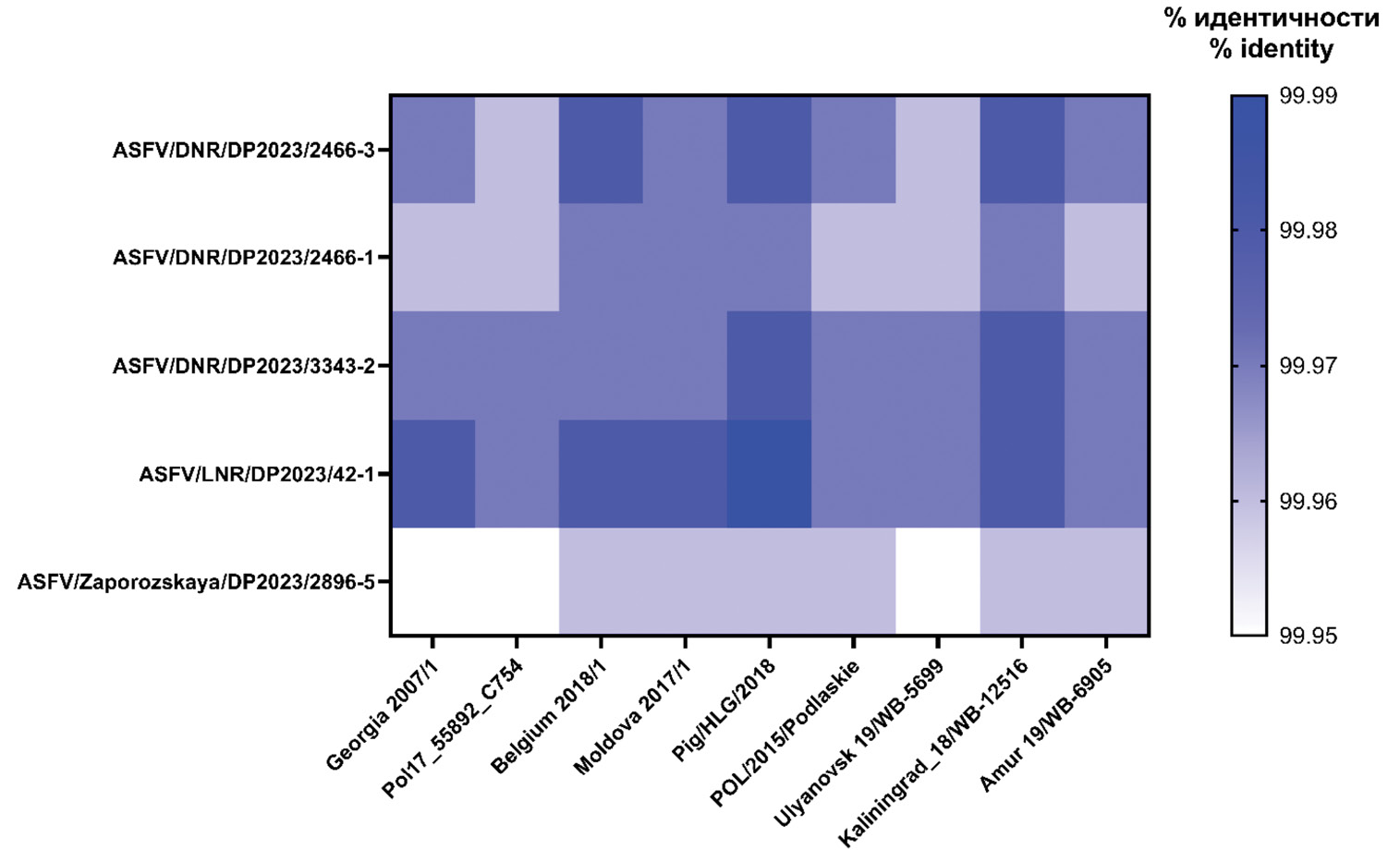
Результаты. Все изоляты отнесены к генотипу II, имеют монофилетическое происхождение, филогенетически относятся к кластерам «Европа» (4/5) и «Брянск 2021» (1/5), а также являются дивергентными от исходных родительских генетических вариантов, составляющих укрупненные клады. Кроме того, обнаружены многочисленные замены в локусах мультигенного семейства MGF 110, 505 и 360, кодирующих факторы вирулентности.
Заключение. На примере изучения филогении показана достоверная для дифференциации вариабельность вируса АЧС генотипа II, происходящего от референс-штамма Georgia 2007/1. Представленные данные обладают теоретической и практической значимостью при усовершенствовании национального и международного надзора за АЧС.
Полный текст
Открыть статью на сайте журналаОб авторах
Роман Сергеевич Чернышев
ФГБУ «Федеральный центр охраны здоровья животных»
Автор, ответственный за переписку.
Email: chernishev_rs@arriah.ru
ORCID iD: 0000-0003-3604-7161
аспирант, ветеринарный врач референтной лаборатории по африканской чуме свиней
Россия, 600901, г. ВладимирАлексей Сергеевич Иголкин
ФГБУ «Федеральный центр охраны здоровья животных»
Email: igolkin_as@arriah.ru
ORCID iD: 0000-0002-5438-8026
канд. вет. наук, заместитель руководителя центра – заведующий референтной лабораторией по африканской чуме свиней лабораторно-диагностического центра
Россия, 600901, г. ВладимирНиколай Геннадьевич Зиняков
ФГБУ «Федеральный центр охраны здоровья животных»
Email: zinyakov@arriah.ru
ORCID iD: 0000-0002-3015-5594
канд. биол. наук, ведущий научный сотрудник референтной лаборатории вирусных болезней птиц
Россия, 600901, г. ВладимирИлья Александрович Чвала
ФГБУ «Федеральный центр охраны здоровья животных»
Email: chvala@arriah.ru
ORCID iD: 0000-0002-1659-3256
канд. вет. наук, заместитель директора
Россия, 600901, г. ВладимирСписок литературы
- Beltrán-Alcrudo D., Lubroth J., Depner K., Rocque S. African swine fever in the Caucasus. EMPRES Watch. 2008; 1(8): 1–8. https://doi.org/10.13140/RG.2.1.3579.1200
- Nix R.J., Gallardo C., Hutchings G., Blanco E., Dixon L.K. Molecular epidemiology of African swine fever virus studied by analysis of four variable genome regions. Arch. Virol. 2006; 151(12): 2475–94. https://doi.org/10.1007/s00705-006-0794-z
- Shen Z.J., Jia H., Xie C.D., Shagainar J., Feng Z., Zhang X., et al. Bayesian phylodynamic analysis reveals the dispersal patterns of African swine fever virus. Viruses. 2022; 14(5): 889. https://doi.org/10.3390/v14050889
- Malogolovkin A., Yelsukova A., Gallardo C., Tsybanov S., Kolbasov D. Molecular characterization of African swine fever virus isolates originating from outbreaks in the Russian Federation between 2007 and 2011. Vet. Microbiol. 2012; 158(3-4): 415–9. https://doi.org/10.1016/j.vetmic.2012.03.002
- Chapman D.A., Darby A.C., Da Silva M., Upton C., Radford A.D., Dixon L.K. Genomic analysis of highly virulent Georgia 2007/1 isolate of African swine fever virus. Emerg. Infect. Dis. 2011; 17(4): 599–605. https://doi.org/10.3201/eid1704.101283
- Mazloum A., van Schalkwyk A., Shotin A., Igolkin A., Shevchenko I., Gruzdev K.N., et al. Comparative analysis of full genome sequences of African swine fever virus isolates taken from wild boars in Russia in 2019. Pathogens. 2021; 10(5): 521. https://doi.org/10.3390/pathogens10050521
- Чернышев Р.С., Спрыгин А.В., Шотин А.Р., Иголкин А.С., Мазлум А. Сравнительный анализ полногеномных последовательностей двух изолятов вируса африканской чумы свиней, выделенных в 2020 году от домашних и диких свиней в Забайкальском крае Российской Федерации. Ветеринария, зоотехния и биотехнология. 2022; (10): 84–97. https://doi.org/10.36871/vet.zoo.bio.202210010 https://elibrary.ru/qcgsux
- Zhang Y., Wang Q., Zhu Z., Wang S., Tu S., Zhang Y., et al. Tracing the origin of genotype II African swine fever virus in China by genomic epidemiology analysis. Transbound. Emerg. Dis. 2023; (1): 4820809. https://doi.org/10.1155/2023/4820809
- Xin G., Kuang Q., Le S., Wu W., Gao Q., Gao H., et al. Origin, genomic diversity and evolution of African swine fever virus in East Asia. Virus Evol. 2023; 9(2): vead060. https://doi.org/10.1093/ve/vead060
- Kovalenko G., Ducluzeau A.L., Ishchenko L., Sushko M., Sapachova M., Rudova N., et al. Complete genome sequence of a virulent African swine fever virus from a domestic pig in Ukraine. Microbiol. Resour. Announc. 2019; 8(42): e00883–19. https://doi.org/10.1128/MRA.00883-19
- Puzankova O., Gavrilova V., Chernyshev R., Kolbin I., Igolkin A., Sprygin A., et al. Novel protocol for the preparation of porcine bone marrow primary cell culture for African swine fever virus isolation. Methods Protoc. 2023; 6(5): 73. https://doi.org/10.3390/mps6050073
- Sun X., Hu Y.H., Wang J., Fang C., Li J., Han M., et al. Efficient and stable metabarcoding sequencing data using a DNBSEQ-G400 sequencer validated by comprehensive community analyses. GigaByte. 2021; 2021: gigabyte16. https://doi.org/10.46471/gigabyte.16
- Tcherepanov V., Ehlers A., Upton C. Genome Annotation Transfer Utility (GATU): rapid annotation of viral genomes using a closely related reference genome. BMC Genomics. 2006; 7: 150. https://doi.org/10.1186/1471-2164-7-150.
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms. Mol. Biol. Evol. 2018; 35(6): 1547–9. https://doi.org/10.1093/molbev/msy096
- Gallardo C., Casado N., Soler A., Djadjovski I., Krivko L., Madueño E., et al. A multi gene-approach genotyping method identifies 24 genetic clusters within the genotype II-European African swine fever viruses circulating from 2007 to 2022. Front. Vet. Sci. 2023; (10): 1112850. https://doi.org/10.3389/fvets.2023.1112850
- Чернышев Р.С., Иголкин А.С., Шотин А.Р., Зиняков Н.Г., Колбин И.С., Садчикова А.С. и др. Пространственно-временной кластерный анализ циркуляции вируса африканской чумы свиней (Asfarviridae: Asfivirus) в Калининградской области на основе трех генетических маркеров. Вопросы вирусологии. 2024; 69(3): 241–54. https://doi.org/10.36233/0507-4088-231 https://elibrary.ru/lbevpz
- Sun E., Huang L., Zhang X., Zhang J., Shen D., Zhang Z., et al. Genotype I African swine fever viruses emerged in domestic pigs in China and caused chronic infection. Emerg. Microbes Infect. 2021; 10(1): 2183–93. https://doi.org/10.1080/22221751.2021.1999779
- Zhao D., Sun E., Huang L., Ding L., Zhu Y., Zhang J., et al. Highly lethal genotype I and II recombinant African swine fever viruses detected in pigs. Nat. Commun. 2023; 14(1): 3096. https://doi.org/10.1038/s41467-023-38868-w
- Zhu Z., Chen H., Liu L., Cao Y., Jiang T., Zou Y., et al. Classification and characterization of multigene family proteins of African swine fever viruses. Brief. Bioinform. 2021; 22(4): bbaa380. https://doi.org/10.1093/bib/bbaa380
- Балышев В.М., Болгова М.В., Балышева В.И., Болгова М.В., Князева М.В., Живодеров С.П. Получение типовых задерживающих гемадсорбцию референс-сывороток к вирусу африканской чумы свиней. Вопросы нормативно-правового регулирования в ветеринарии. 2015; (2): 23–5. https://elibrary.ru/twnfvt
- Wu L., Yang B., Yuan X., Hong J., Peng M., Chen J.L., et al. Regulation and evasion of host immune response by African swine fever virus. Front. Microbiol. 2021; 12: 698001. https://doi.org/0.3389/fmicb.2021.698001
- Redrejo-Rodríguez M., Rodríguez J.M., Suárez C., Salas J., Salas M.L. Involvement of the reparative DNA polymerase Pol X of African swine fever virus in the maintenance of viral genome stability in vivo. J. Virol. 2013; 87(17): 9780–7. https://doi.org/10.1128/JVI.01173-13
- Chen S., Zhang X., Nie Y., Li H., Chen W., Lin W., et al. African swine fever virus protein E199L promotes cell autophagy through the interaction of PYCR2. Virol. Sin. 2021; 36(2): 196–206. https://doi.org/10.1007/s12250-021-00375-x
Дополнительные файлы