Defective HIV proviruses: possible involvement in the HIV infection pathogenesis
- Authors: Bobkova M.R.1
-
Affiliations:
- I. Mechnikov Research Institute for Vaccines and Sera
- Issue: Vol 69, No 5 (2024)
- Pages: 399-414
- Section: REVIEWS
- URL: https://ogarev-online.ru/0507-4088/article/view/269829
- DOI: https://doi.org/10.36233/0507-4088-261
- EDN: https://elibrary.ru/pselci
- ID: 269829
Cite item
Abstract
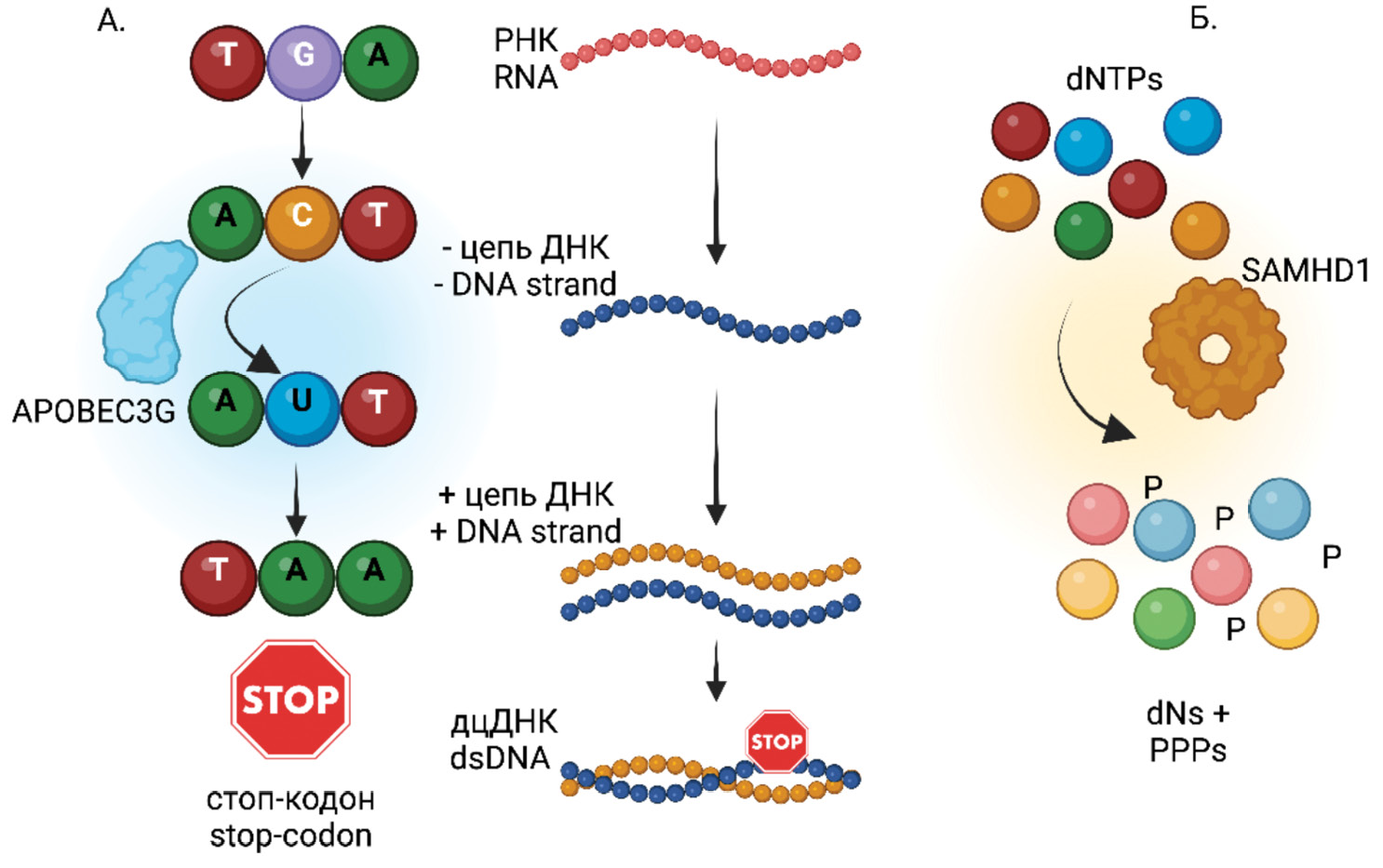
This review article analyzes information obtained from a literature search on defective HIV genomes (HIV-1, Human Immunodeficiency Virus, Lentivirus, Orthoretrovirinae, Retroviridae). It discusses the origins of defective HIV genomes, their potential for transcription and translation, and the role of defective RNA and proteins in stimulating both innate and adaptive immunity. The article also explores their contribution to HIV pathogenesis, immune system hyperactivation despite successful antiretroviral therapy (ART), and the evolutionary processes in HIV proviral populations under ART. Additionally, it addresses challenges in reservoir elimination and HIV eradication that arise from the existence of defective HIV viruses.
Full Text
##article.viewOnOriginalSite##About the authors
Marina R. Bobkova
I. Mechnikov Research Institute for Vaccines and Sera
Author for correspondence.
Email: mrbobkova@mail.ru
ORCID iD: 0000-0001-5481-8957
D.Sci. (Biol.), Chief Researcher of the laboratory of biology of lentiviruses
Russian Federation, 105064, MoscowReferences
- Bobkova M.R. HIV infection cure strategies: basic methodological approaches and difficulties of their implementation. VICH-infektsiya i immunosupressii. 2020; 12(1): 22–31. https://doi.org/10.22328/2077-9828-2020-12-1-22-31 https://elibrary.ru/gsllxf (in Russian)
- Bobkova M.R. HIV Latency [Latentnost’ VICh]. Moscow: Chelovek; 2021. (in Russian)
- Grund B., Baker J.V., Deeks S.G., Wolfson J., Wentworth D., Cozzi-Lepri A., et al. Relevance of interleukin-6 and D-dimer for serious non-AIDS morbidity and death among HIV-positive adults on suppressive antiretroviral therapy. PLoS One. 2016; 11(5): e0155100. https://doi.org/10.1371/journal.pone.0155100
- Singh K., Natarajan V., Dewar R., Rupert A., Badralmaa Y., Zhai T., et al. Long-term persistence of transcriptionally active ‘defective’ HIV-1 proviruses: implications for persistent immune activation during antiretroviral therapy. AIDS. 2023; 37(14): 2119–30. https://doi.org/10.1097/qad.0000000000003667
- Trickey A., May M.T., Vehreschild J., Obel N., Gill M.J., Crane H., et al. Cause-specific mortality in HIV-positive patients who survived ten years after starting antiretroviral therapy. PLoS One. 2016; 11(8): e0160460. https://doi.org/10.1371/journal.pone.0160460
- Bandera A., Colella E., Rizzardini G., Gori A., Clerici M. Strategies to limit immune-activation in HIV patients. Expert Rev. Anti Infect. Ther. 2017; 15(1): 43–54. https://doi.org/10.1080/14787210.2017.1250624
- Elvstam O., Medstrand P., Jansson M., Isberg P.E., Gisslén M., Björkman P. Is low-level HIV-1 viraemia associated with elevated levels of markers of immune activation, coagulation and cardiovascular disease? HIV Med. 2019; 20(9): 571–80. https://doi.org/10.1111/hiv.12756
- Utay N.S., Hunt P.W. Role of immune activation in progression to AIDS. Curr. Opin. HIV AIDS. 2016; 11(2): 131–7. https://doi.org/10.1097/coh.0000000000000242
- Younas M., Psomas C., Reynes C., Cezar R., Kundura L., Portalès P., et al. Residual viremia is linked to a specific immune activation profile in HIV-1-infected adults under efficient antiretroviral therapy. Front. Immunol. 2021; 12: 663843. https://doi.org/10.3389/fimmu.2021.663843
- Fombellida-Lopez C., Berkhout B., Darcis G., Pasternak A.O. Persistent HIV-1 transcription during ART: time to reassess its significance? Curr. Opin. HIV AIDS. 2024; 19(3): 124–32. https://doi.org/10.1097/coh.0000000000000849
- Kuniholm J., Armstrong E., Bernabe B., Coote C., Berenson A., Patalano S.D., et al. Intragenic proviral elements support transcription of defective HIV-1 proviruses. PLoS Pathog. 2021; 17(12): e1009982. https://doi.org/10.1371/journal.ppat.1009982
- Marchetti G., Tincati C., Silvestri G. Microbial translocation in the pathogenesis of HIV infection and AIDS. Clin. Microbiol. Rev. 2013; 26(1): 2–18. https://doi.org/10.1128/cmr.00050-12
- Freeman M.L., Lederman M.M., Gianella S. Partners in Crime: The Role of CMV in immune dysregulation and clinical outcome during HIV infection. Curr. HIV/AIDS Rep. 2016; 13(1): 10–9. https://doi.org/10.1007/s11904-016-0297-9
- Sherman B.T., Hu X., Singh K., Haine L., Rupert A.W., Neaton J.D., et al. Genome-wide association study of high-sensitivity C-reactive protein, D-dimer, and interleukin-6 levels in multiethnic HIV+ cohorts. AIDS. 2021; 35(2): 193–204. https://doi.org/10.1097/qad.0000000000002738
- Shirley D.K., Kaner R.J., Glesby M.J. Effects of smoking on non-AIDS-related morbidity in HIV-infected patients. Clin. Infect. Dis. 2013; 57(2): 275–82. https://doi.org/10.1093/cid/cit207
- Rapid Response Service. Low-level HIV viremia: Definitions, predictors, mechanisms, and clinical outcomes. Toronto, ON: The Ontario HIV Treatment Network; 2022. Available at: https://www.ohtn.on.ca/wp-content/uploads/2022/01/RR166_Low-level-viremia_version2.pdf
- Wu F., Simonetti F.R. Learning from persistent viremia: mechanisms and implications for clinical care and HIV-1 cure. Curr. HIV/AIDS Rep. 2023; 20(6): 428–39. https://doi.org/10.1007/s11904-023-00674-w
- Bobkova M.R. Low-level viremia in HIV infection: causes and consequences. VICh-infektsiya i immunosupressii. 2024; 16(2): 7–22. https://doi.org/10.22328/2077-9828-2024-16-2-7-22 https://elibrary.ru/zlmcgr (in Russian)
- Genoyer E., Lopez C.B. The impact of defective viruses on infection and immunity. Annu. Rev. Virol. 2019; 6(1): 547–66. https://doi.org/10.1146/annurev-virology-092818-015652
- Wang H., Cui X., Cai X., An T. Recombination in positive-strand RNA viruses. Front. Microbiol. 2022; 13: 870759. https://doi.org/10.3389/fmicb.2022.870759
- Sanchez G., Xu X., Chermann J.C., Hirsch I. Accumulation of defective viral genomes in peripheral blood mononuclear cells of human immunodeficiency virus type 1-infected individuals. J. Virol. 1997; 71(3): 2233–40. https://doi.org/10.1128/jvi.71.3.2233-2240.1997
- Imamichi H., Dewar R.L., Adelsberger J.W., Rehm C.A., O’Doherty U., Paxinos E.E., et al. Defective HIV-1 proviruses produce novel protein-coding RNA species in HIV-infected patients on combination antiretroviral therapy. Proc. Natl Acad. Sci. USA. 2016; 113(31): 8783–8. https://doi.org/10.1073/pnas.1609057113
- Kilroy J.M., Leal A.A., Henderson A.J. Chronic HIV transcription, translation, and persistent inflammation. Viruses. 2024; 16(5): 751. https://doi.org/10.3390/v16050751
- Berkhout B., van Wamel J., Klaver B. Requirements for DNA strand transfer during reverse transcription in mutant HIV-1 virions. J. Mol. Biol. 1995; 252(1): 59–69. https://doi.org/10.1006/jmbi.1994.0475
- Ho Y.C., Shan L., Hosmane N.N., Wang J., Laskey S.B., Rosenbloom D.I., et al. Replication-competent noninduced proviruses in the latent reservoir increase barrier to HIV-1 cure. Cell. 2013; 155(3): 540–51. https://doi.org/10.1016/j.cell.2013.09.020
- López CB. Defective viral genomes: critical danger signals of viral infections. J. Virol. 2014; 88(16): 8720–3. https://doi.org/10.1128/jvi.00707-14
- Vignuzzi M., Lopez C.B. Defective viral genomes are key drivers of the virus-host interaction. Nat. Microbiol. 2019; 4(7): 1075–87. https://doi.org/10.1038/s41564-019-0465-y
- Kuniholm J., Coote C., Henderson A.J. Defective HIV-1 genomes and their potential impact on HIV pathogenesis. Retrovirology. 2022; 19(1): 13. https://doi.org/10.1186/s12977-022-00601-8
- Reeves D.B., Gaebler C., Oliveira T.Y., Peluso M.J., Schiffer J.T., Cohn L.B., et al. Impact of misclassified defective proviruses on HIV reservoir measurements. Nat. Commun. 2023; 14(1): 4186. https://doi.org/10.1038/s41467-023-39837-z
- Bobkova M.R. Cellular proteins as potential targets for antiretroviral therapy. Voprosy virusologii. 2023; 68(6): 488–504. https://doi.org/10.36233/0507-4088-207 https://elibrary.ru/klgwak (in Russian)
- Hadpech S., Moonmuang S., Chupradit K., Yasamut U., Tayapiwatana C. Updating on roles of HIV intrinsic factors: a review of their antiviral mechanisms and emerging functions. Intervirology. 2022; 65(2): 67–79. https://doi.org/10.1159/000519241
- Ramdas P., Sahu A.K., Mishra T., Bhardwaj V., Chande A. From entry to egress: strategic exploitation of the cellular processes by HIV-1. Front. Microbiol. 2020; 11: 559792. https://doi.org/10.3389/fmicb.2020.559792
- Nchioua R., Bosso M., Kmiec D., Kirchhoff F. Cellular factors targeting HIV-1 transcription and viral RNA transcripts. Viruses. 2020; 12(5): 495. https://doi.org/10.3390/v12050495
- Ghimire D., Rai M., Gaur R. Novel host restriction factors implicated in HIV-1 replication. J. Gen. Virol. 2018; 99(4): 435–46. https://doi.org/10.1099/jgv.0.001026
- Colomer-Lluch M., Ruiz A., Moris A., Prado J.G. Restriction factors: from intrinsic viral restriction to shaping cellular immunity against HIV-1. Front. Immunol. 2018; 9: 2876. https://doi.org/10.3389/fimmu.2018.02876
- Schaller T., Herold N. The early bird catches the worm – can evolution teach us lessons in fighting HIV? Curr. HIV Res. 2016; 14(3): 183–210. https://doi.org/10.2174/1570162x14999160224094914
- Bedwell G.J., Engelman A.N. Factors that mold the nuclear landscape of HIV-1 integration. Nucleic Acids Res. 2021; 49(2): 621–35. https://doi.org/10.1093/nar/gkaa1207
- Craigie R., Bushman F.D. HIV DNA integration. Cold Spring Harb. Perspect. Med. 2012; 2(7): a006890. https://doi.org/10.1101/cshperspect.a006890
- Rodgers K., McVey M. Error-prone repair of DNA double-strand breaks. J. Cell. Physiol. 2016; 231(1): 15–24. https://doi.org/10.1002/jcp.25053
- Wiegand A., Spindler J., Hong F.F., Shao W., Cyktor J.C., Cillo A.R., et al. Single-cell analysis of HIV-1 transcriptional activity reveals expression of proviruses in expanded clones during ART. Proc. Natl Acad. Sci. USA. 2017; 114(18): E3659–68. https://doi.org/10.1073/pnas.1617961114
- Dutilleul A., Rodari A., Van Lint C. Depicting HIV-1 transcriptional mechanisms: a summary of what we know. Viruses. 2020; 12(12): 1385. https://doi.org/10.3390/v12121385
- Ding P., Kharytonchyk S., Waller A., Mbaekwe U., Basappa S., Kuo N., et al. Identification of the initial nucleocapsid recognition element in the HIV-1 RNA packaging signal. Proc. Natl Acad. Sci. USA. 2020; 117(30): 17737–46. https://doi.org/10.1073/pnas.2008519117
- Ishizaka A., Sato H., Nakamura H., Koga M., Kikuchi T., Hosoya N., et al. Short intracellular HIV-1 transcripts as biomarkers of residual immune activation in patients on antiretroviral therapy. J. Virol. 2016; 90(12): 5665–76. https://doi.org/10.1128/jvi.03158-15
- Sertznig H., Hillebrand F., Erkelenz S., Schaal H., Widera M. Behind the scenes of HIV-1 replication: Alternative splicing as the dependency factor on the quiet. Virology. 2018; 516: 176–88. https://doi.org/10.1016/j.virol.2018.01.011
- Mancarella A., Procopio F.A., Achsel T., De Crignis E., Foley B.T., Corradin G., et al. Detection of antisense protein (ASP) RNA transcripts in individuals infected with human immunodeficiency virus type 1 (HIV-1). J. Gen. Virol. 2019; 100(5): 863–76. https://doi.org/10.1099/jgv.0.001244
- Imamichi H., Smith M., Adelsberger J.W., Izumi T., Scrimieri F., Sherman B.T., et al. Defective HIV-1 proviruses produce viral proteins. Proc. Natl Acad. Sci. USA. 2020; 117(7): 3704–10. https://doi.org/10.1073/pnas.1917876117
- Vanhee-Brossollet C., Thoreau H., Serpente N., D’Auriol L., Levy J.P., Vaquero C. A natural antisense RNA derived from the HIV-1 env gene encodes a protein which is recognized by circulating antibodies of HIV+ individuals. Virology. 1995; 206(1): 196–202. https://doi.org/10.1016/s0042-6822(95)80034-4
- Pollack R.A., Jones R.B., Pertea M., Bruner K.M., Martin A.R., Thomas A.S., et al. Defective HIV-1 proviruses are expressed and can be recognized by cytotoxic T lymphocytes, which shape the proviral landscape. Cell Host Microbe. 2017; 21(4): 494–506.e4. https://doi.org/10.1016/j.chom.2017.03.008
- Decout A., Katz J.D., Venkatraman S., Ablasser A. The cGAS-STING pathway as a therapeutic target in inflammatory diseases. Nat. Rev. Immunol. 2021; 21(9): 548–69. https://doi.org/10.1038/s41577-021-00524-z
- Unterholzner L., Keating S.E., Baran M., Horan K.A., Jensen S.B., Sharma S., et al. IFI16 is an innate immune sensor for intracellular DNA. Nat. Immunol. 2010; 11(11): 997–1004. https://doi.org/10.1038/ni.1932
- Wu B., Hur S. How RIG-I like receptors activate MAVS. Curr. Opin. Virol. 2015; 12: 91–8. https://doi.org/10.1016/j.coviro.2015.04.004
- Ferdin J., Goričar K., Dolžan V., Plemenitaš A., Martin J.N., Peterlin B.M., et al. Viral protein Nef is detected in plasma of half of HIV-infected adults with undetectable plasma HIV RNA. PLoS One. 2018; 13(1): e0191613. https://doi.org/10.1371/journal.pone.0191613
- Fenwick C., Joo V., Jacquier P., Noto A., Banga R., Perreau M., et al. T-cell exhaustion in HIV infection. Immunol. Rev. 2019; 292(1): 149–63. https://doi.org/10.1111/imr.12823
- Verdon D.J., Mulazzani M., Jenkins M.R. Cellular and molecular mechanisms of CD8(+) T cell differentiation, dysfunction and exhaustion. Int. J. Mol. Sci. 2020; 21(19): 7357. https://doi.org/10.3390/ijms21197357
- Lichterfeld M., Gao C., Yu X.G. An ordeal that does not heal: understanding barriers to a cure for HIV-1 infection. Trends Immunol. 2022; 43(8): 608–16. https://doi.org/10.1016/j.it.2022.06.002
- Roux H., Chomont N. Measuring human immunodeficiency virus reservoirs: do we need to choose between quantity and quality? J. Infect. Dis. 2024; 229(3): 635–43. https://doi.org/10.1093/infdis/jiad381
- Halvas E.K., Joseph K.W., Brandt L.D., Guo S., Sobolewski M.D., Jacobs J.L., et al. HIV-1 viremia not suppressible by antiretroviral therapy can originate from large T cell clones producing infectious virus. J. Clin. Invest. 2020; 130(11): 5847–57. https://doi.org/10.1172/jci138099
- Virgilio M.C., Collins K.L. The impact of cellular proliferation on the HIV-1 reservoir. Viruses. 2020; 12(2): 127. https://doi.org/10.3390/v12020127
- White J.A., Wu F., Yasin S., Moskovljevic M., Varriale J., Dragoni F., et al. Clonally expanded HIV-1 proviruses with 5’-leader defects can give rise to nonsuppressible residual viremia. J. Clin. Invest. 2023; 133(6): e165245. https://doi.org/10.1172/jci165245
- Bui J.K., Sobolewski M.D., Keele B.F., Spindler J., Musick A., Wiegand A., et al. Proviruses with identical sequences comprise a large fraction of the replication-competent HIV reservoir. PLoS Pathog. 2017; 13(3): e1006283. https://doi.org/10.1371/journal.ppat.1006283
- Maldarelli F., Wu X., Su L., Simonetti F.R., Shao W., Hill S., et al. HIV latency. Specific HIV integration sites are linked to clonal expansion and persistence of infected cells. Science. 2014; 345(6193): 179–83. https://doi.org/10.1126/science.1254194
- Linden N., Jones R.B. Potential multi-modal effects of provirus integration on HIV-1 persistence: lessons from other viruses. Trends Immunol. 2022; 43(8): 617–29. https://doi.org/10.1016/j.it.2022.06.001
- Mohammadi A., Etemad B., Zhang X., Li Y., Bedwell G.J., Sharaf R., et al. Viral and host mediators of non-suppressible HIV-1 viremia. Nat. Med. 2023; 29(12): 3212–23. https://doi.org/10.1038/s41591-023-02611-1
- Crespo-Bermejo C., de Arellano E.R., Lara-Aguilar V., Valle-Millares D., Gomez-Lus M.L., Madrid R., et al. Persistent low-Level viremia in persons living with HIV undertreatment: An unresolved status. Virulence. 2021; 12(1): 2919–31. https://doi.org/10.1080/21505594.2021.2004743
- Sannier G., Dubé M., Dufour C., Richard C., Brassard N., Delgado G.G., et al. Combined single-cell transcriptional, translational, and genomic profiling reveals HIV-1 reservoir diversity. Cell Rep. 2021; 36(9): 109643. https://doi.org/10.1016/j.celrep.2021.109643
- Brodin J., Zanini F., Thebo L., Lanz C., Bratt G., Neher R.A., et al. Establishment and stability of the latent HIV-1 DNA reservoir. Elife. 2016; 5: e18889. https://doi.org/10.7554/elife.18889
- Lian X., Gao C., Sun X., Jiang C., Einkauf K.B., Seiger K.W., et al. Signatures of immune selection in intact and defective proviruses distinguish HIV-1 elite controllers. Sci. Transl. Med. 2021; 13(624): eabl4097. https://doi.org/10.1126/scitranslmed.abl4097
- Abrahams M.R., Joseph S.B., Garrett N., Tyers L., Moeser M., Archin N., et al. The replication-competent HIV-1 latent reservoir is primarily established near the time of therapy initiation. Sci. Transl. Med. 2019; 11(513): eaaw5589. https://doi.org/10.1126/scitranslmed.aaw5589
- Lai M., Maori E., Quaranta P., Matteoli G., Maggi F., Sgarbanti M., et al. CRISPR/Cas9 ablation of integrated HIV-1 accumulates proviral DNA circles with reformed long terminal repeats. J. Virol. 2021; 95(23): e0135821. https://doi.org/10.1128/jvi.01358-21
Supplementary files