Evaluation of the effectiveness of chemical inactivation and immunogenicity of the Omicron variant of the SARS-CoV-2 virus
- Authors: Zhapparova G.A.1, Myrzakhmetova B.S.1, Tlenchiyeva T.M.1, Tussipova A.A.1, Bissenbayeva K.B.1, Toytanova A.S.1, Kutumbetov L.B.1
-
Affiliations:
- Research Institute for Biological Safety Problems
- Issue: Vol 69, No 5 (2024)
- Pages: 459-469
- Section: ORIGINAL RESEARCHES
- URL: https://ogarev-online.ru/0507-4088/article/view/269835
- DOI: https://doi.org/10.36233/0507-4088-253
- EDN: https://elibrary.ru/vpecfj
- ID: 269835
Cite item
Full Text
Abstract
Introduction. The rapid spread of coronavirus infection COVID-19 among the population of many countries around the world has contributed to the emergence of many genetic variants of SARS-CoV-2. Compared to previous coronavirus variants, the new Omicron variants have shown a noticeable degree of mutation. Virus inactivation is one of the most important steps in the development of inactivated vaccines. The chemical inactivation agents currently used are β-propiolactone and formaldehyde, but there is no uniform standard for designing and specifying the inactivation process.
Objective. Evaluation and comparison of the effectiveness of chemical inactivation of two agents, formaldehyde and β-propiolactone against immunogenicity of the Omicron variant of the SARS-CoV-2 virus.
Materials and methods. Nasopharyngeal swabs were used to obtain the SARS-CoV-2 Omicron variant virus. Vero cell culture was used to isolate, reproduce, titrate the virus, and perform a neutralization reaction. The kinetics of studying the inactivation of the virus by chemical agents such as formaldehyde and β-propiolactone was carried out.
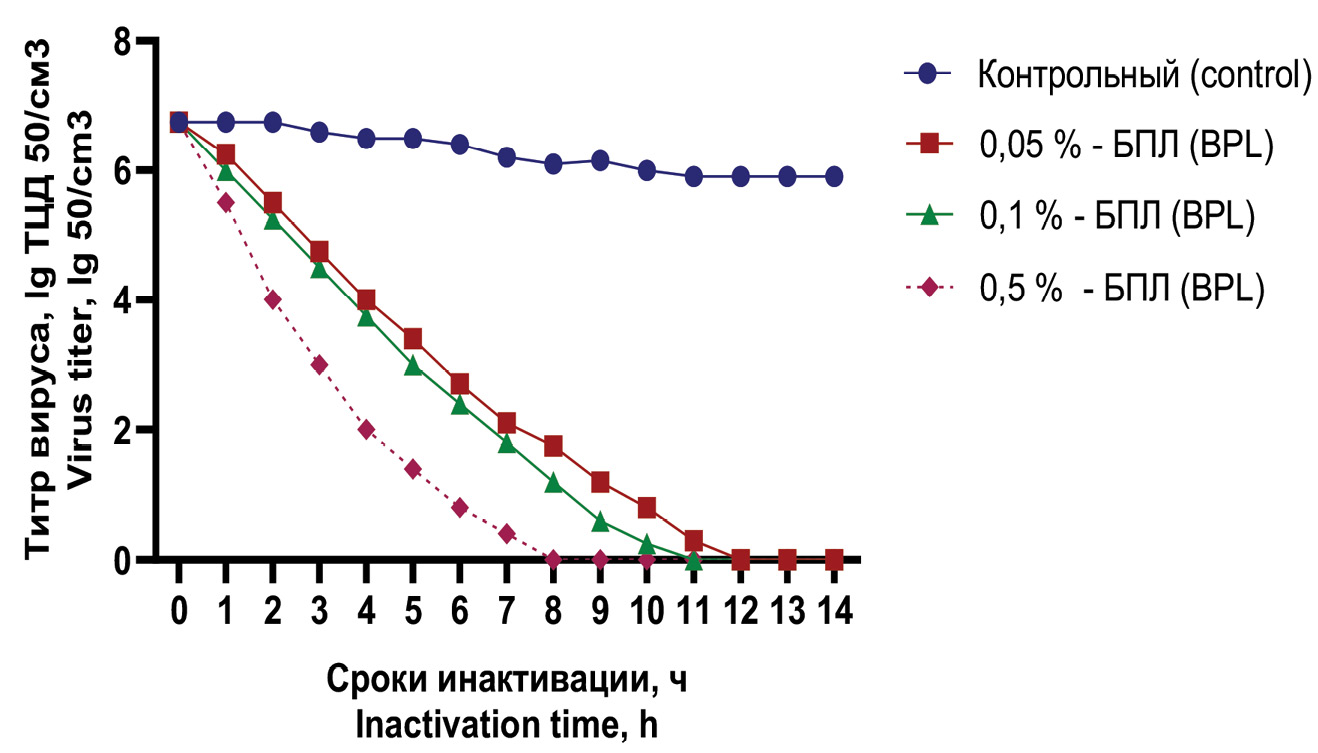
Results. Studies have been conducted to comparatively evaluate the effectiveness of chemical agents used to inactivate the SARS-CoV-2 virus of the Omicron variant, planned for use in the production of an inactivated whole-virion vaccine. Formaldehyde and β-propiolactone were used as inactivation agents in concentrations of 0.05, 0.1, 0.5% of the total volume of the virus suspension. It has been established that complete inactivation of the virus by formaldehyde in the concentrations used at a temperature of 37 °C occurs within up to 2 hours, and when using beta-propiolactone, within up to 12 hours.
Conclusion. Inactivated virus samples have different antigenic activity depending on the concentration of the inactivation agents used. The most pronounced antigenic activity is manifested in samples of the pathogen that were treated with an inactivation agent at a mild concentration of 0.05%. Increasing the concentration of inactivation agent by 5 or more times leads to a significant decrease in the antigenicity of the SARS-CoV-2 virus. With the inactivation modes used, the loss of biological activity of the virus occurs faster and antigenicity is retained largely when treated with formaldehyde.
Full Text
##article.viewOnOriginalSite##About the authors
Gulzhan A. Zhapparova
Research Institute for Biological Safety Problems
Author for correspondence.
Email: Gulzhan1003@mail.ru
ORCID iD: 0000-0001-5382-831X
master of biology, senior researcher of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, Gvardeyskiy
Balzhan S. Myrzakhmetova
Research Institute for Biological Safety Problems
Email: b.myrzakhmetova@biosafety.kz
ORCID iD: 0000-0002-4141-7174
candidate of biological sciences, head of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, GvardeyskiyTalshyngul M. Tlenchiyeva
Research Institute for Biological Safety Problems
Email: t.m.tlenchieva@mail.ru
ORCID iD: 0009-0006-7831-4212
master of chemistry, senior researcher of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, GvardeyskiyAiganym A. Tussipova
Research Institute for Biological Safety Problems
Email: aiganym.t24@gmail.com
ORCID iD: 0000-0002-7767-0542
master of Natural Sciences, senior laboratory assistant of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, GvardeyskiyKarina B. Bissenbayeva
Research Institute for Biological Safety Problems
Email: bisenbayeva.karina@bk.ru
ORCID iD: 0000-0001-5788-6074
master of biology, senior researcher of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, GvardeyskiyAizhan S. Toytanova
Research Institute for Biological Safety Problems
Email: aizhana-1308@mail.ru
ORCID iD: 0009-0004-9526-3539
master of biology, senior researcher of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, GvardeyskiyLespek B. Kutumbetov
Research Institute for Biological Safety Problems
Email: lespek.k@gmail.com
ORCID iD: 0000-0001-8481-0673
doctor of veterinary sciences, professor, chief researcher of the laboratory Especially Dangerous Infectious Diseases
Kazakhstan, 080409, GvardeyskiyReferences
- Han Q., Lin Q., Jin S., You L. Coronavirus 2019-nCoV: A brief perspective from the front line. J. Infect. 2020; 80(4): 373–7. https://doi.org/10.1016/j.jinf.2020.02.010
- Wrapp D., Wang N., Corbett K.S., Goldsmith J.A., Hsieh C.L., Abiona O., et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020; 367(6483): 1260–3. https://doi.org/10.1126/science.abb2507
- Xu D., Zhang Z., Chu F., Li Y., Jin L., Zhang L., et al. Genetic variation of SARS coronavirus in Beijing Hospital. Emerg. Infect. Dis. 2004; 10(5): 789–94. https://doi.org/10.3201/eid1005.030875
- Bai Y., Du Z., Xu M., Wang L., Wu P., Lau E.H.Y., et al. International risk of SARS-CoV-2 Omicron variant importations originating in South Africa. medRxiv. 2021; 2021.12.07.21267410. Preprint. https://doi.org/10.1101/2021.12.07.21267410
- Viana R., Moyo S., Amoako D.G., Tegally H., Scheepers C., Althaus C.L., et al. Rapid epidemic expansion of the SARS-CoV-2 Omicron variant in southern Africa. Nature. 2022; 603(7902): 679–86. https://doi.org/10.1038/s41586-022-04411-y
- Tegally H., Moir M., Everatt J., Giovanetti M., Scheepers C., Wilkinson E., et al. Emergence of SARS-CoV-2 Omicron lineages BA.4 and BA.5 in South Africa. Nat. Med. 2022; 28(9): 1785–90. https://doi.org/10.1038/s41591-022-01911-2
- Rahimi F., Bezmin Abadi A.T. The Omicron subvariant BA.2: Birth of a new challenge during the COVID-19 pandemic. Int. J. Surg. 2022; 99: 106261. https://doi.org/10.1016/j.ijsu.2022.106261
- Fonager J, Bennedbæk M, Bager P., Wohlfahrt J., Ellegaard K.M., Ingham A.C., et al. Molecular epidemiology of the SARS-CoV-2 variant Omicron BA.2 sub-lineage in Denmark, 29 November 2021 to 2 January 2022. Euro Surveill. 2022; 27(10): 2200181. https://doi.org/10.2807/1560-7917.ES.2022.27.10.2200181
- Chen L.L., Abdullah S.M.U., Chan W.M., Chan B.P., Ip J.D., Chu A.W., et al. Contribution of low population immunity to the severe Omicron BA.2 outbreak in Hong Kong. Nat. Commun. 2022; 13(1): 3618. https://doi.org/10.1038/s41467-022-31395-0
- O’Toole Á., Pybus O.G., Abram M.E., Kelly E.J., Rambaut A. Pango lineage designation and assignment using SARS-CoV-2 spike gene nucleotide sequences. BMC Genomics. 2022; 23(1): 121. https://doi.org/10.1186/s12864-022-08358-2
- Rambaut A., Holmes E.C., O’Toole Á., Hill V., McCrone J.T., Ruis C., et al. A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology. Nat. Microbiol. 2020; 5(11): 1403–7. https://doi.org/10.1038/s41564-020-0770-5
- Jung C., Kmiec D., Koepke L., Zech F., Jacob T., Sparrer K.M.J., et al. Omicron: what makes the latest SARS-CoV-2 variant of concern so concerning? J. Virol. 2022; 96(6): e020772. https://doi.org/10.1128/jvi.02077-21
- Our world in data. Mathieu E., Ritchie H., Rodés-Guirao L., Appel C., Giattino C., Hasell J., et al. Coronavirus Pandemic (COVID-19); 2020. Available at: https://ourworldindata.org/coronavirus
- Malik J.A., Ahmed S., Mir A., Shinde M., Bender O., Alshammari F., et al. The SARS-CoV-2 mutations versus vaccine effectiveness: New opportunities to new challenges. J. Infect. Public Health. 2022; 15(2): 228–40. https://doi.org/10.1016/j.jiph.2021.12.014
- Wang Q., Ye S.B., Zhou Z.J., Song A.L., Zhu X., Peng J.M., et al. Key mutations in the spike protein of SARS-CoV-2 affecting neutralization resistance and viral internalization. J. Med. Virol. 2023; 95(1): e28407. https://doi.org/10.1002/jmv.28407
- Murdin A.D., Barreto L., Plotkin S. Inactivated poliovirus vaccine: past and present experience. Vaccine. 1996; 14(8): 735–46. https://doi.org/10.1016/0264-410x(95)00211-i
- Zakarya K., Kutumbetov L., Orynbayev M., Abduraimov Y., Sultankulova K., Kassenov M., et al. Safety and immunogenicity of a QazCovid-in® inactivated whole-virion vaccine against COVID-19 in healthy adults: A single-centre, randomised, single-blind, placebo-controlled phase 1 and an open-label phase 2 clinical trials with a 6 months follow-up in Kazakhstan. EClinicalMedicine. 2021; 39: 101078. https://doi.org/10.1016/j.eclinm.2021.101078
- Gupta D., Parthasarathy H., Sah V., Tandel D., Vedagiri D., Reddy S., et al. Inactivation of SARS-CoV-2 by β-propiolactone causes aggregation of viral particles and loss of antigenic potential. Virus Res. 2021; 305: 198555. https://doi.org/10.1016/j.virusres.2021.198555
- Widera M., Westhaus S., Rabenau H.F., Hoehl S., Bojkova D., Cinatl J., et al. Evaluation of stability and inactivation methods of SARS-CoV-2 in context of laboratory settings. Med. Microbiol. Immunol. 2021; 210(4): 235–44. https://doi.org/10.1007/s00430-021-00716-3
- Awadasseid A., Wu Y., Tanaka Y., Zhang W. Current advances in the development of SARS-CoV-2 vaccines. Int. J. Biol. Sci. 2021; 17(1): 8–19. https://doi.org/10.7150/ijbs.52569
- Herrera-Rodriguez J., Signorazzi A., Holtrop M., de Vries-Idema J., Huckriede A. Inactivated or damaged? Comparing the effect of inactivation methods on influenza virions to optimize vaccine production. Vaccine. 2019; 37(12): 1630–7. https://doi.org/10.1016/j.vaccine.2019.01.086
- Patterson E.I., Prince T., Anderson E.R., Casas-Sanchez A., Smith S.L., Cansado-Utrilla C., et al. Methods of inactivation of SARS-CoV-2 for downstream biological assays. J. Infect. Dis. 2020; 222(9): 1462–7. https://doi.org/10.1093/infdis/jiaa507
- Auerswald H., Yann S., Dul S., In S., Dussart P., Martin N.J., et al. Assessment of inactivation procedures for SARS-CoV-2. J. Gen. Virol. 2021; 102(3): 001539. https://doi.org/10.1099/jgv.0.001539
- Goldstein M.A., Tauraso N.M. Effect of formalin, beta-propiolactone, merthiolate, and ultraviolet light upon influenza virus infectivity chicken cell agglutination, hemagglutination, and antigenicity. Appl. Microbiol. 1970; 19(2): 290–4. https://doi.org/10.1128/am.19.2.290-294.1970
- Fan C., Ye X., Ku Z., Kong L., Liu Q., Xu C., et al. Beta-propiolactone inactivation of coxsackievirus A16 induces structural alteration and surface modification of viral capsids. J. Virol. 2017; 91(8): e00038-17. https://doi.org/10.1128/JVI.00038-17
- Wang H., Zhang Y., Huang B., Deng W., Quan Y., Wang W., et al. Development of an inactivated vaccine candidate, BBIBP-CorV, with potent protection against SARS-CoV-2. Cell. 2020; 182(3): 713–21.e9. https://doi.org/10.1016/j.cell.2020.06.008
- Gao Q., Bao L., Mao H., Wang L., Xu K., Yang M., et al. Development of an inactivated vaccine candidate for SARS-CoV-2. Science. 2020; 369(6499): 77–81. https://doi.org/10.1126/science.abc1932
- Zhang X.Y., Guo J., Wan X., Zhou J.G., Jin W.P., Lu J., et al. Biochemical and antigenic characterization of the structural proteins and their post-translational modifications in purified SARS-CoV-2 virions of an inactivated vaccine candidate. Emerg. Microbes Infect. 2020; 9(1): 2653–62. https://doi.org/10.1080/22221751.2020.1855945
- Chen H., Xie Z., Long R., Fan S., Li H., He Z., et al. A valid protective immune response elicited in rhesus macaques by an inactivated vaccine is capable of defending against SARS-CoV-2 infection. bioRxiv. 2020; 2020.08.04.235747. Preprint. https://doi.org/10.1101/2020.08.04.235747
- Basso C.R., Malossi C.D., Haisi A., de Albuquerque Pedrosa V., Barbosa A.N., Grotto R.T., et al. Fast and reliable detection of SARS-CoV-2 antibodies based on surface plasmon resonance. Anal. Methods. 2021; 13(29): 3297–306. https://doi.org/10.1039/d1ay00737h
- Reed L., Muench H. A simple method of estimation fifty percent and pints. J. Amer. Hyg. 1938; 27(3): 493–7. https://doi.org/10.1093/oxfordjournals.aje.a118408
- Myrzakhmetova B.Sh., Zhapparova G.A., Bisenbaeva K.B., Toytanova A.S., Tuyskanova M.S., Zhugunisov K.D., et al. Immune reactivity of two biological models to vaccination with the inactivated vaccine QazVac against coronavirus infection COVID-19. Voprosy virusologii. 2024; 69(3): 219–30. https://doi.org/10.36233/0507-4088-222 https://elibrary.ru/mcackf (in Russian)
- Zhugunisov K.D., Kerimbayev A.A., Kopeev S.K., Myrzakhmetova B.Sh., Tuyskanova M.S., Nakhanov A.K., et al. SARS-CoV-2 virus: isolation, growth, thermostability, inactivation and passages. Vestnik KazNU. Seriya biologicheskaya. 2022; 90(1): 73–89. https://doi.org/10.26577/eb.2022.v90.i1.07 (in Russian)
- Chan J.F., Yip C.C., To K.K., Tang T.H., Wong S.C., Leung K.H., et al. Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. J. Clin. Microbiol. 2020; 58(5): e00310-20. https://doi.org/10.1128/jcm.00310-20
- Yuan Y., Wang R.T., Xia J., Cao H.J. Interventions for preventing influenza: an overview of Cochrane systematic reviews and a Bayesian network meta-analysis. J. Integr. Med. 2021; 19(6): 503–14. https://doi.org/10.1016/j.joim.2021.09.001
- Stuurman A.L., Marano C., Bunge E.M., De Moerlooze L., Shouval D. Impact of universal mass vaccination with monovalent inactivated hepatitis A vaccines – а systematic review. Hum. Vaccin. Immunother. 2017; 13(3): 724–36. https://doi.org/10.1080/21645515.2016.1242539
- Hegde N.R., Gore M.M. Japanese encephalitis vaccines: immunogenicity, protective efficacy, effectiveness, and impact on the burden of disease. Hum. Vaccin. Immunother. 2017; 13(6): 1–18. https://doi.org/10.1080/21645515.2017.1285472
- Yu S., Wei Y., Liang H., Ji W., Chang Z., Xie S., et al. Comparison of physical and biochemical characterizations of SARS-CoV-2 inactivated by different treatments. Viruses. 2022, 14(9): 1938. https://doi.org/10.3390/v14091938
- Kordyukova L.V., Moiseenko A.V., Serebryakova M.V., Shuklina M.A., Sergeeva M.V., Lioznov D.A., et al. Structural and immunoreactivity properties of the SARS-CoV-2 spike protein upon the development of an inactivated vaccine. Viruses. 2023; 15(2): 480. https://doi.org/10.3390/v15020480
Supplementary files