First detection of influenza A virus subtypes H1N1 and H3N8 in the Antarctic region: King George Island, 2023
- Authors: Ohlopkova O.V.1,2, Goncharov A.E.3,4, Aslanov B.I.4, Fadeev A.V.5, Davidyuk Y.N.6, Moshkin A.D.2, Stolbunova K.A.2, Stepanyuk M.A.2, Sobolev I.A.2, Tyumentseva M.A.1, Tyumentsev A.I.1, Shestopalov A.M.2, Akimkin V.G.1
-
Affiliations:
- Central Research Institute of Epidemiology, Rospotrebnadzor
- Federal Research Center for Fundamental and Translational Medicine
- Institute of Experimental Medicine
- Northwestern State Medical University named after I.I. Mechnikov
- A.A. Smorodintsev Research Institute of Influenza, Ministry of Health of the Russian Federation
- Federal State Educational Institution of Higher Education «Kazan (Volga Region) Federal University»
- Issue: Vol 69, No 4 (2024)
- Pages: 377-389
- Section: ORIGINAL RESEARCHES
- URL: https://ogarev-online.ru/0507-4088/article/view/265998
- DOI: https://doi.org/10.36233/0507-4088-257
- EDN: https://elibrary.ru/qujzfv
- ID: 265998
Cite item
Abstract
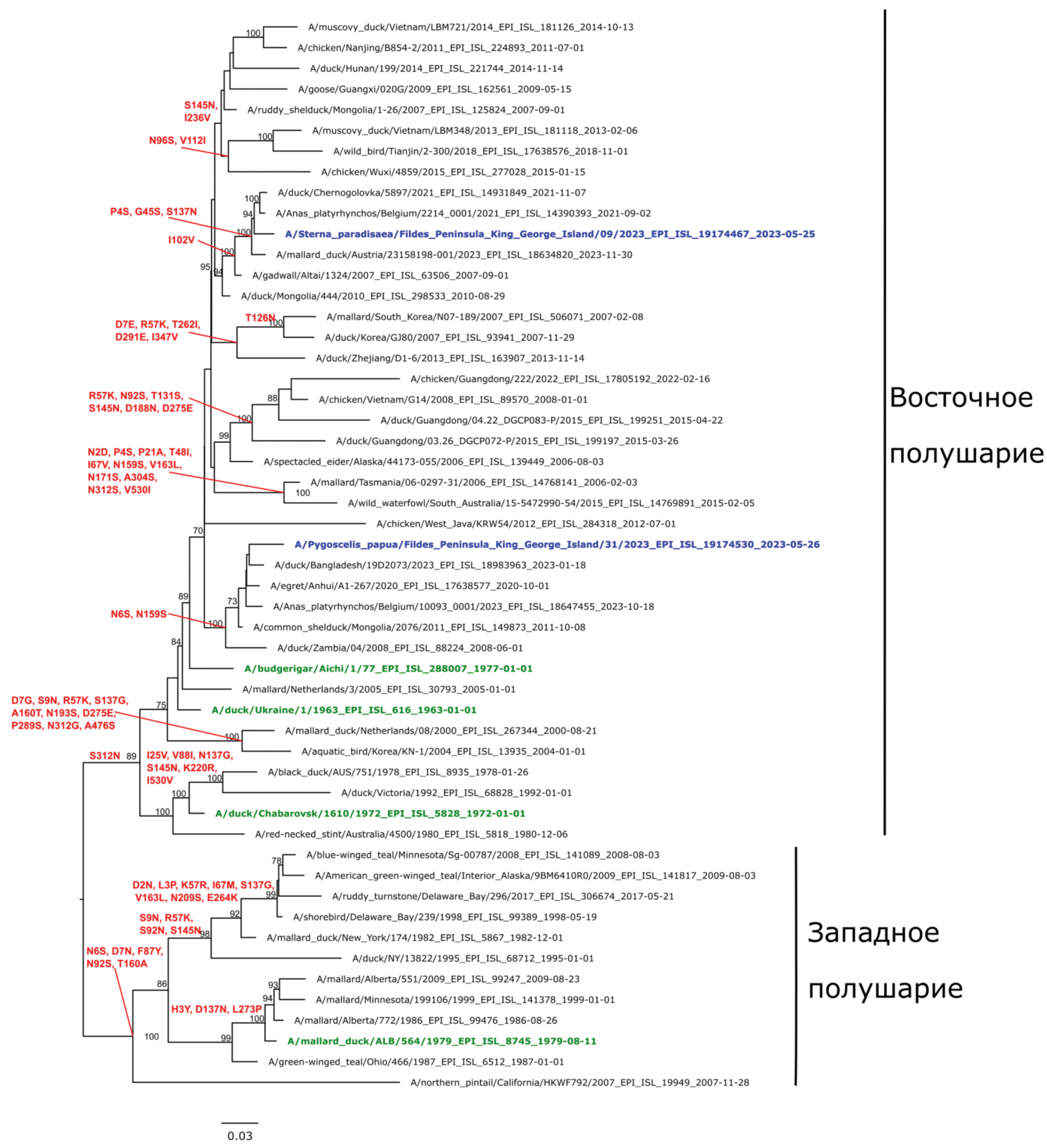
Relevance. Influenza A virus is characterized by a segmented single-stranded RNA genome. Such organization of the virus genome determines the possibility of reassortment, which can lead to the emergence of new virus variants. The main natural reservoir of most influenza A virus subtypes are wild waterfowl. Seasonal migrations gather waterfowl from all major migration routes to nesting areas near the northern and southern polar circles. This makes intercontinental spread of influenza A viruses possible.
Objective ‒ to conduct molecular genetic monitoring and study the phylogenetic relationships of influenza A virus variants circulating in Antarctica in 2023.
Materials and methods. We studied 84 samples of biological material obtained from birds and marine mammals in April‒May 2023 in coastal areas of Antarctica. For 3 samples, sequencing was performed on the Miseq, Illumina platform and phylogenetic analysis of the obtained nucleotide sequences of the influenza A virus genomes was performed.
Results. The circulation of avian influenza virus in the Antarctic region was confirmed. Heterogeneity of the pool of circulating variants of the influenza A virus (H3N8, H1N1) was revealed. Full-length genomes of the avian influenza virus were sequenced and posted in the GISAID database (EPI_ISL_19032103, 19174530, 19174467).
Conclusion. The study of the genetic diversity of influenza A viruses circulating in the polar regions of the Earth and the identification of the conditions for the emergence of new genetic variants is a relevant task for the development of measures to prevent biological threats.
Full Text
##article.viewOnOriginalSite##About the authors
Olesia V. Ohlopkova
Central Research Institute of Epidemiology, Rospotrebnadzor; Federal Research Center for Fundamental and Translational Medicine
Author for correspondence.
Email: ohlopkova.lesia@yandex.ru
ORCID iD: 0000-0002-8214-7828
PhD in Biology, Senior Researcher of the Laboratory of Experimental Pharmacology, Virology Research Institute
Russian Federation, 111123, Moscow; 630060, NovosibirskArtemy E. Goncharov
Institute of Experimental Medicine; Northwestern State Medical University named after I.I. Mechnikov
Email: phage1@yandex.ru
ORCID iD: 0000-0002-5206-6656
Doctor of Medical Sciences, Head of the Laboratory of Functional Genomics and Proteomics of Microorganisms; Professor of the Department of Epidemiology, Parasitology and Disinfection
Russian Federation, 197022, St. Petersburg; 191015, St. PetersburgBatyrbek I. Aslanov
Northwestern State Medical University named after I.I. Mechnikov
Email: Batyrbek.Aslanov@szgmu.ru
ORCID iD: 0000-0002-6890-8096
Doctor of Medical Sciences, Professor, and Head of the Department of Epidemiology, Parasitology and Disinfectology
Russian Federation, 191015, St. PetersburgArtem V. Fadeev
A.A. Smorodintsev Research Institute of Influenza, Ministry of Health of the Russian Federation
Email: artem.fadeev@influenza.spb.ru
ORCID iD: 0000-0003-3558-3261
Senior Researcher, Laboratory of Molecular Virology
Russian Federation, 197376, St. PetersburgYuri N. Davidyuk
Federal State Educational Institution of Higher Education «Kazan (Volga Region) Federal University»
Email: JNDavidjuk@kpfu.ru
ORCID iD: 0000-0002-4409-2942
PhD in Biology, Senior Researcher at the Institute of Fundamental Medicine and Biology
Russian Federation, 420008, KazanAlexey D. Moshkin
Federal Research Center for Fundamental and Translational Medicine
Email: alex.moshkin727@gmail.com
ORCID iD: 0000-0002-1182-8247
researcher at the Research Institute of Virology
Russian Federation, 630060, NovosibirskKristina A. Stolbunova
Federal Research Center for Fundamental and Translational Medicine
Email: kristina.sunwo@yandex.ru
ORCID iD: 0000-0003-3376-945X
researcher, Research Institute of Virology
Russian Federation, 630060, NovosibirskMarina A. Stepanyuk
Federal Research Center for Fundamental and Translational Medicine
Email: stepanunya1996@gmail.com
ORCID iD: 0009-0002-2658-7746
researcher, Research Institute of Virology
Russian Federation, 630060, NovosibirskIvan A. Sobolev
Federal Research Center for Fundamental and Translational Medicine
Email: sobolev_i@centercem.ru
ORCID iD: 0000-0002-4561-6517
PhD in Biology, Head of the Laboratory of Genomics and Evolution of Viruses, Research Institute of Virology
Russian Federation, 630060, NovosibirskMarina A. Tyumentseva
Central Research Institute of Epidemiology, Rospotrebnadzor
Email: tyumantseva@cmd.ru
ORCID iD: 0000-0002-3145-3702
PhD in Biology, Head of the Laboratory of Genomic Editing of the Department of Molecular Diagnostics and Epidemiology
Russian Federation, 111123, MoscowAlexander I. Tyumentsev
Central Research Institute of Epidemiology, Rospotrebnadzor
Email: tyumantsev@cmd.ru
ORCID iD: 0000-0003-0537-2586
PhD in Biology, Head of the Laboratory of Experimental Pharmacology, Department of Molecular Diagnostics and Epidemiology
Russian Federation, 111123, MoscowAlexander M. Shestopalov
Federal Research Center for Fundamental and Translational Medicine
Email: amshestopalov@frcftm.ru
ORCID iD: 0000-0002-9734-0620
Doctor of Biological Sciences, Professor, Honored Worker of Science of the Russian Federation, Director of the Research Institute of Virology
Russian Federation, 630060, NovosibirskVasily G. Akimkin
Central Research Institute of Epidemiology, Rospotrebnadzor
Email: akinkin@pcr.ms
ORCID iD: 0000-0003-4228-9044
Doctor of Medical Sciences, Academician of the Russian Academy of Sciences, Professor, Honored Doctor of the Russian Federation, Laureate of the Prize of the Government of the Russian Federation in the field of science and technology, Director
Russian Federation, 111123, MoscowReferences
- Bouvier N.M., Palese P. The biology of influenza viruses. Vaccine. 2008; 26(Suppl. 4): D49–53. https://doi.org/10.1016/j.vaccine.2008.07.039
- Fries A.C., Nolting J.M., Bowman A.S., Lin X., Halpin R.A., Wester E., et al. Spread and persistence of influenza A viruses in waterfowl hosts in the North American Mississippi migratory flyway. J. Virol. 2015; 89(10): 5371–81. https://doi.org/10.1128/JVI.03249-14
- Lvov D.K., Zhdanov V.M. Circulation of influenza virus genes in the biosphere. In: Zhdanov V.M., ed. Soviet Medical Reviews. Section E: Virology Reviews. Volume 1. Harwood Academic Publ. GmbH; 1984: 129–52.
- Lvov D.K. Circulation of Influenza Virus in Natural Biocenosis. In: Kurstak E., Maramorosh K., eds. Viruses and Enviroment. Academic Press; 1978: 351–80.
- Lvov D.K. Influenza A virus – a sum of populations with a common protected gene pool. In: Zhdanov V.M., ed. Soviet Medical Reviews. Section E: Virology Reviews. Volume 2. Harwood Academic Publ. GmbH; 1984: 15–37.
- Lvov D.K., Kaverin N.V. Avian influenza in Northern Eurasia. In: Klenk H.D., Matrosovich M.N., eds. Avian Influenza. Volume 27. Basel: Karger; 2008: 41–58.
- Lvov D.K., Ilyichev V.D., ed. Bird Migration and Transmission of Infectious Agents [Migratsiya ptits i perenos vozbuditelei infektsii]. Moscow: Nauka; 1979. (in Russian)
- Walker P.J., Siddell S.G., Lefkowitz E.J., Mushegian A.R., Adriaenssens E.M., Alfenas-Zerbini P., et al. Recent changes to virus taxonomy ratified by the International Committee on Taxonomy of Viruses. Arch. Virol. 2022; 167(11): 2429–40. https://doi.org/10.1007/s00705-022-05516-5
- Gass J.D. Jr., Dusek R.J., Hall J.S., Hallgrimsson G.T., Halldórsson H.P., Vignisson S.R., et al. Global dissemination of influenza A virus is driven by wild bird migration through arctic and subarctic zones. Mol. Ecol. 2023; 32(1): 198–213. https://doi.org/10.1111/mec.16738
- Wille M., Holmes E.C. The ecology and evolution of influenza viruses. Cold. Spring Harb. Perspect. Med. 2020; 10(7): a038489. https://doi.org/10.1101/cshperspect.a038489
- Cowling B.J., Ip D.K., Fang V.J., Suntarattiwong P., Olsen S.J., Levy J., et al. Aerosol transmission is an important mode of influenza A virus spread. Nat. Commun. 2013; 4: 1935. https://doi.org/10.1038/ncomms2922
- Barratclough A., Ferguson S.H., Lydersen C., Thomas P.O., Kovacs K.M. A review of circumpolar arctic marine mammal health – a call to action in a time of rapid environmental change. Pathogens. 2023; 12(7): 937. https://doi.org/10.3390/pathogens12070937
- Lvov D.K. Ecology of viruses. In: Lvov D.K., ed. A Guide to Virology. Viruses and Viral Infections of Humans and Animals [Rukovodstvo po virusologii. Virusy i virusnye infektsii cheloveka i zhivotnykh]. Moscow: MIA; 2013: 66–86. (in Russian)
- Lvov D.K., Zdanov V.M., Sazonov A.A., Braude N.A., Vladimirtceva E.A., Agafonova L.V., et al. Comparison of influenza viruses isolated from man and from whales. Bull World Health Organ. 1978; 56(6): 923–30.
- Nielsen O., Clavijo A., Boughen J.A. Serologic evidence of influenza A infection in marine mammals of Arctic Canada. J. Wildl. Dis. 2001; 37(4): 820–25. https://doi.org/10.7589/0090-3558-37.4.820
- Celis J.E., Espejo W., Barra R., Gonzalez-Acuña D., Gonzalez F., Jara S. Assessment of trace metals in droppings of Adélie penguins (Pygoscelis Adeliae) from different locations of the Antarctic peninsula area. Adv. Polar Sci. 2015; 26(1): 1–7. https://doi.org/10.13679/j.advps.2015.1.00001
- Hahn S., Peter H.U., Quillfeldt P., Reinhardt K. The birds of the Potter Peninsula, King George Island, South Shetland Islands, Antarctica, 1965-1998. Marine Ornithology. 1998; 26: 1–6.
- Wille M., Aban M., Wang J., Moore N., Shan S., Marshall J., et al. Antarctic penguins as reservoirs of diversity for avian avulaviruses. J. Virol. 2019; 93(11): e00271-19. https://doi.org/10.1128/JVI.00271-19
- Siniff D.B., Garrott R.A., Rotella J.J., Fraser W.R., Ainley D.G. Opinion: Projecting the effects of environmental change on Antarctic seals. Antarct. Sci. 2008; 20(5): 425–35. doi: 10.1017/S0954102008001351
- Shao W., Li X., Goraya M.U., Wang S., Chen J.L. Evolution of influenza A virus by mutation and re-assortment. Int. J. Mol. Sci. 2017; 18(8): 1650. https://doi.org/10.3390/ijms18081650
- Thorsson E., Zohari S., Roos A., Banihashem F., Bröjer C., Neimanis A. Highly pathogenic avian influenza A(H5N1) virus in a Harbor porpoise, Sweden. Emerg. Infect. Dis. 2023; 29(4): 852–5. https://doi.org/10.3201/eid2904.221426
- Markin A., Wagle S., Grover S., Vincent Baker A.L., Eulenstein O., Anderson T.K. PARNAS: objectively selecting the most representative taxa on a phylogeny. Syst. Biol. 2023; 72(5): 1052–63. https://doi.org/10.1093/sysbio/syad028
- Stamatakis A. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies. Bioinformatics. 2014; 30(9): 1312–3. https://doi.org/10.1093/bioinformatics/btu033
- Short K.R., Richard M., Verhagen J.H., van Riel D., Schrauwen E.J., van den Brand J.M., et al. One health, multiple challenges: The inter-species transmission of influenza A virus. One Health. 2015; 1: 1–13. https://doi.org/10.1016/j.onehlt.2015.03.001
- Al Hajjar S., McIntosh K. The first influenza pandemic of the 21st century. Ann. Saudi Med. 2010; 30(1): 1–10. https://doi.org/10.4103/0256-4947.59365
- Yu J., Yao Q., Liu J., Zhou Y., Huo M., Ge Y. Concern regarding H3-subtype avian influenza virus. Front. Microbiol. 2023; 14: 1327470. https://doi.org/10.3389/fmicb.2023.1327470
- Scholtissek C., von Hoyningen-Huene V. Genetic relatedness of the gene which codes for the nonstructural (NS) protein of different influenza A strains. Virology. 1980; 102(1): 13–20. https://doi.org/10.1016/0042-6822(80)90065-3
- Jahangir A., Ruenphet S., Sultana N., Shoham D., Takehara K. Genetic analysis of avian influenza viruses: cocirculation of avian influenza viruses with allele A and B nonstructural gene in Northern Pintail (Anas acuta) ducks wintering in Japan. Influenza Res. Treat. 2012; 2012: 847505. https://doi.org/10.1155/2012/847505
- Herfst S., Zhang J., Richard M., McBride R., Lexmond P., Bestebroer T.M., et al. Hemagglutinin traits determine transmission of avian A/H10N7 influenza virus between mammals. Cell Host Microbe. 2020; 28(4): 602–13.e7. https://doi.org/10.1016/j.chom.2020.08.011
- Shi Y., Wu Y., Zhang W., Qi J., Gao G.F. Enabling the ‘host jump’: structural determinants of receptor-binding specificity in influenza A viruses. Nat. Rev. Microbiol. 2014; 12(12): 822–31. https://doi.org/10.1038/nrmicro3362
Supplementary files