The Molecular and Biological Patterns Underlying Sustained SARS-CoV-2 Circulation in the Human Population
- Authors: Kustova D.D.1,2, Pochtovyi A.A.1,2,3, Shpakova O.G.4, Shtinova I.A.4, Kuznetsova N.A.1, Kleimenov D.A.1, Komarov A.G.4, Gushchin V.A.1,2,3
-
Affiliations:
- National Research Centre for Epidemiology and Microbiology Named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
- Federal State Budgetary Educational Institution of Higher Education Lomonosov Moscow State University
- I.M. Sechenov First Moscow State Medical University of the Ministry of Health of the Russian Federation (Sechenov University)
- Moscow Healthcare Department
- Issue: Vol 69, No 4 (2024)
- Pages: 329-340
- Section: ORIGINAL RESEARCHES
- URL: https://ogarev-online.ru/0507-4088/article/view/265959
- DOI: https://doi.org/10.36233/0507-4088-242
- EDN: https://elibrary.ru/uxnluj
- ID: 265959
Cite item
Abstract
Introduction. For four years, SARS-CoV-2, the etiological agent of COVID-19, has been circulating among humans. By the end of the second year, an absence of immunologically naive individuals was observed, attributable to extensive immunization efforts and natural viral exposure. This study focuses on delineating the molecular and biological patterns that facilitate the persistence of SARS-CoV-2, thereby informing predictions on the epidemiological trajectory of COVID-19 toward refining pandemic countermeasures.
The aim of this study was to describe the molecular biological patterns identified that contribute to the persistence of the virus in the human population.
Materials and methods. For over three years since the beginning of the COVID-19 pandemic, molecular genetic monitoring of SARS-CoV-2 has been conducted, which included the collection of nasopharyngeal swabs from infected individuals, assessment of viral load, and subsequent whole-genome sequencing.
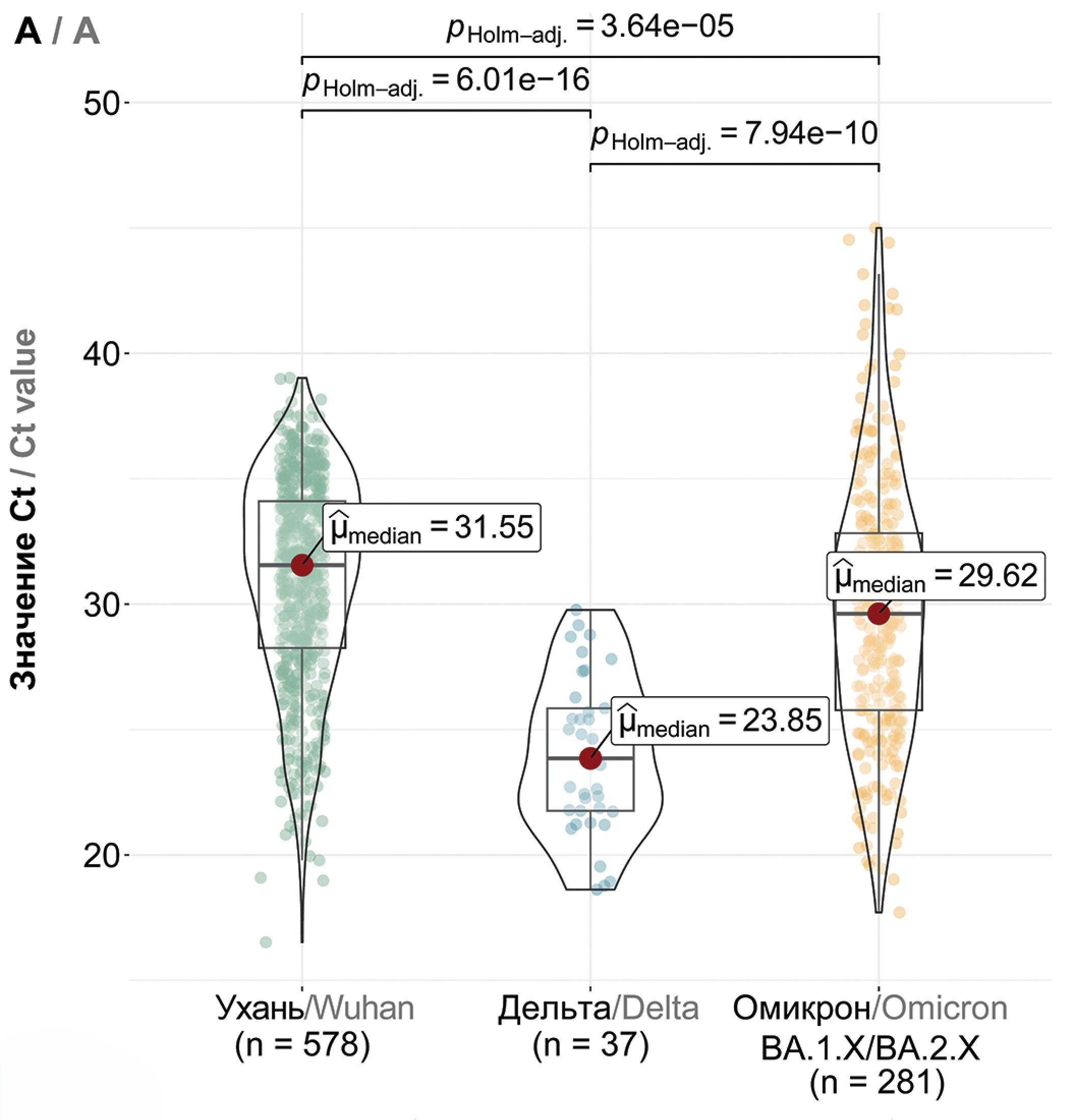
Results. We discerned dominant genetic lineages correlated with rising disease incidence. We scrutinized amino acid substitutions across SARS-CoV-2 proteins and quantified viral loads in swab samples from patients with emerging COVID-19 variants. Our findings suggest a model of viral persistence characterized by 1) periodic serotype shifts causing substantial diminutions in serum virus-neutralizing activity (> 10-fold), 2) serotype-specific accrual of point mutations in the receptor-binding domain (RBD) to modestly circumvent neutralizing antibodies and enhance receptor affinity, and 3) a gradually increasing amount of virus being shed in mucosal surfaces within a single serotype.
Conclusion. This model aptly accounts for the dynamics of COVID-19 incidence in Moscow. For a comprehensive understanding of these dynamics, acquiring population-level data on immune tension and antibody neutralization relative to genetic lineage compositions is essential.
Keywords
Full Text
##article.viewOnOriginalSite##About the authors
Daria D. Kustova
National Research Centre for Epidemiology and Microbiology Named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation; Federal State Budgetary Educational Institution of Higher Education Lomonosov Moscow State University
Email: kustovadaria@yandex.ru
ORCID iD: 0000-0002-8382-275X
junior researcher, Laboratory of mechanisms of population variability of pathogenic microorganisms; PhD student, Department of Virology, Faculty of Biology
Russian Federation, 123098, Moscow; 119991, MoscowAndrei A. Pochtovyi
National Research Centre for Epidemiology and Microbiology Named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation; Federal State Budgetary Educational Institution of Higher Education Lomonosov Moscow State University; I.M. Sechenov First Moscow State Medical University of the Ministry of Health of the Russian Federation (Sechenov University)
Author for correspondence.
Email: a.pochtovyy@gamaleya.org
ORCID iD: 0000-0003-1107-9351
Cand. Sci. (Biol.), senior researcher, Laboratory of mechanisms of population variability of pathogenic microorganisms
Russian Federation, 123098, Moscow; 119991, Moscow; 119435, MoscowOlga G. Shpakova
Moscow Healthcare Department
Email: shpakovaog@dcli.ru
Head of the laboratory of the Moscow Scientific and Practical Center for Laboratory Research
Russian Federation, 127006, MoscowIrina A. Shtinova
Moscow Healthcare Department
Email: shtinovaia@dcli.ru
Head of Laboratory Center of the Moscow Scientific and Practical Center for Laboratory Research
Russian Federation, 127006, MoscowNadezhda A. Kuznetsova
National Research Centre for Epidemiology and Microbiology Named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: nadyakuznetsova0@yandex.ru
ORCID iD: 0000-0002-7399-7628
Cand. Sci. (Biol.), senior researcher, Laboratory of mechanisms of population variability of pathogenic microorganisms
Russian Federation, 123098, MoscowDenis A. Kleimenov
National Research Centre for Epidemiology and Microbiology Named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation
Email: mne10000let@yandex.ru
ORCID iD: 0000-0001-9422-7238
Cand. Sci. (Med.), Head, Laboratory of translational biomedicine
Russian Federation, 123098, MoscowAndrey G. Komarov
Moscow Healthcare Department
Email: komarovag@zdrav.mos.ru
ORCID iD: 0009-0000-8597-7125
Cand. Sci. (Med.), Head of the Moscow Scientific and Practical Center for Laboratory Research
Russian Federation, 127006, MoscowVladimir A. Gushchin
National Research Centre for Epidemiology and Microbiology Named after Honorary Academician N.F. Gamaleya of the Ministry of Health of the Russian Federation; Federal State Budgetary Educational Institution of Higher Education Lomonosov Moscow State University; I.M. Sechenov First Moscow State Medical University of the Ministry of Health of the Russian Federation (Sechenov University)
Email: wowaniada@yandex.ru
ORCID iD: 0000-0002-9397-3762
Dr. Sci. (Biology), Head, Laboratory of mechanisms of population variability of pathogenic microorganisms, Reference center for coronavirus infection; senior researcher, Department of virology, Biological faculty
Russian Federation, 123098, Moscow; 119991, Moscow; 119435, MoscowReferences
- COVID-19 epidemiological update – 12 April 2024. Available at: https://who.int/publications/m/item/covid-19-epidemiological-update-edition-166
- CDC: SARS-CoV-2 Variant Classifications and Definitions. Available at: https://www.cdc.gov/coronavirus/2019-ncov/variants/variant-classifications.html
- Polack F.P., Thomas S.J., Kitchin N., Absalon J., Gurtman A., Lockhart S., et al. Safety and efficacy of the BNT162b2 mRNA COVID-19 vaccine. N. Engl. J. Med. 2020; 383(27): 2603–15. https://doi.org/10.1056/NEJMoa2034577
- Baden L.R., El Sahly H.M., Essink B., Kotloff K., Frey S., Novak R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N. Engl. J. Med. 2021; 384(5): 403–16. https://doi.org/10.1056/nejmoa2035389
- Logunov D.Y., Dolzhikova I.V., Shcheblyakov D.V., Tukhvatulin A.I., Zubkova O.V., Dzharullaeva A.S., et al. Safety and efficacy of an rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine: an interim analysis of a randomised controlled phase 3 trial in Russia. Lancet. 2021; 397(10275): 671–81. https://doi.org/10.1016/S0140-6736(21)00234-8
- Voysey M., Clemens S.A.C., Madhi S.A., Weckx L.Y., Folegatti P.M., Aley P.K., et al. Safety and efficacy of the ChAdOx1 nCoV-19 vaccine (AZD1222) against SARS-CoV-2: an interim analysis of four randomised controlled trials in Brazil, South Africa, and the UK. Lancet. 2021; 397(10269): 99–111. https://doi.org/10.1016/S0140-6736(20)32661-1
- Lau J.J., Cheng S.M.S., Leung K., Lee C.K., Hachim A., Tsang L.C.H., et al. Real-world COVID-19 vaccine effectiveness against the Omicron BA.2 variant in a SARS-CoV-2 infection-naive population. Nat. Med. 2023; 29(2): 348–57. https://doi.org/10.1038/s41591-023-02219-5
- Kurhade C., Zou J., Xia H., Liu M., Chang H.C., Ren P., et al. Low neutralization of SARS-CoV-2 Omicron BA.2.75.2, BQ.1.1 and XBB.1 by parental mRNA vaccine or a BA.5 bivalent booster. Nat. Med. 2023; 29(2): 344–7. https://doi.org/10.1038/s41591-022-02162-x
- Miteva D., Kitanova M., Batselova H., Lazova S., Chervenkov L., Peshevska-Sekulovska M., et al. The end or a new era of development of SARS-CoV-2 virus: genetic variants responsible for severe COVID-19 and clinical efficacy of the most commonly used vaccines in clinical practice. Vaccines (Basel). 2023; 11(7): 1181. https://doi.org/10.3390/vaccines11071181
- Munro A.P.S., Janani L., Cornelius V., Aley P.K., Babbage G., Baxter D., et al. Safety and immunogenicity of seven COVID-19 vaccines as a third dose (booster) following two doses of ChAdOx1 nCov-19 or BNT162b2 in the UK (COV-BOOST): a blinded, multicentre, randomised, controlled, phase 2 trial. Lancet. 2021; 398(10318): 2258–76. https://doi.org/10.1016/S0140-6736(21)02717-3
- Chenchula S., Karunakaran P., Sharma S., Chavan M. Current evidence on efficacy of COVID-19 booster dose vaccination against the Omicron variant: A systematic review. J. Med. Virol. 2022; 94(7): 2969–76. https://doi.org/10.1002/jmv.27697
- Chalkias S., Harper C., Vrbicky K., Walsh S.R., Essink B., Brosz A., et al. A bivalent Omicron-containing booster vaccine against COVID-19. N. Engl. J. Med. 2022; 387(14): 1279–91. https://doi.org/10.1056/NEJMoa2208343
- Scheaffer S.M., Lee D., Whitener B., Ying B., Wu K., Liang C.Y., et al. Bivalent SARS-CoV-2 mRNA vaccines increase breadth of neutralization and protect against the BA.5 Omicron variant in mice. Nat. Med. 2023; 29(1): 247–57. https://doi.org/10.1038/s41591-022-02092-8
- Winokur P., Gayed J., Fitz-Patrick D., Thomas S.J., Diya O., Lockhart S., et al. Bivalent Omicron BA.1-adapted BNT162b2 booster in adults older than 55 years. N. Engl. J. Med. 2023; 388(3): 214–27. https://doi.org/10.1056/NEJMoa2213082
- Kirsebom F.C.M., Andrews N., Stowe J., Ramsay M., Lopez Bernal J. Duration of protection of ancestral-strain monovalent vaccines and effectiveness of bivalent BA.1 boosters against COVID-19 hospitalisation in England: a test-negative case-control study. Lancet Infect. Dis. 2023; 23(11): 1235–43. https://doi.org/10.1016/S1473-3099(23)00365-1
- Iketani S., Mohri H., Culbertson B., Hong S.J., Duan Y., Luck M.I., et al. Multiple pathways for SARS-CoV-2 resistance to nirmatrelvir. Nature. 2023; 613(7944): 558–64. https://doi.org/10.1038/s41586-022-05514-2
- Stevens L.J., Pruijssers A.J., Lee H.W., Gordon C.J., Tchesnokov E.P., Gribble J., et al. Mutations in the SARS-CoV-2 RNA-dependent RNA polymerase confer resistance to remdesivir by distinct mechanisms. Sci. Transl. Med. 2022; 14(656): eabo0718. https://doi.org/10.1126/scitranslmed.abo0718
- Imai M., Ito M., Kiso M., Yamayoshi S., Uraki R., Fukushi S., et al. Efficacy of Antiviral Agents against Omicron Subvariants BQ.1.1 and XBB. N. Engl. J. Med. 2023; 388(1): 89–91. https://doi.org/10.1056/NEJMc2214302
- Cao Y., Wang J., Jian F., Xiao T., Song W., Yisimayi A., et al. Omicron escapes the majority of existing SARS-CoV-2 neutralizing antibodies. Nature. 2022; 602(7898): 657–63. https://doi.org/10.1038/s41586-021-04385-3
- Wang Q., Guo Y., Iketani S., Nair M.S., Li Z., Mohri H., et al. Antibody evasion by SARS-CoV-2 Omicron subvariants BA.2.12.1, BA.4 and BA.5. Nature. 2022; 608(7923): 603–8. https://doi.org/10.1038/s41586-022-05053-w
- Pochtovyi A.A., Kustova D.D., Siniavin A.E., Dolzhikova I.V., Shidlovskaya E.V., Shpakova O.G., et al. In vitro efficacy of antivirals and monoclonal antibodies against SARS-CoV-2 Omicron lineages XBB.1.9.1, XBB.1.9.3, XBB.1.5, XBB.1.16, XBB.2.4, BQ.1.1.45, CH.1.1, and CL.1. Vaccines (Basel). 2023; 11(10): 1533. https://doi.org/10.3390/vaccines11101533
- Puhach O., Meyer B., Eckerle I. SARS-CoV-2 viral load and shedding kinetics. Nat. Rev. Microbiol. 2023; 21(3): 147–61. https://doi.org/10.1038/s41579-022-00822-w
- Gushchin V.A., Pochtovyi A.A., Kustova D.D., Ogarkova D.A., Tarnovetskii I.Y., Belyaeva E.D., et al. Dynamics of SARS-CoV-2 major genetic lineages in moscow in the context of vaccine prophylaxis. Int. J. Mol. Sci. 2022; 23(23): 14670. https://doi.org/10.3390/ijms232314670
- Wickham H., François R., Henry L., Müller K., Vaughan D. dplyr: A Grammar of Data Manipulation. Available at: https://dplyr.tidyverse.org/reference/dplyr-package.html
- Wickham H. ggplot2. New York, NY: Springer; 2009. https://doi.org/10.1007/978-0-387-98141-3
- Patil I. Visualizations with statistical details: The “ggstatsplot” approach. J. Open. Source. Softw. 2021; 6(61): 3167. https://doi.org/10.21105/joss.03167
- Korber B., Fischer W.M., Gnanakaran S., Yoon H., Theiler J., Abfalterer W., et al. Tracking changes in SARS-CoV-2 spike: evidence that D614G increases infectivity of the COVID-19 virus. Cell. 2020; 182(4): 812–27.e19. https://doi.org/10.1016/j.cell.2020.06.043
- Markov P.V., Ghafari M., Beer M., Lythgoe K., Simmonds P., Stilianakis N.I., et al. The evolution of SARS-CoV-2. Nat. Rev. Microbiol. 2023; 21(6): 361–79. https://doi.org/10.1038/s41579-023-00878-2
- Klink G.V., Safina K.R., Nabieva E., Shvyrev N., Garushyants S., Alekseeva E., et al. The rise and spread of the SARS-CoV-2 AY.122 lineage in Russia. Virus Evol. 2022; 8(1): veac017. https://doi.org/10.1093/ve/veac017
- Teyssou E., Delagrèverie H., Visseaux B., Lambert-Niclot S., Brichler S., Ferre V., et al. The Delta SARS-CoV-2 variant has a higher viral load than the Beta and the historical variants in nasopharyngeal samples from newly diagnosed COVID-19 patients. J. Infect. 2021; 83(4): e1–3. https://doi.org/10.1016/j.jinf.2021.08.027
- von Wintersdorff C.J.H., Dingemans J., van Alphen L.B., Wolffs P.F.G., van der Veer B.M.J.W., Hoebe C.J.P.A., et al. Infections with the SARS-CoV-2 Delta variant exhibit fourfold increased viral loads in the upper airways compared to Alpha or non-variants of concern. Sci. Rep. 2022; 12(1): 13922. https://doi.org/10.1038/s41598-022-18279-5
- Guo H., Jiang J., Shen S., Ge X., Fan Q., Zhou B., et al. Additional mutations based on Omicron BA.2.75 mediate its further evasion from broadly neutralizing antibodies. IScience. 2023; 26(4): 106283. https://doi.org/10.1016/j.isci.2023.106283
- Motozono C., Toyoda M., Zahradnik J., Saito A., Nasser H., Tan T.S., et al. SARS-CoV-2 spike L452R variant evades cellular immunity and increases infectivity. Cell Host Microbe. 2021; 29(7): 1124–36.e11. https://doi.org/10.1016/j.chom.2021.06.006
- Singh P., Sharma K., Singh P., Bhargava A., Negi S.S., Sharma P., et al. Genomic characterization unravelling the causative role of SARS-CoV-2 Delta variant of lineage B.1.617.2 in 2nd wave of COVID-19 pandemic in Chhattisgarh, India. Microb. Pathog. 2022; 164: 105404. https://doi.org/10.1016/j.micpath.2022.105404
- Sarkar P., Banerjee S., Saha S.A., Mitra P., Sarkar S. Genome surveillance of SARS-CoV-2 variants and their role in pathogenesis focusing on second wave of COVID-19 in India. Sci. Rep. 2023; 13(1): 4692. https://doi.org/10.1038/s41598-023-30815-5
- Lin X., Sha Z., Trimpert J., Kunec D., Jiang C., Xiong Y., et al. The NSP4 T492I mutation increases SARS-CoV-2 infectivity by altering non-structural protein cleavage. Cell Host Microbe. 2023; 31(7): 1170–84. https://doi.org/10.1016/j.chom.2023.06.002
- Kim S.M., Kim E.H., Anthony M., Casel B., Kim Y.I., Sun R., et al. SARS-CoV-2 variants show temperature-dependent enhanced polymerase activity in the upper respiratory tract and high transmissibility. bioRxiv. 2022. Preprint. https://doi.org/10.1101/2022.09.27.509689.
- Campbell F., Archer B., Laurenson-Schafer H., Jinnai Y., Konings F., Batra N., et al. Increased transmissibility and global spread of SARS-CoV-2 variants of concern as at June 2021. Euro Surveill. 2021; 26(24): 2100509. https://doi.org/10.2807/1560-7917.ES.2021.26.24.2100509
- Earnest R., Uddin R., Matluk N., Renzette N., Turbett S.E., Siddle K.J., et al. Comparative transmissibility of SARS-CoV-2 variants Delta and Alpha in New England, USA. Cell Rep. Med. 2022; 3(4): 100583. https://doi.org/10.1016/j.xcrm.2022.100583
- Fan Y., Li X., Zhang L., Wan S., Zhang L., Zhou F. SARS-CoV-2 Omicron variant: recent progress and future perspectives. Signal Transduct. Target. Ther. 2022; 7(1): 141. https://doi.org/10.1038/s41392-022-00997-x
- Sukhikh G.T., Priputnevich T.V., Ogarkova D.A., Pochtovyi A.A., Kustova D.D., Zlobin V.I., et al. Sputnik light and Sputnik V vaccination is effective at protecting medical personnel from COVID-19 during the period of Delta variant dominance. Vaccines (Basel). 2022; 10(11): 1804. https://doi.org/10.3390/vaccines10111804
- Planas D., Veyer D., Baidaliuk A., Staropoli I., Guivel-Benhassine F., Rajah M.M., et al. Reduced sensitivity of SARS-CoV-2 variant Delta to antibody neutralization. Nature. 2021; 596(7871): 276–80. https://doi.org/10.1038/s41586-021-03777-9
- Simon-Loriere E., Schwartz O. Towards SARS-CoV-2 serotypes? Nat. Rev. Microbiol. 2022; 20(4): 187–8. https://doi.org/10.1038/s41579-022-00708-x
- Wu H., Xing N., Meng K., Fu B., Xue W., Dong P., et al. Nucleocapsid mutations R203K/G204R increase the infectivity, fitness, and virulence of SARS-CoV-2. Cell Host Microbe. 2021; 29(12): 1788–801. https://doi.org/10.1016/j.chom.2021.11.005
- Bills C.J., Xia H., Chen J.Y.C., Yeung J., Kalveram B.K., Walker D., et al. Mutations in SARS-CoV-2 variant nsp6 enhance type-I interferon antagonism. Emerg. Microbes Infect. 2023; 12(1): 2209208. https://doi.org/10.1080/22221751.2023.2209208
- Liu Y., Liu J., Plante K.S., Plante J.A., Xie X., Zhang X., et al. The N501Y spike substitution enhances SARS-CoV-2 infection and transmission. Nature. 2022; 602(7896): 294–99. https://doi.org/10.1038/s41586-021-04245-0
- Escalera A., Gonzalez-Reiche A.S., Aslam S., Mena I., Laporte M., Pearl R.L., et al. Mutations in SARS-CoV-2 variants of concern link to increased spike cleavage and virus transmission. Cell Host Microbe. 2022; 30(3): 373–87.e7. https://doi.org/10.1016/j.chom.2022.01.006
- Starr T.N., Greaney A.J., Hannon W.W., Loes A.N., Hauser K., Dillen J.R., et al. Shifting mutational constraints in the SARS-CoV-2 receptor-binding domain during viral evolution. Science. 2022; 377(6604): 420–4. https://doi.org/10.1126/science.abo7896
- Hirotsu Y., Maejima M., Shibusawa M., Natori Y., Nagakubo Y., Hosaka K., et al. SARS-CoV-2 Omicron sublineage BA.2 replaces BA.1.1: Genomic surveillance in Japan from September 2021 to March 2022. J. Infect. 2022; 85(2): 174–211. https://doi.org/10.1016/j.jinf.2022.04.040
- Mastrorosa I., Cozzi-Lepri A., Colavita F., Lalle E., Mazzotta V., Cimaglia C., et al. SARS-CoV-2 nasopharyngeal viral load in individuals infected with BA.2, compared to Alpha, Gamma, Delta and BA.1 variants: A single-center comparative analysis. J. Clin. Virol. 2022; 157: 105299. https://doi.org/10.1016/j.jcv.2022.105299
- Tozer K., Sjaarda C.P., Moslinger E., Wong H., Mubareka S., Maguire F., et al. Comparison of SARS-CoV-2 viral loads in the nasal mucosa of patients infected with BA.1, BA.2, or BA.5 Omicron lineages. Open Forum Infect. Dis. 2022; 9(12): ofac564. https://doi.org/10.1093/ofid/ofac564
- Takatsuki Y., Takahashi Y., Nakajima J., Iwasaki Y., Nagano K., Tani-Sassa C., et al. Viral load of SARS-CoV-2 Omicron BA.5 is lower than that of BA.2 despite the higher infectivity of BA.5. Immun. Inflamm. Dis. 2023; 11(2): e783. https://doi.org/10.1002/iid3.783
- Cao Y., Yisimayi A., Jian F., Song W., Xiao T., Wang L., et al. BA.2.12.1, BA.4 and BA.5 escape antibodies elicited by Omicron infection. Nature. 2022; 608(7923): 593–602. https://doi.org/10.1038/s41586-022-04980-y
- Tuekprakhon A., Nutalai R., Dijokaite-Guraliuc A., Zhou D., Ginn H.M., Selvaraj M., et al. Antibody escape of SARS-CoV-2 Omicron BA.4 and BA.5 from vaccine and BA.1 serum. Cell. 2022; 185(14): 2422–33. https://doi.org/10.1016/j.cell.2022.06.005
- Rashid F., Xie Z., Suleman M., Shah A., Khan S., Luo S. Roles and functions of SARS-CoV-2 proteins in host immune evasion. Front. Immunol. 2022; 13: 940756. https://doi.org/10.3389/fimmu.2022.940756
- Github. BA.5.1.29 sublineage with S:K150E, S:460K, Orf8:F41C. Available at: https://github.com/cov-lineages/pango-designation/issues/1187
- Khare S., Gurry C., Freitas L., Schultz M.B., Bach G., Diallo A., et al. GISAID’s role in pandemic response. China CDC Wkly. 2021; 3(49): 1049–51. https://doi.org/10.46234/ccdcw2021.255
- Haslwanter D., Dieterle M.E., Wec A.Z., O’Brien C.M., Sakharkar M., Florez C., et al. A combination of receptor-binding domain and N-terminal domain neutralizing antibodies limits the generation of SARS-CoV-2 spike neutralization-escape mutants. mBio. 2021; 12(5): e0247321. https://doi.org/10.1128/mBio.02473-21
- Kee J., Thudium S., Renner D.M., Glastad K., Palozola K., Zhang Z., et al. SARS-CoV-2 disrupts host epigenetic regulation via histone mimicry. Nature. 2022; 610(7931): 381–8. https://doi.org/10.1038/s41586-022-05282-z
- Hossain A., Akter S., Rashid A.A., Khair S., Alam A.S.M.R.U. Unique mutations in SARS-CoV-2 Omicron subvariants’ non-spike proteins: Potential impacts on viral pathogenesis and host immune evasion. Microb. Pathog. 2022; 170: 105699. https://doi.org/10.1016/j.micpath.2022.105699
- Github. BJ.1/BM.1.1.1 (=BA.2.75.3.1.1.1) recombinant with breakpoint in S1 (>=5 sequences, 3x Singapore, 2x US as of 2022-09-12). Available at: https://github.com/cov-lineages/pango-designation/issues/1058
- Tamura T., Ito J., Uriu K., Zahradnik J., Kida I., Anraku Y., et al. Virological characteristics of the SARS-CoV-2 XBB variant derived from recombination of two Omicron subvariants. Nat. Commun. 2023; 14(1): 2800. https://doi.org/10.1038/s41467-023-38435-3
- Ao D., He X., Hong W., Wei X. The rapid rise of SARS-CoV-2 Omicron subvariants with immune evasion properties: XBB.1.5 and BQ.1.1 subvariants. MedComm. (2020). 2023; 4(2): e239. https://doi.org/10.1002/mco2.239
- Parums D.V. Editorial: The XBB.1.5 (‘Kraken’) subvariant of Omicron SARS-CoV-2 and its rapid global spread. Med. Sci. Monitor. 2023; 29: e939580. https://doi.org/10.12659/MSM.939580
Supplementary files