Филогенетический анализ вариантов вируса Пуумала (Hantaviridae: Orthohantavirus), циркулирующих на территории Саратовской области
- Авторы: Краснов Я.М.1, Найденова Е.В.1, Гусева Н.П.1, Полунина Т.А.1, Шарапова Н.А.1, Соседова Е.А.1, Котова Н.В.1, Захаров К.С.1, Казанцев А.В.1, Доманина И.В.1, Чекашов В.Н.1, Шилов М.М.1, Кондратьев Е.Н.1, Осина Н.А.1, Кутырев В.В.1
-
Учреждения:
- ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
- Выпуск: Том 69, № 2 (2024)
- Страницы: 162-174
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://ogarev-online.ru/0507-4088/article/view/256877
- DOI: https://doi.org/10.36233/0507-4088-224
- EDN: https://elibrary.ru/ijmubu
- ID: 256877
Цитировать
Аннотация
Цель работы – определение полной нуклеотидной последовательности и проведение филогенетического анализа вариантов геномов вируса Пуумала, выделенных на территории Саратовской области.
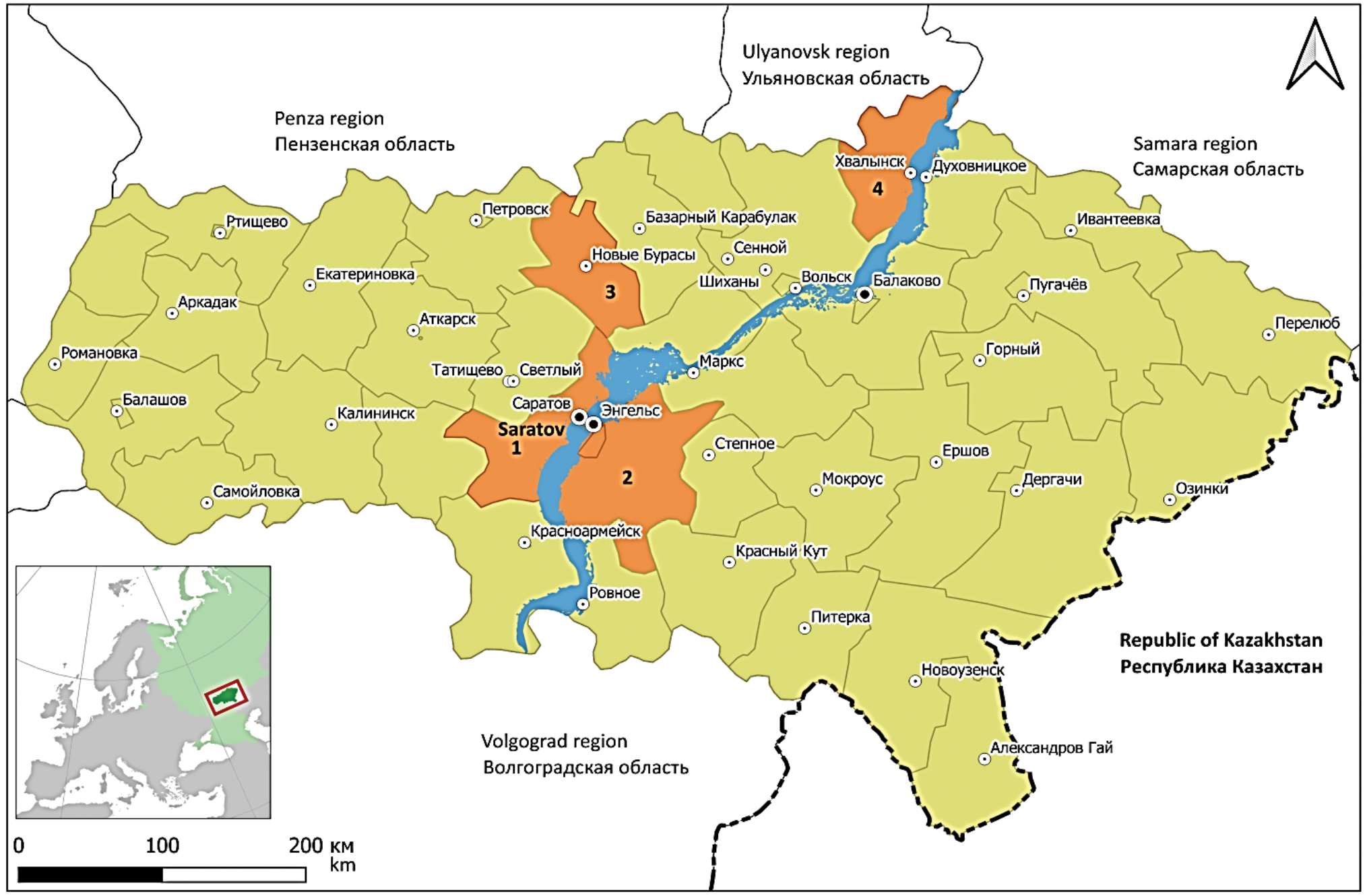
Материалы и методы. Образцами для исследования послужил полевой материал, собранный в Гагаринском (бывшем Саратовском), Энгельсском, Новобурасском и Хвалынском районах Саратовской области в период с 2019 по 2022 г. Для специфического обогащения генома вируса Пуумала в образцах использовали ПЦР и панель праймеров, подготовленную для данного исследования. Далее проводили секвенирование полученных продуктов реакции и сборку фрагментов в одну последовательность для каждого из сегментов генома вируса. При построении филогенетических деревьев применяли алгоритм maximum parsimony.
Результаты. Показано, что генетические варианты вируса Пуумала, выделенные в Саратовской области, имеют высокую степень подобия генома, что говорит о единстве их происхождения. По данным филогенетического анализа, все выделенные варианты вируса (за исключением изолятов вируса из Хвалынского района) образуют обособленную ветвь в кластере, сформированном хантавирусами из других субъектов Приволжского федерального округа. Самыми близкими к образцам из Саратовской области являются варианты вируса из республик Удмуртия и Татарстан, а также из Самарской и Ульяновской областей.
Заключение. Полученные данные указывают на наличие выраженной территориальной приуроченности штаммов к определенным регионам или областям, являющимся природными биотопами их носителей. Этот факт позволяет довольно точно определять территорию возможного инфицирования заболевших и/или циркуляцию переносчиков данных вариантов вируса по последовательности отдельных сегментов их генома.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Ярослав Михайлович Краснов
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4909-2394
кандидат хим. наук, ведущий научный сотрудник
Россия, 410005, СаратовЕкатерина Владимировна Найденова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Автор, ответственный за переписку.
Email: katim2003@mail.ru
ORCID iD: 0000-0001-6474-3696
кандидат биологических наук, ведущий научный сотрудник
Россия, 410005, СаратовНаталья Петровна Гусева
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0003-3763-9708
кандидат биол. наук, старший научный сотрудник
Россия, 410005, СаратовТатьяна Алексеевна Полунина
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@mail.ru
ORCID iD: 0000-0002-2234-2760
кандидат мед. наук, старший научный сотрудник
Россия, 410005, СаратовНаталья Анатольевна Шарапова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-5289-7783
кандидат биол. наук, научный сотрудник
Россия, 410005, СаратовЕкатерина Анатольевна Соседова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0009-0004-4443-2646
научный сотрудник
Россия, 410005, СаратовНина Владимировна Котова
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-9270-523X
научный сотрудник
Россия, 410005, СаратовКирилл Сергеевич Захаров
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: zaharov_ks@mail.ru
ORCID iD: 0000-0002-4726-309X
кандидат биол. наук, старший научный сотрудник
Россия, 410005, СаратовАндрей Владимирович Казанцев
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: andreikazancev@mail.ru
ORCID iD: 0000-0003-1790-0411
научный сотрудник
Россия, 410005, СаратовИрина Владимировна Доманина
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4731-8089
научный сотрудник
Россия, 410005, СаратовВладимир Николаевич Чекашов
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-9593-4353
кандидат биол. наук, старший научный сотрудник
Россия, 410005, СаратовМихаил Михайлович Шилов
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-0083-8212
кандидат биол. наук, научный сотрудник
Россия, 410005, СаратовЕвгений Николаевич Кондратьев
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-7508-4355
научный сотрудник
Россия, 410005, СаратовНаталья Александровна Осина
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: davidova_n_work@mail.ru
ORCID iD: 0000-0003-0954-5683
кандидат биол. наук, заведующий отделом микробиологии
Россия, 410005, СаратовВладимир Викторович Кутырев
ФКУН «Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора
Email: rusrapi@mail.ru
ORCID iD: 0000-0003-3788-3452
академик РАН, профессор, директор
Россия, 410005, СаратовСписок литературы
- Савицкая Т.А., Иванова А.В., Исаева Г.Ш., Решетникова И.Д., Трифонов В.А., Зиатдинов В.Б. и др. Анализ эпидемиологической ситуации по геморрагической лихорадке с почечным синдромом в Российской Федерации в 2022 г. и прогноз ее развития на 2023 г. Проблемы особо опасных инфекций. 2023; (1): 85–95. https://doi.org/10.21055/0370-1069-2023-1-85-95 https://elibrary.ru/mgxnza
- Иванова А.В., Сафронов В.А., Попов Н.В., Куклев Е.В. Эпидемиологическое районирование территории Приволжского федерального округа по уровню потенциальной эпидемической опасности природных очагов геморрагической лихорадки с почечным синдромом. Проблемы особо опасных инфекций. 2020; (1): 91–6. https://doi.org/10.21055/0370-1069-2020-1-91-96 https://elibrary.ru/jrywjk
- Чумачкова Е.А., Иванова А.В., Поршаков А.М., Вяткин И.Н., Форостяная М.В., Чумачков К.Я. и др. Районирование территории Саратовской области по интенсивности эпидемических проявлений ГЛПС с использованием ГИС-анализа. Проблемы особо опасных инфекций. 2023; (3): 156–63. https://doi.org/10.21055/0370-1069-2023-3-156-163 https://elibrary.ru/dwrlpq
- Tkachenko E.A., Ishmukhametov A.A., Dzagurova T.K., Bernshtein A.D., Morozov V.G., Siniugina A.A., et al. Hemorrhagic fever with renal syndrome, Russia. Emerg. Infect. Dis. 2019; 25(12): 2325–8. https://doi.org/10.3201/eid2512.181649
- Ишмухаметов А.А., Дзагурова Т.К., Морозов В.Г., Курашова С.С., Баловнева М.В., Соцкова С.Е. и др. Характеристика хантавирусов – возбудителей зоонозных геморрагических лихорадок. Эпидемиология и вакцинопрофилактика. 2017; 16(3): 26–32. https://doi.org/10.31631/2073-3046-2017-16-3-26-32 https://elibrary.ru/yrhmch
- ICTV. Taxonomy Browser. Available at: https://ictv.global/taxonomy
- Kabwe E., Davidyuk Y., Shamsutdinov A., Garanina E., Martynova E., Kitaeva K., et al. Orthohantaviruses, emerging zoonotic pathogens. Pathogens. 2020; 9(9): 775. https://doi.org/10.3390/pathogens9090775
- Яшина Л.Н., Трегубчак Т.В., Малышев Б.С., Сметанникова Н.А., Грищенко И.В., Дольский А.А. и др. Возбудитель вспышки ГЛПС в Саратовской области, 2019 г. Проблемы особо опасных инфекций. 2021; (4): 150–6. https://doi.org/10.21055/0370-1069-2021-4-150-156 https://elibrary.ru/dxxsey
- Davidyuk Y.N., Kabwe E., Shamsutdinov A.F., Knyazeva A.V., Martynova E.V., Ismagilova R.K., et al. The Distribution of Puumala orthohantavirus genome variants correlates with the regional landscapes in the Trans-Kama area of the Republic of Tatarstan. Pathogens. 2021; 10(9): 1169. https://doi.org/10.3390/pathogens10091169
- Kabwe E., Shamsutdinov A.F., Suleimanova S., Martynova E.V., Ismagilova R.K., Shakirova V.G., et al. Puumala orthohantavirus reassortant genome variants likely emerging in the watershed forests. Int. J. Mol. Sci. 2023; 24(2): 1018. https://doi.org/10.3390/ijms24021018
- Kabwe E., Al Sheikh W., Shamsutdinov A.F., Ismagilova R.K., Martynova E.V., Ohlopkova O.V., et al. Analysis of Puumala orthohantavirus genome variants identified in the territories of Volga Federal District. Trop. Med. Infect. Dis. 2022; 7(3): 46. https://doi.org/10.3390/tropicalmed7030046
- Blinova E., Deviatkin A., Makenov M., Popova Y., Dzagurova T. Evolutionary formation and distribution of Puumala virus genome variants, Russia. Emerg. Infect. Dis. 2023; 29(7): 1420–4. https://doi.org/10.3201/eid2907.221731
- Castel G., Chevenet F., Razzauti M., Murri S., Marianneau P., Cosson J.F., et al. Phylogeography of Puumala orthohantavirus in Europe. Viruses. 2019; 11(8): 679. https://doi.org/10.3390/v11080679
- Souza W.M., Bello G., Amarilla A.A., Alfonso H.L., Aquino V.H., Figueiredo L.T. Phylogeography and evolutionary history of rodent-borne hantaviruses. Infect. Genet. Evol. 2014; 21: 198–204. https://doi.org/10.1016/j.meegid.2013.11.015
- Ramsden C., Holmes E.C., Charleston M.A. Hantavirus evolution in relation to its rodent and insectivore hosts: no evidence for codivergence. Mol. Biol. Evol. 2009; 26(1): 143–53. https://doi.org/10.1093/molbev/msn234
Дополнительные файлы