Detection SARS-CoV-2 (Coronaviridae: Coronavirinae: Betacoronavirus: Sarbecovirus) in children with acute intestinal infection in Nizhny Novgorod during 2020–2021
- Authors: Morozova O.V.1, Novikova N.A.1, Epifanova N.V.1, Novikov D.V.1, Mokhonov V.V.1, Sashina T.A.1, Zaytseva N.N.1
-
Affiliations:
- FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
- Issue: Vol 67, No 1 (2022)
- Pages: 69-76
- Section: ORIGINAL RESEARCHES
- URL: https://ogarev-online.ru/0507-4088/article/view/118213
- DOI: https://doi.org/10.36233/0507-4088-95
- ID: 118213
Cite item
Full Text
Abstract
Introduction. The novel coronavirus infection COVID-19 is a major public health problem worldwide. Several publications show the presence of gastrointestinal (GI) symptoms (nausea, vomiting, and diarrhea) in addition to respiratory disorders.
The aim of this study was the monitoring of RNA of COVID-19 pathogen, coronavirus SARS-CoV-2 (Coronaviridae: Coronavirinae: Betacoronavirus; Sarbecovirus) in children hospitalized with acute intestinal infection (AII), with following molecular-genetic characterization of detected strains.
Material and methods. Fecal samples of children with AII hospitalized in infectious hospital of Nizhny Novgorod (Russia) in the period from 01.07.2020 to 31.10.2021 were used as material for the study. Viral RNA detection was performed by real-time polymerase chain reaction (RT-PCR). The nucleotide sequence of S-protein gene fragment was determined by Sanger sequencing.
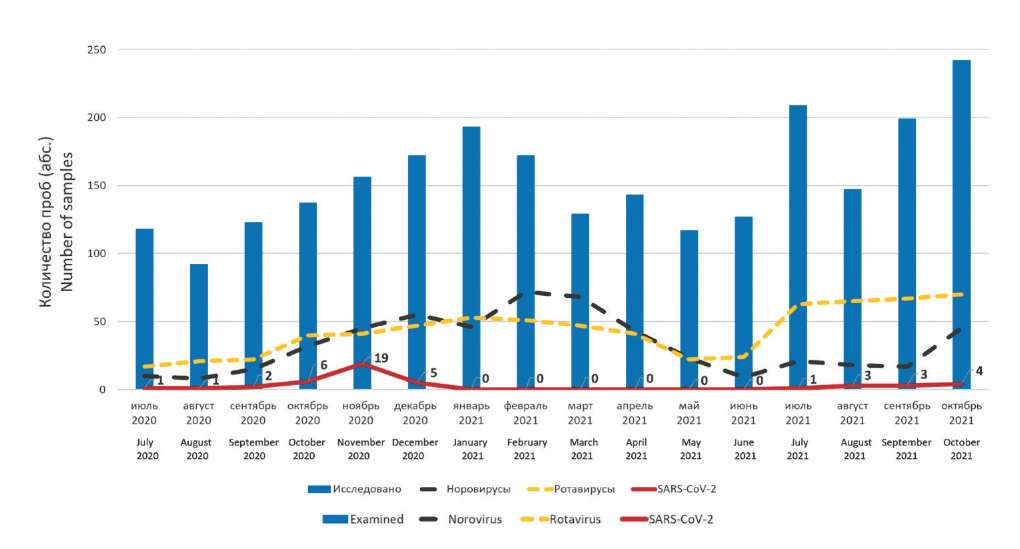
Results and discussion. SARS-CoV-2 genetic material was detected in 45 out of 2476 fecal samples. The maximum number of samples containing RNA of the virus occurred in November 2020 (detection rate of 12.2%). In 20.0% of cases, SARS-CoV-2 RNA was detected in combination with rota-, noro-, and adenoviruses. 28 nucleotide sequences of S-protein gene fragment complementary DNA (cDNA) were determined. Phylogenetic analysis showed that the studied SARS-CoV-2 strains belonged to two variants. Analysis of the S-protein amino acid sequence of the strains studied showed the absence of the N501Y mutation in the 2020 samples, which is a marker for variants with a high epidemic potential, called variants of concern (VOC) according to the World Health Organization (WHO) definition (lines Alpha B.1.1.7, Beta B.1.351, Gamma P.1). Delta line variant B.1.617.2 was identified in two samples isolated in September 2021.
Conclusion. The detection of SARS-CoV-2 RNA in the fecal samples of children with AII, suggesting that the fecal-oral mechanism of pathogen transmission may exist, determines the necessity to optimize its monitoring and to develop an algorithm of actions with patients with signs of AII under the conditions of a novel coronavirus infection pandemic.
Full Text
##article.viewOnOriginalSite##About the authors
O. V. Morozova
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Author for correspondence.
Email: olga.morozova.bsc@gmail.com
ORCID iD: 0000-0002-8058-8187
Olga V. Morozova, Ph.D., Researcher of the Molecular Epidemiology of Viral Infections Laboratory
603950, Nizhny Novgorod
Russian FederationN. A. Novikova
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-3710-6648
603950, Nizhny Novgorod
Russian FederationN. V. Epifanova
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0001-7679-8029
603950, Nizhny Novgorod
Russian FederationD. V. Novikov
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0001-7049-6935
603950, Nizhny Novgorod
Russian FederationV. V. Mokhonov
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-8542-5723
603950, Nizhny Novgorod
Russian FederationT. A. Sashina
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0003-3203-7863
603950, Nizhny Novgorod
Russian FederationN. N. Zaytseva
FSBI «Academician I.N. Blokhina Nizhny Novgorod Scientific Research Institute of Epidemiology and Microbiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0001-5370-4026
603950, Nizhny Novgorod
Russian FederationReferences
- Xiang Y.T., Yang Y., Li W., Zhang L., Zhang Q., Cheung T., et al. Timely mental health care for the 2019 novel coronavirus outbreak is urgently needed. Lancet Psychiatry. 2020; 7(3): 228–9. https://doi.org/10.1016/S2215-0366(20)30046-8
- Holshue M.L., DeBolt C., Lindquist S., Lofy K.H., Wiesman J., Bruce H., et al. First case of 2019 novel coronavirus in the United States. N. Engl. J. Med. 2020; 382(10): 929–36. https://doi.org/10.1056/NEJMoa2001191
- Leung W.K., To K.F., Chan P.K., Chan H.L.Y., Wu A.K.L., Lee N., et al. Enteric involvement of severe acute respiratory syndromeassociated coronavirus infection. Gastroenterology. 2003; 125(4): 1011–7. https://doi.org/10.1016/s0016-5085(03)01215-0
- Wang X.W., Li J.S., Guo T.K., Zhen B., Kong Q.X., Yi B., et al. Concentration and detection of SARS coronavirus in sewage from Xiao Tang Shan Hospital and the 309th Hospital. J. Virol. Methods. 2005; 128(1-2): 156–61. https://doi.org/10.1016/j.jviromet.2005.03.022
- Puelles V.G., Lütgehetmann M., Lindenmeyer M.T., Sperhake J.P., Wong M.N., Allweiss L., et al. Multiorgan and renal tropism of SARS-CoV-2. N. Engl. J. Med. 2020; 383(6): 590–2. https://doi.org/10.1056/NEJMc2011400
- Wu F., Zhao S., Yu B., Chen Y.M., Wang W., Song Z.G., et al. A new coronavirus associated with human respiratory disease in China. Nature. 2020; 579(7798): 265–9. https://doi.org/10.1038/s41586-020-2008-3
- Burgueño J.F., Reich A., Hazime H., Quintero M.A., Fernandez I., Fritsch J., et al. Expression of SARS-CoV-2 entry molecules ACE2 and TMPRSS2 in the gut of patients with IBD. Inflamm. Bowel. Dis. 2020; 26(6): 797e808. https://doi.org/10.1093/ibd/izaa085
- Harmer D., Gilbert M., Borman R., Clark K.L. Quantitative mRNA expression profiling of ACE 2, a novel homologue of angiotensin converting enzyme. FEBS Lett. 2002; 532(1-2): 107–10. https://doi.org/10.1016/s0014-5793(02)03640-2
- Xiao F., Tang M., Zheng X., Liu Y., Li X., Shan H. Evidence for gastrointestinal infection of SARS-CoV-2. Gastroenterology. 2020; 158(6): 1831–3.e3. https://doi.org/10.1053/j.gastro.2020.02.055
- Du M., Cai G., Chen F., Christiani D.C., Zhang Z., Wang M. Multi-omics evaluation of gastrointestinal and other clinical characteristics of SARS-CoV-2 and COVID-19. Gastroenterology. 2020; 158(8): 2298–301. https://doi.org/10.1053/j.gastro.2020.03.045
- Tian Y., Rong L., Nian W., He Y. Review article: gastrointestinal features in COVID-19 and the possibility of faecal transmission. Aliment. Pharmacol. Ther. 2020; 51(9): 843–51. https://doi.org/10.1111/apt.15731
- Wang D., Hu B., Hu C., Zhu F., Liu X., Zhang J., et al. Clinical characteristics of 138 hospitalized patients with 2019 novel coronavirus- infected pneumonia in Wuhan, China. JAMA. 2020; 323(11): 1061–9. https://doi.org/10.1001/jama.2020.1585
- Vespa E., Pugliese N., Colapietro F., Aghemo A. Stay (GI) Healthy: COVID-19 and gastrointestinal manifestations. Tech. Innov. Gastrointest. Endosc. 2021; 23(2): 179–89. https://doi.org/10.1016/j.tige.2021.01.006
- An P., Chen H., Ren H., Su L., Ji M., Kang J., et al. Gastrointestinal symptoms onset in COVID-19 patients in Wuhan, China. Dig. Dis. Sci. 2020; 66(10): 3578–87. https://doi.org/10.1007/s10620-020-06693-6
- Xu Y., Li X., Zhu B., Liang H., Fang C., Gong Y., et al. Characteristics of pediatric SARS-CoV-2 infection and potential evidence for persistent fecal viral shedding. Nat. Med. 2020; 26(4): 502–5. https://doi.org/10.1038/s41591-020-0817-4
- Yang Z., Li G., Dai X., Liu G., Li G., Jie Y. Three cases of novel coronavirus pneumonia with viral nucleic acids still positive in stool after throat swab detection turned negative. Chin. J. Dig. 2020; 40: E002– E002. https://doi.org/10.3760/cma.j.issn.0254-1432.2020.0002 (in Chinese)
- Ling Y., Xu S.B., Lin Y.X., Tian D., Zhu Z.Q., Dai F.H., et al. Persistence and clearance of viral RNA in 2019 novel coronavirus disease rehabilitation patients. Chin. Med. J. (Engl.). 2020; 133(9): 1039–43. https://doi.org/10.1097/CM9.0000000000000774
- Zhang J.J., Dong X., Cao Y.Y., Yuan Y.D., Yang Y.B., Yan Y.Q., et al. Clinical characteristics of 140 patients infected with SARSCoV- 2 in Wuhan, China. Allergy. 2020; 75(7): 1730–41. https://doi.org/10.1111/all.14238
- Xiao F., Sun J., Xu Y., Li F., Huang X., Li H., et al. Infectious SARSCoV-2 in feces of patient with severe COVID-19. Emerg. Infect. Dis. 2020; 26(8): 1920–2. https://doi.org/10.3201/eid2608.200681
- VGARus. Russian platform for aggregating information about virus genomes. Available at: https://genome.crie.ru/app/index (accessed December 29, 2021).
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across Computing Platforms. Mol. Biol. Evol. 2018; 35(6): 1547–9. https://doi.org/10.1093/molbev/msy096
- Jevšnik M., Steyer A., Zrim T., Pokorn M., Mrvič T., Grosek Š., et al. Detection of human coronaviruses in simultaneously collected stool samples and nasopharyngeal swabs from hospitalized children with acute gastroenteritis. Virol. J. 2013; 10: 46. https://doi.org/10.1186/1743-422X-10-46
- Paloniemi M., Lappalainen S., Vesikari T. Commonly circulating human coronaviruses do not have a significant role in the etiology of gastrointestinal infections in hospitalized children. J. Clin. Virol. 2015; 62: 114–7. https://doi.org/10.1016/j.jcv.2014.10.017
- Leung T.F., Chan P.K., Wong W.K., Ip M., Cheng W.T., Ng P.C. Human coronavirus NL63 in children: epidemiology, disease spectrum, and genetic diversity. Hong Kong Med. J. 2012; 18(Suppl. 2): 27–30.
- Abdelghany T.M., Ganash M., Bakri M.M., Qanash H., Al-Rajhi A.M.H., Elhussieny N.I. SARS-CoV-2, the other face to SARSCoV and MERS-CoV: Future predictions. Biomed. J. 2021; 44(1): 86–93. https://doi.org/10.1016/j.bj.2020.10.008
- Laxminarayan R., Mohan C.B., Vinay T.G., Arjun Kumar K.V., Wahl B., Lewnard J.A. SARS-CoV-2 infection and mortality during the first epidemic wave in Madurai, south India: a prospective, active surveillance study. Lancet Infect. Dis. 2021; 21(12): 1665–76. https://doi.org/10.1016/S1473-3099(21)00393-5
- Zhou J., Li C., Zhao G., Chu H., Wang D., Yan H.H., et al. Human intestinal tract serves as an alternative infection route for Middle East respiratory syndrome coronavirus. Sci. Adv. 2017; 3(11): eaao4966. https://doi.org/10.1126/sciadv.aao4966
- Donnelly C.A., Ghani A.C., Leung G.M., Hedley A.J., Fraser C., Riley S., et al. Epidemiological determinants of spread of causal agent of severe acute respiratory syndrome in Hong Kong. Lancet. 2003; 361(9371): 1761–6. https://doi.org/10.1016/S0140-6736(03)13410-1
- Booth C.M., Matukas L.M., Tomlinson G.A., Rachlis A.R., Rose D.B., Dwosh H.A., et al. Clinical features and short-term outcomes of 144 patients with SARS in the greater Toronto area. JAMA. 2003; 289(21): 2801–9. https://doi.org/10.1001/jama.289.21.JOC30885
- Lee N., Hui D., Wu A., Chan P., Cameron P., Joynt G.M., et al. A major outbreak of severe acute respiratory syndrome in Hong Kong. N. Engl. J. Med. 2003; 348(20): 1986–94. https://doi.org/10.1056/NEJMoa030685
- Stockman L.J., Massoudi M.S., Helfand R., Erdman D., Siwek A.M., Anderson L.J., et al. Severe acute respiratory syndrome in children. Pediatr. Infect. Dis. J. 2007; 26(1): 68–74. https://doi.org/10.1097/01.inf.0000247136.28950.41
- Ding Y., He L., Zhang Q., Huang Z., Che X., Hou J., et al. Organ distribution of severe acute respiratory syndrome (SARS) associated coronavirus (SARS-CoV) in SARS patients: implications for pathogenesis and virus transmission pathways. J. Pathol. 2004; 203(2): 622–30. https://doi.org/10.1002/path.1560
- Moura I.B., Buckley A.M., Wilcox M.H. Can SARS-CoV-2 be transmitted via faeces? Curr. Opin. Gastroenterol. 2022; 38(1): 26–9. https://doi.org/10.1097/MOG.0000000000000794
- D’Amico F., Baumgart D.C., Danese S., Peyrin-Biroulet L. Diarrhea during COVID-19 infection: Pathogenesis, epidemiology, prevention, and management. Clin. Gastroenterol. Hepatol. 2020; 18(8): 1663–72. https://doi.org/10.1016/j.cgh.2020.04.001
- Fratev F. N501Y and K417N mutations in the spike protein of SARS-CoV-2 alter the interactions with both hACE2 and human- derived antibody: a free energy of perturbation retrospective study. J. Chem. Inf. Model. 2021; 61(12): 6079–84. https://doi.org/10.1021/acs.jcim.1c01242
Supplementary files