DNA mapping in the capsid of giant bacteriophage phiEL (Caudovirales: Myoviridae: Elvirus) by analytical electron microscopy
- Authors: Trifonova T.S.1,2, Moiseenko A.V.2,3, Bourkaltseva M.V.4, Shaburova O.V.4, Shaytan A.K.2, Krylov V.N.4, Sokolova O.S.2
-
Affiliations:
- FSAEI HE «People’s Friendship University of Russia», Physical, Mathematical, and Natural Sciences Department
- FSBEI HE «Lomonosov Moscow State University», Bioengineering Department, Biological Faculty
- FSBIS «N.N. Semenov Federal Research Center for Chemical Physics, Russian Academy of Sciences»
- FSBRI «I.I. Mechnikov Research Institute of Vaccines and Sera»
- Issue: Vol 66, No 6 (2021)
- Pages: 434-441
- Section: ORIGINAL RESEARCHES
- URL: https://ogarev-online.ru/0507-4088/article/view/118210
- DOI: https://doi.org/10.36233/0507-4088-80
- ID: 118210
Cite item
Abstract
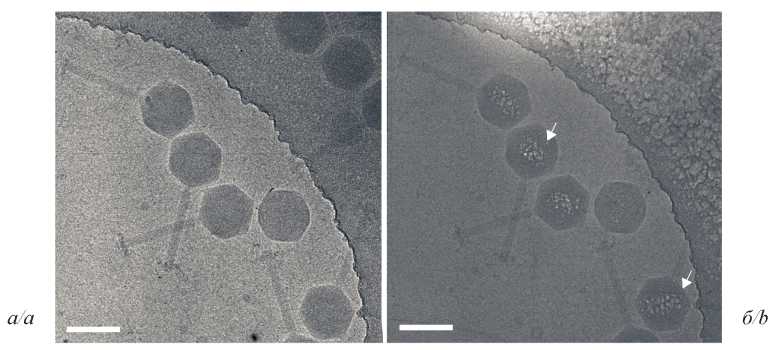
Introduction. Giant phiKZ-like bacteriophages have a unique protein formation inside the capsid, an inner body (IB) with supercoiled DNA molecule wrapped around it. Standard cryo-electron microscopy (cryo-EM) approaches do not allow to distinguish this structure from the surrounding nucleic acid of the phage. We previously developed an analytical approach to visualize protein-DNA complexes on Escherichia coli bacterial cell slices using the chemical element phosphorus as a marker. In the study presented, we adapted this technique for much smaller objects, namely the capsids of phiKZ-like bacteriophages.
Material and methods. Following electron microscopy techniques were used in the study: analytical (AEM) (electron energy loss spectroscopy, EELS), and cryo-EM (images of samples subjected to low and high dose of electron irradiation were compared).
Results. We studied DNA packaging inside the capsids of giant bacteriophages phiEL from the Myoviridae family that infect Pseudomonas aeruginosa. Phosphorus distribution maps were obtained, showing an asymmetrical arrangement of DNA inside the capsid.
Discussion. We developed and applied an IB imaging technique using a high angle dark-field detector (HAADF) and the STEM-EELS analytical approach. Phosphorus mapping by EELS and cryo-electron microscopy revealed a protein formation as IB within the phage phiEL capsid. The size of IB was estimated using theoretical calculations.
Conclusion. The developed technique can be applied to study the distribution of phosphorus in other DNA- or RNA-containing viruses at relatively low concentrations of the element sought.
Full Text
##article.viewOnOriginalSite##About the authors
T. S. Trifonova
FSAEI HE «People’s Friendship University of Russia», Physical, Mathematical, and Natural Sciences Department; FSBEI HE «Lomonosov Moscow State University», Bioengineering Department, Biological Faculty
ORCID iD: 0000-0003-2042-5244
115419, Moscow, Russia
119234, Moscow, Russia
Russian FederationA. V. Moiseenko
FSBEI HE «Lomonosov Moscow State University», Bioengineering Department, Biological Faculty; FSBIS «N.N. Semenov Federal Research Center for Chemical Physics, Russian Academy of Sciences»
ORCID iD: 0000-0003-1112-2356
119234, Moscow, Russia
119234, Moscow, Russia
Russian FederationM. V. Bourkaltseva
FSBRI «I.I. Mechnikov Research Institute of Vaccines and Sera»
ORCID iD: 0000-0003-3793-1354
105064, Moscow, Russia
Russian FederationO. V. Shaburova
FSBRI «I.I. Mechnikov Research Institute of Vaccines and Sera»
ORCID iD: 0000-0003-0368-3794
105064, Moscow, Russia
Russian FederationA. K. Shaytan
FSBEI HE «Lomonosov Moscow State University», Bioengineering Department, Biological Faculty
ORCID iD: 0000-0003-0312-938X
119234, Moscow, Russia
Russian FederationV. N. Krylov
FSBRI «I.I. Mechnikov Research Institute of Vaccines and Sera»
ORCID iD: 0000-0001-5775-5146
105064, Moscow, Russia
Russian FederationO. S. Sokolova
FSBEI HE «Lomonosov Moscow State University», Bioengineering Department, Biological Faculty
Author for correspondence.
Email: sokolova@mail.bio.msu.ru
ORCID iD: 0000-0003-4678-232X
Olga S. Sokolova, D.Sci. (Biol.), RAS Professor, Professor of Bioengineering Department, Biological Faculty
119234, Moscow, Russia
Russian FederationReferences
- Ochman H., Lawrence J., Groisman E. Lateral gene transfer and the nature of bacterial innovation. Nature. 2000; 405(6784): 299–304. https://doi.org/10.1038/35012500
- Duplessis C.A., Biswas B. A review of topical phage therapy for chronically infected wounds and preparations for a randomized adaptive clinical trial evaluating topical phage therapy in chronically infected diabetic foot ulcers. Antibiotics. 2020; 9(7): 377. https://doi.org/10.3390/antibiotics9070377
- Sharma R., Pielstick B., Bell K., Nieman T., Stubbs O., Yeates E., et al. A Novel, Highly Related Jumbo Family of Bacteriophages That Were Isolated Against Erwinia. Front. Microbiol. 2019; 10: 1533. https://doi.org/10.3389/fmicb.2019.01533
- Fokine A., Kostyuchenko V.A., Efimov A.V., Kurochkina L.P., Sykilinda N.N., Robben J., et al. A three-dimensional cryo-electron microscopy structure of the bacteriophage ϕKZ head. J. Mol. Biol. 2005; 352(1): 117–24. https://doi.org/10.1016/j.jmb.2005.07.018
- Sokolova O.S., Shaburova O.V., Pechnikova E.V., Shaytan A.K., Krylov S.V., Kiselev N.A., et al. Genome packaging in EL and Lin68, two giant phiKZ-like bacteriophages of P. aeruginosa. Virology. 2014; 468–470: 472–8. https://doi.org/10.1016/j.virol.2014.09.002
- Hertveldt K., Lavigne R., Pleteneva E., Sernova N., Kurochkina L., Korchevskii R., et al. Genome comparison of Pseudomonas aeruginosa large phages. J. Mol. Biol. 2005; 354(3): 536–45. https://doi.org/10.1016/j.jmb.2005.08.075
- Mesyanzhinov V.V., Robben J., Grymonprez B., Kostyuchenko V.A., Bourkaltseva M.V., Sykilinda N.N., et al. The genome of bacteriophage phiKZ of Pseudomonas aeruginosa. J. Mol. Biol. 2002; 317(1): 1–19. https://doi.org/10.1006/jmbi.2001.5396
- Thomas J.A., Rolando M.R., Carroll C.A., Shen P.S., Belnap D.M., Weintraub S.T., et al. Characterization of Pseudomonas chlororaphis myovirus 201ϕ2-1 via genomic sequencing, mass spectrometry, and electron microscopy. Virology. 2008; 376(2): 330–8. https://doi.org/10.1016/j.virol.2008.04.004
- Krylov V.N., Smirnova T.A., Minenkova I.B., Plotnikova T.G., Zhazikov I.Z., Khrenova E.A. Pseudomonas bacteriophage contains an inner body in its capsid. Can. J. Microbiol. 1984; 30(6): 758–62. https://doi.org/10.1139/m84-116
- Wu W., Thomas J., Naiqian C., Black L., Steven A.C. Bubblegrams reveal the inner body of bacteriophage phiKZ. Science. 2012; 335(6065): 182. https://doi.org/10.1126/science.1214120
- Yakunina M., Artamonova T., Borukhov S., Makarova K.S., Severinov K., Minakhin L. A non-canonical multisubunit RNA polymerase encoded by a giant bacteriophage. Nucleic Acids res. 2015; 43(21): 10411–20. https://doi.org/10.1093/nar/gkv1095
- Danilova Y.A., Belousova V.V., Moiseenko A.V., Vishnyakov I.E., Yakunina M.V., Sokolova O.S. Maturation of Pseudo-Nucleus Compartment in P. aeruginosa, Infected with Giant phiKZ Phage. Viruses. 2020; 12(10): 1197. https://doi.org/10.3390/v12101197
- Matsko N., Klinov D., Manykin A., Demin V., Klimenko S. Atomic force microscopy analysis of bacteriophages phiKZ and T4. J. Electron. Microsc. (Tokyo). 2001; 50(5): 417–22. https://doi.org/10.1093/jmicro/50.5.417
- Fontana J., Jurado K.A., Cheng N., Ly N.L., Fuchs J.R., Gorelick R.J., et al. Distribution and Redistribution of HIV-1 Nucleocapsid Protein in Immature, Mature, and Integrase-Inhibited Virions: a Role for Integrase in Maturation. J. Virol. 2015; 89(19): 9765–80. https://doi.org/10.1128/JVI.01522-15
- Wu W., Leavitt J.C., Cheng N., Gilcrease E.B., Motwani T., Teschke C.M., et al. Localization of the houdinisome (Ejection Proteins) inside the bacteriophage P22 virion by bubblegram imaging. mBio. 2016; 7(4): e01152–16. https://doi.org/10.1128/mBio.01152-16
- Wu W., Newcomb W.W., Cheng N., Aksyuk A., Winkler D.C., Steven A.C. Internal Proteins of the Procapsid and Mature Capsids of Herpes Simplex Virus 1 Mapped by Bubblegram Imaging. J. Virol. 2016; 90(10): 5176–86. https://doi.org/10.1128/JVI.03224-15
- Shebanova A., Ismagulova T., Solovchenko A., Baulina O., Lobakova E., Ivanova A., et al. Versatility of the green microalga cell vacuole function as revealed by analytical transmission electron microscopy. Protoplasma. 2017; 254(3): 1323–40. https://doi.org/10.1007/s00709-016-1024-5
- Scotuzzi M., Kuipers J., Wensveen D.I., De Boer P., Hagen K.C.W., Hoogenboom J.P., et al. Multi-color electron microscopy by element- guided identification of cells, organelles and molecules. Sci. Rep. 2017; 7: 45970. https://doi.org/10.1038/srep45970
- Allard-Vannier E., Hervé-Aubert K., Kaaki K., Blondy T., Shebanova A., Shaitan K.V., et al. Folic acid-capped PEGylated magnetic nanoparticles enter cancer cells mostly via clathrin-dependent endocytosis. Biochim. Biophys. Acta Gen. Subj. 2017; 1861(6): 1578–86. https://doi.org/10.1016/j.bbagen.2016.11.045
- Loiko N., Danilova Y., Moiseenko A., Kovalenko V., Tereshkina K., Tutukina M., et al. Morphological peculiarities of the DNA-protein complexes in starved Escherichia coli cells. PLoS One. 2020; 15(10): e0231562. https://doi.org/10.1371/journal.pone.0231562
- Bazett-Jones D.P., Ottensmeyer F.P. Phosphorus distribution in the nucleosome. Science. 1981; 211(4478): 169–70. https://doi.org/10.1126/science.7444457
- Ottensmeyer F.P., Andrew J.W. High-resolution microanalysis of biological specimens by electron energy loss spectroscopy and by electron spectroscopic imaging. J. Ultrastruct. Res. 1980; 72(3):336–48. https://doi.org/10.1016/s0022-5320(80)90069-6
- Aronova M.A., Kim Y.C., Harmon R., Sousa A.A., Zhang G., Leapman R.D. Three-dimensional elemental mapping of phosphorus by quantitative electron spectroscopic tomography (QuEST). J. Struct. Biol. 2007; 160(1): 35–48. https://doi.org/10.1016/j.jsb.2007.06.008
- Nevsten P., Evilevitch A., Wallenberg R. Chemical mapping of DNA and counter-ion content inside phage by energy-filtered TEM. J. Biol. Phys. 2012; 38(2): 229–40. https://doi.org/10.1007/s10867-011-9234-8
- Sambrook J., Fritsch E.F., Maniatis T. Molecular Cloning: a Laboratory Manual. New York: Cold Spring Harbor Laboratory Press; 1989.
- Печникова Е.В., Кирпичников М.П., Соколова О.С. Радиационные повреждения в криомикроскопии: всегда ли во вред? Природа. 2015; (3): 25–9.
- Mishyna M., Volokh O., Danilova Ya., Gerasimova N., Pechnikova E., Sokolova O.S. Effects of radiation damage in studies of protein-DNA complexes by cryo-EM. Micron. 2017; 96: 57–64. https://doi.org/10.1016/j.micron.2017.02.004
- Petrov A.S., Harvey S.C. Packaging double-helical DNA into viral capsids: structures, forces, and energetics. Biophys. J. 2008; 95(2):497–502. https://doi.org/10.1529/biophysj.108.131797
- Буркальцева М.В., Крылов В.Н., Плетенева Е.А., Шабурова О.В., Крылов С.В., Волкарт Г., и др. Феногенетическая характеристика группы гигантских φKZ-подобных бактериофагов Pseudomonas aeruginosa. Генетика. 2002; 38(11): 1470–9.
- Bagrov D.V., Glukhov G.S., Moiseenko A.V., Karlova M.G., Litvinov D.S., Zaitsev P.А., et al. Structural characterization of β-propiolactone inactivated severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) particles. Microsc. Res. Tech. 2021. https://doi.org/10.1002/jemt.23931
Supplementary files