Вирус Эпштейна-Барр (Herpesviridae: Gammaherpesvirinae: Lymphocryptovirus: Human gammaherpesvirus 4): репликативные стратегии
- Авторы: Якушина С.А.1, Кистенева Л.Б.1
-
Учреждения:
- ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
- Выпуск: Том 65, № 4 (2020)
- Страницы: 191-202
- Раздел: ОБЗОРЫ
- URL: https://ogarev-online.ru/0507-4088/article/view/118120
- DOI: https://doi.org/10.36233/0507-4088-2020-65-4-191-202
- ID: 118120
Цитировать
Полный текст
Аннотация
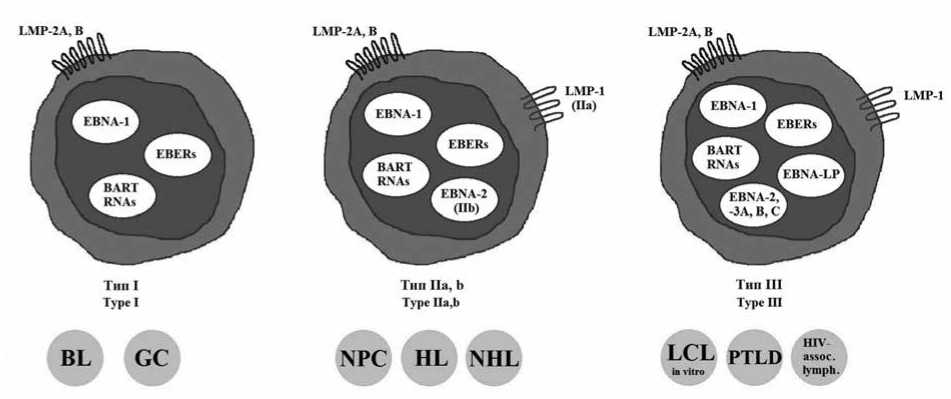
Вирус Эпштейна-Барр (ВЭБ) - один из наиболее распространённых в человеческой популяции, он способен на протяжении всей жизни персистировать в покоящихся В-клетках памяти, в Т-клетках (ВЭБ 2-го типа) и в некоторых недифференцированных эпителиальных клетках. У большинства людей персистенция ВЭБ не сопровождается значительными симптомами, но при частых активациях вируса возрастают риски тяжёлых сопутствующих заболеваний, включая хроническую активную ВЭБ-инфекцию, гемофагоцитарный лимфогистиоцитоз, рассеянный склероз, системную красную волчанку, карциному желудка и носоглотки, а также различные Т- и В-клеточные лимфомы. Большой интерес представляют молекулярные вирусные и клеточные процессы во время бессимптомной или малосимптомной персистенции ВЭБ. В этом обзоре рассматриваются поведение вирусной ДНК в заражённой клетке, формы её существования (линейная, циркулярная эписома, хромосомно-интегрированная форма), а также методы копирования генома ВЭБ. Рассмотрены два тесно связанных цикла вируса - литический и латентный. Литическая активация неблагоприятна для выживания конкретного вирусного генома в клетке, она запускается в результате дифференцировки латентно инфицированной клетки или появления стресс-сигналов из-за неблагоприятных условий внеклеточной среды. ВЭБ обладает большим количеством адаптивных механизмов для предотвращения литической реактивации и снижения враждебности иммунных клеток хозяина. Понимание молекулярных аспектов персистенции ВЭБ поможет в будущем разработать более эффективные, таргетные препараты для лечения как самой вирусной инфекции, так и сопутствующих заболеваний.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
С. А. Якушина
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: sofia.iakushina@gmail.com
ORCID iD: 0000-0003-0507-0174
Якушина Софья Александровна – младший научный сотрудник лаборатории хронических вирусных инфекций.
123098, Москва, ул. Гамалеи, д. 18
РоссияЛ. Б. Кистенева
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: lidia.kisteneva@gmail.com
ORCID iD: 0000-0001-7336-409X
Кистенева Лидия Борисовна – доктор медицинских наук, зав. лабораторией хронических вирусных инфекций.
123098, Москва, ул. Гамалеи, д. 18
РоссияСписок литературы
- Kieff E. Epstein-Barr virus and its replication. In: Fields B.N., Knipe D.M., Howley P.M., eds. Field’s virology. Volume 2. Philadelphia: Lippincott-Raven Publishers; 1996: 2343-96.
- Бошьян Р.Е. Инфекция, вызванная вирусом Эпштейна-Барр: эпидемиологические проявления и лабораторная диагностика: Автореф. дисс... канд. мед. наук. М.; 2009.
- Hutt-Fletcher LM. Epstein-Barr virus entry. J. Virol. 2007; 81(15): 7825-32. DOI: http://doi.org/10.1128/JVI.00445-07
- Fearon D.T., Carter R.H. The CD19/CR2/TAPA-1 complex of B lymphocytes: linking natural to acquired immunity. Annu. Rev. Immunol. 1995; 13: 127-49. DOI: http://doi.org/10.1146/annurev.iy.13.040195.001015
- Fingeroth J.D., Diamond M.E., Sage D.R., Hayman J., Yates J.L. CD21-Dependent infection of an epithelial cell line, 293, by Epstein-Barr virus. J. Virol. 1999; 73(3): 2115-25. DOI: http://doi.org/10.1128/JVI.73.3.2115-2125.1999
- Maruo S., Yang L., Takada K. Roles of Epstein-Barr virus glycoproteins gp350 and gp25 in the infection of human epithelial cells. J. Gen. Virol. 2001; 82(Pt. 10): 2373-83. DOI: http://doi.org/10.1099/0022-1317-82-10-2373
- Xiao J., Palefsky J.M., Herrera R., Berline J., Tugizov S.M. EBV BMRF-2 facilitates cell-to-cell spread of virus within polarized oral epithelial cells. Virology. 2009; 388(2): 335-43. DOI: http://doi.org/10.1016/j.virol.2009.03.030
- Rickinson A.B., Kieff E. Epstein-Barr virus. In: Fields B.N., Knipe D.M., Howley P.M., eds. Field’s virology. Volume 2. Philadelphia: Lippincott-Raven Publishers; 2007: 2655-700.
- Souza T.A., Stollar B.D., Sullivan J.L., Luzuriaga K., Thorley-Law-son D.A. Peripheral B cells latently infected with Epstein-Barr virus display molecular hallmarks of classical antigen-selected memory B cells. Proc. Natl. Acad. Sci. USA. 2005; 102(50): 18093-8. DOI: http://doi.org/10.1073/pnas.0509311102
- Thorley-Lawson D.A. EBV persistence - introducing the virus. Curr. Top. Microbiol. Immunol. 2015; 390(Pt. 1): 151-209. DOI: http://doi.org/10.1007/978-3-319-22822-8_8
- Hochberg D., Souza T., Catalina M., Sullivan J.L., Luzuriaga K., Thorley-Lawson D.A. Acute infection with Epstein-Barr Virus targets and overwhelms the peripheral memory B-cell compartment with resting, latently infected cells. J. Virol. 2004; 78(10): 5194-204. DOI: http://doi.org/rn.n28/JVI.78.rn.5194-5204.2004
- Coleman C.B., Wohlford E.M., Smith N.A., King C.A., Ritchie J.A., Baresel P.C., et al. Epstein-Barr virus type 2 latently infects T-cells, inducing an atypical activation characterized by expression of lymphotactic cytokines. J. Virol. 2015; 89(4): 2301-12. DOI: http://doi.org/10.1128/JVI.03001-14
- Якушина С.А., Кистенева Л.Б. Влияние персистенции вируса Эпштейна-Барр на развитие иммуноопосредованных соматических заболеваний. Российский вестник перинатологии и педиатрии. 2018; 63(1): 22-7. DOI: http://doi.org/10.21508/1027-4065-2018-63-1-22-27
- Loebel M., Eckey M., Sotzny F., Hahn E., Bauer S., Grabowski P., et al. Serological profiling of the EBV immune response in Chronic Fatigue Syndrome using a peptide microarray. PLoS One. 2017; 12(6): e0179124. DOI: http://doi.org/10.1371/journal.pone.0179124
- Handel A.E., Williamson A.J., Disanto G., Handunnetthi L., Giovannoni G., Ramagopalan S.V. An updated meta-analysis of risk of multiple sclerosis following infectious mononucleosis. PLoS One. 2010; 5(9): e12496. DOI: http://doi.org/10.1371/journal.pone.0012496
- Draborg A.H., Duus K., Houen G. Epstein-Barr virus in systemic autoimmune diseases. Clin. Dev. Immunol. 2013; 2013: 535738. DOI: http://doi.org/10.1155/2013/535738
- McGeoch D.J., Gatherer D. Lineage structures in the genome sequences of three Epstein-Barr virus strains. Virology. 2007; 359(1): 1-5. DOI: http://doi.org/10.1016/j.virol.2006.10.009
- Kanda T., Yajima M., Ikuta K. Epstein-Barr virus strain variation and cancer. CancerSci. 2019;110(4): 1132-9. DOI: http://doi.org/10.1111/cas.13954
- Zeng M.S., Li D.J., Liu Q.L., Song L.B., Li M.Z., Zhang R.H., et al. Genomic sequence analysis of Epstein-Barr virus strain GD1 from a nasopharyngeal carcinoma patient. J. Virol. 2005; 79(24): 15323-30. DOI: http://doi.org/10.1128/JVI.79.24.15323-15330.2005
- Tsai M.H., Lin X., Shumilov A., Bernhardt K., Feederle R., Poirey R., et al. The biological properties of different Epstein-Barr virus strains explain their association with various types of cancers. On-cotarget. 2016; 8(6): 10238-54. DOI: http://doi.org/10.18632/oncotarget.14380
- Lin Z., Wang X., Strong M.J., Concha M., Baddoo M., Xu G., et al. Whole-genome sequencing of the Akata and Mutu Epstein-Barr virus strains. J. Virol. 2013; 87(2): 1172-82. DOI: http://doi.org/10.1128/JVI.02517-12
- Palser A.L., Grayson N.E., White R.E., Corton C., Correia S., Ba Abdullah M.M., et al. Genome diversity of Epstein-Barr virus from multiple tumor types and normal infection. J. Virol. 2015; 89(10): 5222-37. DOI: http://doi.org/10.1128/JVI.03614-14
- Correia S., Bridges R., Wegner F., Venturini C., Palser A., Middeldorp J.M., et al. Sequence variation of Epstein-Barr Virus: viral types, geography, codon usage, and diseases. J. Virol. 2018; 92(22): e01132-18. DOI: http://doi.org/10.1128/JVI.01132-18
- Neves M., Marinho-Dias J., Ribeiro J., Sousa H. Epstein-Barr virus strains and variations: Geographic or disease-specific variants? J. Med. Virol. 2017; 89(3): 373-87. DOI: http://doi.org/10.1002/jmv.24633
- Adamson A.L., Darr D., Holley-Guthrie E., Johnson R.A., Mauser A., Swenson J., et al. Epstein-Barr virus immediate-early proteins BZLF1 and BRLF1 activate the ATF2 transcription factor by increasing the levels of phosphorylated p38 and c-Jun N-terminal kinases. J. Virol. 2000; 74(3): 1224-33. DOI: http://doi.org/10.1128/jvi.74.3.1224-1233.2000
- Abbott R.J., Quinn L.L., Leese A.M., Scholes H.M., Pachnio A., Rickinson A.B. CD8+ T cell responses to lytic EBV infection: late antigen specificities as subdominant components of the total response. J. Immunol. 2013; 191(11): 5398-409. DOI: http://doi. org/10.4049/jimmunol.1301629
- Kanegane H., Wakiguchi H., Kanegane C., Kurashige T., Tosato G. Viral interleukin-10 in chronic active Epstein-Barr virus infection. J. Infect. Dis. 1997; 176(1): 254-7. DOI: http://doi.org/10.1086/517260
- Kang M.S., Kieff E. Epstein-Barr virus latent genes. Exp. Mol. Med. 2015; 47(1): e131. DOI: http://doi.org/10.1038/emm.2014.84
- Niedobitek G., Agathanggelou A., Herbst H., Whitehead L., Wright D.H., Young L.S. Epstein-Barr virus (EBV) infection in infectious mononucleosis: virus latency, replication and phenotype of EBV-infected cells. J. Pathol. 1997; 182: 151-9. DOI: http://doi.org/10.1002/(SICI)1096-9896(199706)182:2<151::AID-PATH824>3.0.CO;2-3
- Niedobitek G., Kremmer E., Herbst H., Whitehead L., Dawson C.W., Niedobitek E., et al. Immunohistochemical detection of the Epstein-Barr virus-encoded latent membrane protein 2A in Hodgkin’s disease and infectious mononucleosis. Blood. 1997; 90(4): 1664-72.
- Gulley M.L., Raab-Traub N. Detection of Epstein-Barr virus in human tissues by molecular genetic techniques. Arch. Pathol. Lab. Med. 1993; 117(11): 1115-20.
- Arvin A., Campadelli-Fiume G., Mocarski E., Moore P.S., Roizman B., Whitley R., et al., eds. Human Herpesviruses: Biology, Therapy, and Immunoprophylaxis. Cambridge; 2007.
- Hurley E.A., Thorley-Lawson D.A. B cell activation and the establishment ofEpstein-Barr virus latency. J. Exp. Med. 1988; 168(6): 2059-75. DOI: http://doi.org/10.1084/jem.168.6.2059
- Yates J.L Epstein-Barr virus DNA replication. In: DePamphilis M. L., ed. DNA Replication in Eukaryotic Cells. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press; 1996: 751-74.
- Hammerschmidt W., Sugden B. Replication of Epstein-Barr viral DNA. Cold Spring Harb. Perspect. Biol. 2013; 5(1): a013029. DOI: http://doi.org/10.1101/cshperspect.a013029
- Hammerschmidt W., Sugden B. Identification and characterization of oriLyt, a lytic origin of DNA replication of Epstein-Barr virus. Cell. 1988; 55(3): 427-33. DOI: http://doi.org/10.1016/0092-8674(88)90028-1
- Neuhierl B., Delecluse H.J. The Epstein-Barr virus BMRF1 gene is essential for lytic virus replication. J. Virol. 2006; 80(10): 5078-81. DOI: http://doi.org/10.1128/JVI.80.10.5078-5081.2006
- Narita Y., Sugimoto A., Kawashima D., Watanabe T., Kanda T., Ki-mura H., et al. A herpesvirus specific motif of Epstein-Barr virus DNA polymerase is required for the efficient lytic genome synthesis. Sci. Rep. 2015; 5: 11767. DOI: http://doi.org/10.1038/srep11767
- Schildgen O., Graper S., Blumel J., Matz B. Genome replication and progeny virion production of herpes simplex virus type 1 mutants with temperature-sensitive lesions in the origin-binding protein. J. Virol. 2005; 79(11): 7273-8. DOI: http://doi.org/10.1128/JVI.79.11.7273-7278.2005
- Daikoku T., Kudoh A., Fujita M., Sugaya Y., Isomura H., Shirata N. , et al. Architecture of replication compartments formed during Epstein-Barr virus lytic replication. J. Virol. 2005; 79(6): 3409-18. DOI: http://doi.org/10.1128/JVI.79.63409-3418.2005
- Tsurumi T., Fujita M., Kudoh A. Latent and lytic Epstein-Barr virus replication strategies. Rev. Med. Virol. 2005; 15(1): 3-15. dOi: http://doi.org/10.1002/rmv.441
- Maul G.G. Nuclear domain 10, the site of DNA virus transcription and replication. Bioessays. 1998; 20(8): 660-7. DOI: http://doi.org/10.1002/(SICI)1521-1878(199808)20:8<660::AID-BIES9>3.0.CO;2-M
- Rivera-Molina Y.A., Martinez F.P., Tang Q. Nuclear domain 10 of the viral aspect. World J. Virol. 2013; 2(3): 110-22. DOI: http://doi.org/10.5501/wjv.v2.i3.110
- Amon W., White R.E., Farrell P.J. Epstein-Barr virus origin of lytic replication mediates association of replicating episomes with promyelocytic leukaemia protein nuclear bodies and replication compartments. J. Gen. Virol. 2006; 87(Pt. 5): 1133-7. DOI: http://doi.org/10.1099/vir.0.81589-0
- Sivachandran N., Wang X., Frappier L. Functions of the Epstein-Barr Virus EBNA1 Protein in Viral Reactivation and Lytic Infection. J. Virol. 2012; 86(11): 6146-58. DOI: http://doi.org/10.1128/JVI.00013-12
- Ling P.D., Peng R.S., Nakajima A., Yu J.H., Tan J., Moses S.M., et al. Mediation of Epstein-Barr virus EBNA-LP transcriptional coactivation by Sp100. EMBO J. 2005; 24: 3565-75. DOI: http://doi.org/10.1038/sj.emboj.7600820
- Tsai K., Thikmyanova N., Wojcechowskyj J.A., Delecluse H.J., Lieberman P.M. EBV tegument protein BNRF1 disrupts DAXX-ATRX to activate viral early gene transcription. PLoSPathog. 2011; 7(11): e1002376. DOI: http://doi.org/10.1371/journal.ppat.1002376
- Shaw J.E., Levinger L.F., Carter C.W. Nucleosomal structure of Epstein-Barr virus DNA in transformed cell lines. J. Virol. 1979; 29(2): 657-65. DOI: http://doi.org/10.1128/JVI.29.2.657-665.1979
- Morissette G., Flamand L. Herpesviruses and chromosomal integration. J. Virol. 2010; 84(23): 12100-9. DOI: http://doi.org/10.n28/JVI.01169-m
- Reisinger J., Rumpler S., Lion T., Ambros P.F. Visualization of ep-isomal and integrated Epstein-Barr virus DNA by fiber fluorescence in situ hybridization. Int. J. Cancer. 2006; 118(7): 1603-8. DOI: http://doi.org/10.1002/ijc.21498
- Nanbo A., Sugden A., Sugden B. The coupling of synthesis and partitioning of EBV’s plasmid replicon is revealed in live cells. EMBO J. 2007; 26(19): 4252-62. DOI: http://doi.org/10.1038/sj.emboj.7601853
- Humme S., Reisbach G., Feederle R., Delecluse H.J., Bousset K., Hammerschmidt W., et al. The EBV nuclear antigen 1 (EBNA1) enhances B cell immortalization several thousandfold. Proc. Natl. Acad. Sci. USA. 2003; 100(19): 10989-94. DOI: http://doi.org/10.1073/pnas.1832776100
- Bell P., Lieberman P.M., Maul G.G. Lytic but not latent replication of Epstein-Barr virus is associated with PML and induces sequential release of nuclear domain 10 proteins. J. Virol. 2000; 74(24): 11800-10. DOI: http://doi.org/10.1128/jvi.74.24.11800-11810.2000
- Yates J.L., Guan N. Epstein-Barr virus-derived plasmids replicate only once per cell cycle and are not amplified after entry into cells. J. Virol. 1991; 65(1): 483-8. DOI: http://doi.org/10.1128/JVI.65.1.483-488.1991
- Gahn T.A., Schildkraut C.L. The Epstein-Barr virus origin of plasmid replication, oriP, contains both the initiation and termination sites of DNA replication. Cell. 1989; 58(3): 527-35. DOI: http://doi.org/10.1016/0092-8674(89)90433-9
- Deng Z., Lezina L., Chen C.J., Shtivelband S., So W., Lieberman P.M. Telomeric proteins regulate episomal maintenance of Epstein-Barr virus origin of plasmid replication. Mol. Cell. 2002; 9(3): 493-503. DOI: http://doi.org/10.1016/s1097-2765(02)00476-8
- Rawlins D.R., Milman G., Hayward S.D., Hayward G.S. Sequence-specific DNA binding of the Epstein-Barr virus nuclear antigen (EBNA-1) to clustered sites in the plasmid maintenance region. Cell. 1985; 42(3): 859-68. DOI: http://doi.org/10.1016/0092-8674(85)90282-x
- Yates J.L., Camiolo S.M., Bashaw J.M. The minimal replicator of Epstein-Barr virus oriP. J. Virol. 2000; 74(10): 4512-22. DOI: http://doi.org/10.1128/jvi.74.10.4512-4522.2000
- Norio P., Schildkraut C.L. Plasticity of DNA replication initiation in Epstein-Barr virus episomes. PLoSBiology. 2004;2(6): e152. DOI: http://doi.org/10.1371/journal.pbio.0020152
- Norio P., Schildkraut C.L., Yates J.L. Initiation of DNA replication within oriP is dispensable for stable replication of the latent Ep-stein-barr virus chromosome after infection of established cell lines. J. Virol. 2000; 74(18): 8563-74. DOI: http://doi.org/10.1128/jvi.74.18.8563-8574.2000
- Wang C.Y., Sugden B. Identifying a property of origins of DNA synthesis required to support plasmids stably in human cells. Proc. Natl. Acad. Sci. USA. 2008; 105(28): 9639-44. DOI: http://doi.org/10.1073/pnas.0801378105
- Zhou J., Snyder A.R., Lieberman P.M. Epstein-Barr virus episome stability is coupled to a delay in replication timing. J. Virol. 2009; 83(5): 2154-62. DOI: http://doi.org/10.1128/JVI.02115-08
- Chang Y., Cheng S.D., Tsai C.H. Chromosomal integration of Ep-stein-Barr virus genomes in nasopharyngeal carcinoma cells. Head Neck. 2002; 24(2): 143-50. DOI: http://doi.org/10.1002/hed.10039
- Epstein M.A., Achong B.G., Barr Y.M., Zajac B., Henle G., Henle W. Morphological and virological investigations on cultured Burkitt tumor lymphoblasts (strain Raji). J. Natl. Cancer Inst. 1966; 37(4): 547-59.
- Cheung S.T., Huang D.P., Hui A.B., Lo K.W., Ko C.W., Tsang Y.S., et al. Nasopharyngeal carcinoma cell line (C666-1) consistently harbouring Epstein-Barr virus. Int. J. Cancer. 1999; 83(1): 121-6. DOI: http://doi.org/10.1002/(sici)1097-0215(19990924)83:1<121::aid-ijc21>3.0.co;2-f
- Delecluse H.J., Bartnizke S., Hammerschmidt W., Bullerdiek J., Bornkamm G.W. Episomal and integrated copies of Epstein-Barr virus coexist in Burkitt lymphoma cell lines. J. Virol. 1993; 67(3): 1292-9. DOI: http://doi.org/10.1128/JVI.67.3.1292-1299.1993
- Traylen C.M., Patel H.R., Fondaw W., Mahatme S., Williams J.F., Walker L.R., et al. Virus reactivation: a panoramic view in human infections. Future Virol. 2011; 6(4): 451-63. DOI: http://doi.org/10.2217/fvl.11.21
- Gao J., Luo X., Tang K., Li X., Li G. Epstein-Barr virus integrates frequently into chromosome 4q, 2q, 1q and 7q of Burkitt’s lymphoma cell line (Raji). J. Virol. Methods. 2006; 136(1-2): 193-9. DOI: http://doi.org/10.1016/jjviromet.2006.05.013
- Xiao K., Yu Z., Li X., Li X., Tang K., Tu C., et al. Genome-wide analysis of Epstein-Barr virus (EBV) integration and strain in C666-1 and Raji cells. J. Cancer. 2016; 7(2): 214-24. DOI: http://doi.org/10.7150/jca.13150
- Takakuwa T., Luo W.J., Ham M.F., Sakane-Ishikawa F., Wada N., Aozasa K. Integration of Epstein-Barr virus into chromosome 6q15 of Burkitt lymphoma cell line (Raji) induces loss of BACH2 expression. Am. J. Pathol. 2004; 164(3): 967-74. DOI: http://doi.org/10.1016/S0002-9440(10)63184-7
- Xu M., Zhang W.L., Zhu Q., Zhang S., Yao Y.Y., Xiang T., et al. Genome-wide profiling of Epstein-Barr virus integration by targeted sequencing in Epstein-Barr virus associated malignancies. Thera-nostics. 2019; 9(4): 1115-24. DOI: http://doi.org/10.7150/thno.29622
- Rose C., Green M., Webber S., Kingsley L., Day R., Watkins S., et al. Detection of Epstein-Barr virus genomes in peripheral blood B cells from solid-organ transplant recipients by fluorescence in situ hybridization. J. Clin. Microbiol. 2002; 40(7): 2533-44. DOI: http://doi.org/10.1128/JCM.40.7.2533-2544.2002
- Hall C.B., Caserta M.T., Schnabel K., Shelley L.M., Marino A.S., Carnahan J.A., et al. Chromosomal integration of human herpesvirus 6 is the major mode of congenital human herpesvirus 6 infection. Pediatrics. 2008; 122(3): 513-20. DOI: http://doi.org/10.1542/peds.2007-2838
Дополнительные файлы