Ancient variants of the Epstein–Barr virus (Herpesviridae, Lymphocryptovirus, HHV-4): hypotheses and facts
- Authors: Smirnova K.V.1, Senyuta N.B.2, Lubenskaya A.K.2, Dushenkina T.E.2, Gurtsevich V.E.2
-
Affiliations:
- PhD, Head of the Laboratory of Viral Carcinogenesis of the N.N. Blokhin NMRC of Oncology, Moscow, 115478, Russia
- Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
- Issue: Vol 65, No 2 (2020)
- Pages: 77-86
- Section: ORIGINAL RESEARCHES
- URL: https://ogarev-online.ru/0507-4088/article/view/118028
- DOI: https://doi.org/10.36233/0507-4088-2020-65-2-77-86
- ID: 118028
Cite item
Full Text
Abstract
Introduction. Molecular studies have shown that viruses appeared in the early stages of the evolution of life. For millions of years, viruses have evolved by changing old and acquiring new sequences in their RNA or DNA. It is assumed that most viruses have common ancestors. Such an ancestor, an ancient strain, probably existed for Epstein-Barr virus (EBV) as well.
Aim. To find out whether ancient strains of EBV persist in modern Russian ethnic groups today.
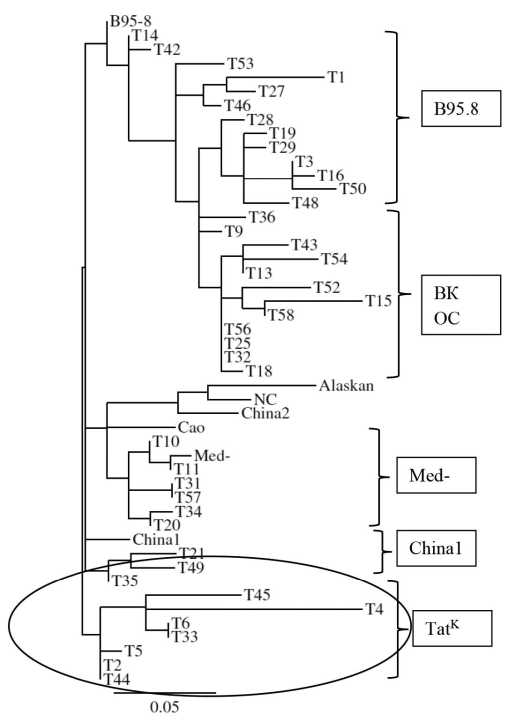
Material and methods. The object of the study was the EBV LMP1oncogene, which is most suitable for molecular genetic analysis. LMP1 was amplified from the oral cavity washings obtained from representatives of two ancient ethnic groups of Russia - Tatars and Slavs. The LMP1 amplicons were sequenced in both directions; their nucleotide sequences translated into amino acid (LMP1) were evaluated using the classification suggested by Edwards et al. 1999. To establish genetic relationships between LMP1 variants, a phylogenetic tree was constructed by the neighbor-joining method using the MEGA software package.
Results and discussion. Analysis of LMP1 sequences from washings of the Slavs oral cavity demonstrated the presence of LMP1 variants with varying degrees of transforming potential: B98.5/A, China1, Med-, and NC. The analysis of LMP1 sequences from washings of Tatar oral cavity also made it possible to identify oncoprotein variants such as B95.8/A, China1, Med-, as well as a group of variants out of classifications (LMP1-OK). An important finding was the identification of 7 variants of LMP1 from Tatars, designated as LMP1-TatK, that contained two unique deletions of 5 aa in codons 312-316 and 382-386, which were absent in the LMP1 variants from Slavs and from previously examined cancer patients and healthy individuals, as well as in sequences from open computer databases. The uniqueness of the LMP1-TatK variant is confirmed both by phylogenetic analysis of LMP1 sequences of Tatar origin and by the analysis of 11 aa repeats and 5 aa insertions in the C-terminal region of the oncoprotein. The morbidity and mortality rates from neoplasms, including EBV-associated pathologies, did not differ significantly between two studied ethnic groups infected with different EBV strains.
Conclusion. The data obtained allowed us to suggest that: 1) LMP1-TatK could be refered to an evolutionarily ancient EBV strain that persists among Tatars and; 2) LMP1-TatK belongs to the so-called “Volga” EBV virus strain, the common strain among the population of the Volga region. Extended studies of EBV isolates from residents of this region may probably shed the light on the origin of LMP1-TatK.
Full Text
##article.viewOnOriginalSite##About the authors
Kseniya V. Smirnova
PhD, Head of the Laboratory of Viral Carcinogenesis of the N.N. Blokhin NMRC of Oncology, Moscow, 115478, Russia
Author for correspondence.
Email: skv.lab@yandex.ru
ORCID iD: 0000-0001-6209-977X
Natalya B. Senyuta
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
ORCID iD: 0000-0001-8915-8274
Russian Federation
Alexandra K. Lubenskaya
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
ORCID iD: 0000-0003-3953-7449
Russian Federation
Tatyana E. Dushenkina
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
ORCID iD: 0000-0001-8279-514X
Russian Federation
Vladimir E. Gurtsevich
Research Institute of Carcinogenesis, N.N. Blokhin National Medical Research Center of Oncology
ORCID iD: 0000-0003-1840-4364
Russian Federation
References
- Koonin E.V., Dolja V.V., Krupovic M. Origins and evolution of viruses of eukaryotes: The ultimate modularity. Virology. 2015; 479-480: 2-25. DOI: http://doi.org/10.1016/j.virol.2015.02.039
- Ehlers B., Spiess K., Leendertz F., Peeters M., Boesch C., Gatherer D., McGeoch D.J. Lymphocryptovirus phylogeny and the origins of Epstein-Barr virus. J.Gen. Virol. 2010; 91: 630-642. doi: 10.1099/vir.0.017251-0
- Zhdanov V.M., Tikchonenko T.I. Viruses as a factor of evolution: exchange of genetic information in the biosphere. Adv. Virus Res. 1974; 19: 361-94. DOI: http://doi.org/10.1016/s0065-3527(08)60664-8
- Forterre P. Giant viruses: conflicts in revisiting the virus concept. Intervirology. 2010; 53(5): 362-78. DOI: http://doi.org/10.1159/000312921
- Krupovic M., Dolja V.V., Koonin E.V. Origin of viruses: primordial replicators recruiting capsids from hosts. Nat. Rev. Microbiol. 2019; 17(7): 449-58. DOI: http://doi.org/10.1038/s41579-019-0205-6
- Leppard K., Dimmock N., Easton A. Introduction to Modern Virology. Oxford: Blackwell Publishing Ltd; 2007.
- Mahy B.W. Strategies of virus persistence. Br. Med. Bull. 1985; 41(1): 50-5. DOI: http://doi.org/10.1093/oxfordjournals.bmb.a072024
- Mahy B.W. Human viral infections: an expanding frontier. Antiviral Res. 1997; 36(2): 75-80. DOI: http://doi.org/10.1016/s0166-3542(97)00042-9
- Walling D.M., Shebib N., Weaver S.C., Nichols C.M., Flaitz C.M., Webster-Cyriaque J. The molecular epidemiology and evolution of Epstein-Barr virus: sequence variation and genetic recombination in the latent membrane protein-1 gene. J. Infect. Dis. 1999; 179(4): 763-74. DOI: http://doi.org/10.1086/314672
- Itakura O., Yamada S., Narita M., Kikuta H. High prevalence of a 30-base pair deletion and single-base mutations within the carboxy terminal end of the LMP-1 oncogene of Epstein-Barr virus in the Japanese population. Oncogene. 1996; 13(7): 1549-53.
- Kingma D.W., Weiss W.B., Jaffe E.S., Kumar S., Frekko K., Raffeld M. Epstein-Barr virus latent membrane protein-1 oncogene deletions: correlations with malignancy in Epstein-Barr virus-associated lymphoproliferative disorders and malignant lymphomas. Blood. 1996; 88(1): 242-51.
- Falk K., Gratama J.W., Rowe M., Zou J.Z., Khanim F., Young L.S., et al. The role of repetitive DNA sequences in the size variation of Epstein-Barr virus (EBV) nuclear antigens, and the identification of different EBV isolates using RFLP and PCR analysis. J. Gen. Virol. 1995; 76(Pt. 4): 779-90. DOI: http://doi.org/10.1099/0022-1317-76-4-779
- Walling D.M., Raab-Traub N. Epstein-Barr virus intrastrain recombination in oral hairy leukoplakia. J. Virol. 1994; 68(12): 7909-17.
- Гурцевич В.Э., Яковлева Л.С., Щербак Л.Н., Гончарова Е.В., Смирнова К.В., Дидук С.В. и др. Сиквенсные варианты онкогена LMP1 у больных опухолями полости рта, ассоциированными и не ассоциированными с вирусом Эпштейна-Барр. Молекулярная биология. 2013; 47(6): 987. DOI: http://doi.org/10.7868/S0026898413050042
- Parra B., Slots J. Detection of human viruses in periodontal pockets using polymerase chain reaction. Oral. Microbiol. Immunol. 1996; 11(5): 289-93. DOI: http://doi.org/10.1111/j.1399-302x.1996.tb00183.x
- Hahn P., Novikova E., Scherback L., Janik C., Pavlish O., Arkhipov V., et al. The LMP1 gene isolated from Russian nasopharyngeal carcinoma has no 30-bp deletion. Int . J. Cancer. 2001; 91(6): 815-21. DOI: http://doi.org/10.1002/1097-0215(200002)9999:9999<::AIDIJC1122>3.0.CO;2-W
- Edwards R.H., Seillier-Moiseiwitsch F., Raab-Traub N. Signature amino acid changes in latent membrane protein 1 distinguish Epstein-Barr virus strains. Virology. 1999; 261(1): 79-95. DOI: http://doi.org/10.1006/viro.1999.9855
- Miller W.E., Edwards R.H., Walling D.M., Raab-Traub N. Sequence variation in the Epstein-Barr virus latent membrane protein 1. J. Gen. Virol. 1994; 75(Pt. 10): 2729-40. DOI: http://doi.org/10.1099/0022-1317-75-10-2729
- Каприн А.Д., Старинский В.В., Петрова Г.В. Состояние онкологической помощи населению России в 2018 году. М.; 2019.
- Senyuta N., Yakovleva L., Goncharova E., Scherback L., Diduk S., Smirnova K., et al. Epstein-Barr virus latent membrane protein 1 polymorphism in nasopharyngeal carcinoma and other oral cavity tumors in Russia. J. Med. Virol. 2014; 86(2): 290-300. DOI: http://doi.org/10.1002/jmv.23729
- Kanai K., Satoh Y., Saiki Y., Ohtani H., Sairenji T. Difference of Epstein-Barr virus isolates from Japanese patients and African Burkitt’s lymphoma cell lines based on the sequence of latent membrane protein 1. Virus Genes. 2007; 34(1): 55-61. DOI: http://doi.org/10.1007/s11262-006-0010-y
- Voevodin A.F., Hirsch I. Immunoprecipitation of Epstein-Barr virus (EBV)-specific proteins by prelymphomatous and normal baboon sera containing antibodies reactive with EBV early antigen. Acta Virol. 1985; 29(3): 242-6.
- Ehlers B., Spiess K., Leendertz F., Peeters M., Boesch C., Gatherer D., et al. Lymphocryptovirus phylogeny and the origins of EpsteinBarr virus. J. Gen. Virol. 2010; 91(Pt. 3): 630-42. DOI: http://doi.org/10.1099/vir.0.017251-0
- Ehlers B., Ochs A., Leendertz F., Goltz M., Boesch C., Mätz-Rensing K. Novel simian homologues of Epstein-Barr virus. J. Virol. 2003; 77(19): 10695-9. DOI: http://doi.org/10.1128/jvi.77.19.10695-10699.2003
Supplementary files