人工智能技术在实验室医学中的应用经验、有效性与应用场景:系统综述
- 作者: Vasilev Y.A.1, Nanova O.G.1, Vladzymyrskyy A.V.1, Goldberg A.S.2, Blokhin I.A.1, Reshetnikov R.V.1
-
隶属关系:
- Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies
- The Russian Medical Academy of Continuous Professional Education
- 期: 卷 6, 编号 2 (2025)
- 页面: 251-267
- 栏目: 系统评价
- URL: https://ogarev-online.ru/DD/article/view/310214
- DOI: https://doi.org/10.17816/DD635349
- EDN: https://elibrary.ru/BXDWFO
- ID: 310214
如何引用文章
全文:
详细
论证。随着实验室医学领域数据量的持续增长,该领域亟需实现常规流程的自动化与标准化,以减轻医务人员的工作负担,使其能够专注于更具专业性的任务。机器学习模型和人工神经网络能够识别图像并分析大规模数据,为其在实验室中承担常规任务的应用与整合提供了潜力。
目的。分析全球文献中人工智能在实验室医学中的应用情况,评估其在解决现有问题方面的能力,并识别限制人工智能融入实验室流程的潜在障碍。
方法。文献检索通过PubMed检索系统、实验室成品解决方案制造商官网以及其他综述文章的参考文献进行。此外,还使用Mendeley软件进行参考文献管理。时间范围为2019年至2024年。提取信息包括文献计量数据、研究领域、主要方法学特征、人工智能与医务人员的诊断效能指标、参与医务人员的数量及经验水平,以及其在实际应用中的验证结果。研究质量评估采用改良版QUADAS-CAD问卷工具。
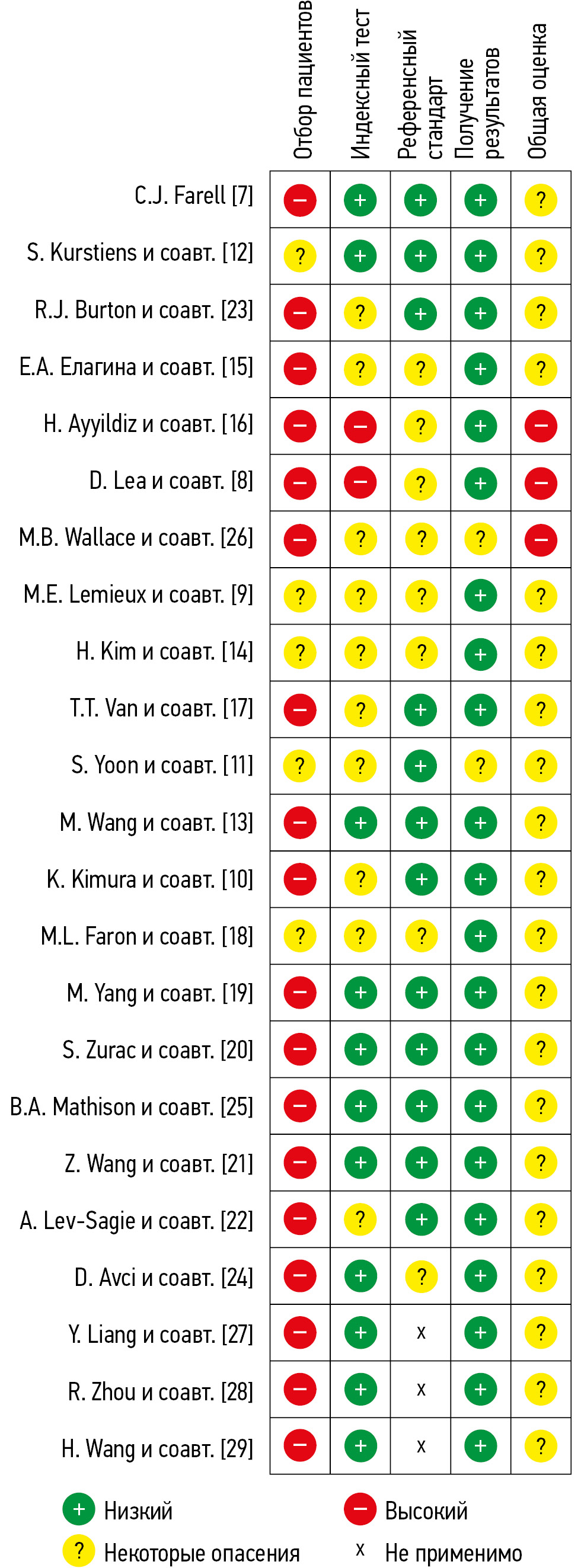
结果。本综述共纳入23篇文献,其中包括分别针对实验室分析前阶段(1项)、分析阶段(19项)和分析后阶段(3项)的研究。大多数研究集中于细胞学和微生物学领域,分别占48%和35%。人工智能在实验室各阶段任务的解决方面表现出较高的效能。此外,其诊断准确性可与医务人员水平相当,且决策速度显著更快。然而,所有研究均存在系统偏倚风险,主要原因包括样本分布不平衡、缺乏外部验证,以及对数据本身及其分析方法的描述不够详细。
结论。人工智能在诊断准确性和处理速度方面具有较高的潜力,因此被认为是推进实验室常规流程自动化和推广应用的有前景工具。然而,为实现这一目标,有必要:对人工智能研究方法进行标准化,以降低系统偏倚风险;为实验室建立参考标准,以确保结果的可重复性与可推广性;提高医务人员和患者对其工作机制的认知,以消除对人工智能的成见;制定可靠的个人数据保护机制,以保障人工智能应用过程中的数据安全。
作者简介
Yuriy A. Vasilev
Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies
Email: npcmr@zdrav.mos.ru
ORCID iD: 0000-0002-5283-5961
SPIN 代码: 4458-5608
MD, Cand. Sci. (Medicine)
俄罗斯联邦, 24 Petrovka st, bldg 1, Moscow, 127051Olga G. Nanova
Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies
编辑信件的主要联系方式.
Email: nanova@mail.ru
ORCID iD: 0000-0001-8886-3684
SPIN 代码: 6135-4872
Cand. Sci. (Biology)
俄罗斯联邦, 24 Petrovka st, bldg 1, Moscow, 127051Anton V. Vladzymyrskyy
Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies
Email: VladzimirskijAV@zdrav.mos.ru
ORCID iD: 0000-0002-2990-7736
SPIN 代码: 3602-7120
MD, Dr. Sci. (Medicine)
俄罗斯联邦, 24 Petrovka st, bldg 1, Moscow, 127051Arcadiy S. Goldberg
The Russian Medical Academy of Continuous Professional Education
Email: goldarcadiy@gmail.com
ORCID iD: 0000-0002-2787-4731
SPIN 代码: 8854-0469
MD, Cand. Sci. (Medicine)
俄罗斯联邦, MoscowIvan A. Blokhin
Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies
Email: BlokhinIA@zdrav.mos.ru
ORCID iD: 0000-0002-2681-9378
SPIN 代码: 3306-1387
MD, Cand. Sci. (Medicine)
俄罗斯联邦, 24 Petrovka st, bldg 1, Moscow, 127051Roman V. Reshetnikov
Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies
Email: ReshetnikovRV1@zdrav.mos.ru
ORCID iD: 0000-0002-9661-0254
SPIN 代码: 8592-0558
Cand. Sci. (Physics and Mathematics)
俄罗斯联邦, 24 Petrovka st, bldg 1, Moscow, 127051参考
- Bonert M, Zafar U, Maung R, et al. Pathologist workload, work distribution and significant absences or departures at a regional hospital laboratory. PLOS ONE. 2022;17(3):e0265905. doi: 10.1371/journal.pone.0265905 EDN: UFNVFE
- Hou H, Zhang R, Li J. Artificial intelligence in the clinical laboratory. Clinica Chimica Acta. 2024;559:119724. doi: 10.1016/j.cca.2024.119724 EDN: PBDERB
- Munari E, Scarpa A, Cima L, et al. Cutting-edge technology and automation in the pathology laboratory. Virchows Archiv. 2023;484(4):555–566. doi: 10.1007/s00428-023-03637-z EDN: OSGENI
- Vasilev YuA, Vladzymyrskyy AV, Omelyanskaya OV, et al. Guidelines for preparing a systematic review. Moscow: State Budget-Funded Health Care Institution of the City of Moscow “Research and Practical Clinical Center for Diagnostics and Telemedicine Technologies of Moscow Health Care Department”; 2023. 34 p. (In Russ.) EDN: XKXHDA
- Anjankar AP, Jha RK, Lambe S. Implementation of artificial intelligence in laboratory medicine. Journal of Datta Meghe Institute of Medical Sciences University. 2023;18(4):598–601. doi: 10.4103/jdmimsu.jdmimsu_486_22 EDN: VBNWUF
- Kodenko MR, Vasilev YuA, Vladzymyrskyy AV, et al. Diagnostic accuracy of ai for opportunistic screening of abdominal aortic aneurysm in ct: a systematic review and narrative synthesis. Diagnostics. 2022;12(12):3197. doi: 10.3390/diagnostics12123197 EDN: ERWYPX
- Farrell CJ. Identifying mislabelled samples: machine learning models exceed human performance. Annals of Clinical Biochemistry: International Journal of Laboratory Medicine. 2021;58(6):650–652. doi: 10.1177/00045632211032991 EDN: MQQLCW
- Lea D, Gudlaugsson EG, Skaland I, et al. Digital image analysis of the proliferation markers Ki67 and phosphohistone H3 in gastroenteropancreatic neuroendocrine neoplasms: accuracy of grading compared with routine manual hot spot evaluation of the Ki67 index. Applied Immunohistochemistry & Molecular Morphology. 2021;29(7):499–505. doi: 10.1097/pai.0000000000000934 EDN: XIKRGL
- Lemieux ME, Reveles XT, Rebeles J, et al. Detection of early-stage lung cancer in sputum using automated flow cytometry and machine learning. Respiratory Research. 2023;24(1):23. doi: 10.1186/s12931-023-02327-3 EDN: HSQBUA
- Kimura K, Tabe Y, Ai T, et al. A novel automated image analysis system using deep convolutional neural networks can assist to differentiate MDS and AA. Scientific Reports. 2019;9(1):1–9. doi: 10.1038/s41598-019-49942-z EDN: PXXHII
- Yoon S, Hur M, Park M, et al. Performance of digital morphology analyzer Vision Pro on white blood cell differentials. Clinical Chemistry and Laboratory Medicine (CCLM). 2021;59(6):1099–1106. doi: 10.1515/cclm-2020-1701 EDN: GVMONA
- Kurstjens S, de Bel T, van der Horst A, et al. Automated prediction of low ferritin concentrations using a machine learning algorithm. Clinical Chemistry and Laboratory Medicine (CCLM). 2022;60(12):1921–1928. doi: 10.1515/cclm-2021-1194 EDN: HDJWKG
- Wang M, Dong C, Gao Y, et al. A deep learning model for the automatic recognition of aplastic anemia, myelodysplastic syndromes, and acute myeloid leukemia based on bone marrow smear. Frontiers in Oncology. 2022;12: 844978. doi: 10.3389/fonc.2022.844978 EDN: BQFWSO
- Kim H, Lee GH, Yoon S, et al. Performance of digital morphology analyzer Medica EasyCell assistant. Clinical Chemistry and Laboratory Medicine (CCLM). 2023;61(10):1858–1866. doi: 10.1515/cclm-2023-0100 EDN: ZDXONI
- Elagina EA, Margun AA. Research of machine learning methods in the problem of identification of blood cells. Scientific and Technical Journal of Information Technologies, Mechanics and Optics. 2021;21(6):903–911. doi: 10.17586/2226-1494-2021-21-6-903-911 EDN: ZVQLEV
- Ayyıldız H, Arslan Tuncer S. Is it possible to determine antibiotic resistance of E. coli by analyzing laboratory data with machine learning? Turkish Journal of Biochemistry. 2021;46(6):623–630. doi: 10.1515/tjb-2021-0040 EDN: JTZHYJ
- Van TT, Mata K, Bard JD. Automated detection of Streptococcus pyogenes pharyngitis by use of Colorex Strep A CHROMagar and WASPLab artificial intelligence chromogenic detection module software. Journal of Clinical Microbiology. 2019;57(11):e00811-19. doi: 10.1128/JCM.00811-19
- Faron ML, Buchan BW, Relich RF, et al. Evaluation of the WASPLab segregation software to automatically analyze urine cultures using routine blood and MacConkey agars. Journal of Clinical Microbiology. 2020;58(4):e01683-19. doi: 10.1128/jcm.01683-19 EDN: UDENAP
- Yang M, Nurzynska K, Walts AE, Gertych A. A CNN-based active learning framework to identify mycobacteria in digitized Ziehl–Neelsen stained human tissues. Computerized Medical Imaging and Graphics. 2020;84:101752. doi: 10.1016/j.compmedimag.2020.101752 EDN: AYLPVY
- Zurac S, Mogodici C, Poncu T, et al. A new artificial intelligence-based method for identifying mycobacterium tuberculosis in Ziehl–Neelsen stain on tissue. Diagnostics. 2022;12(6):1484. doi: 10.3390/diagnostics12061484 EDN: IJUCYT
- Wang Z, Zhang L, Zhao M, et al. Deep neural networks offer morphologic classification and diagnosis of bacterial vaginosis. Journal of Clinical Microbiology. 2021;59(2):e02236-20. doi: 10.1128/JCM.02236-20 EDN: GBZITD
- Lev-Sagie A, Strauss D, Ben Chetrit A. Diagnostic performance of an automated microscopy and pH test for diagnosis of vaginitis. NPJ Digital Medicine. 2023;6(1):66. doi: 10.1038/s41746-023-00815-w EDN: SVUVPJ
- Burton RJ, Albur M, Eberl M, Cuff SM. Using artificial intelligence to reduce diagnostic workload without compromising detection of urinary tract infections. BMC Medical Informatics and Decision Making. 2019;19:171. doi: 10.1186/s12911-019-0878-9
- Avci D, Sert E, Dogantekin E, et al. A new super resolution Faster R-CNN model based detection and classification of urine sediments. Biocybernetics and Biomedical Engineering. 2023;43(1):58–68. doi: 10.1016/j.bbe.2022.12.001 EDN: HQRRRR
- Mathison BA, Kohan JL, Walker JF, et al. Detection of intestinal protozoa in trichrome-stained stool specimens by use of a deep convolutional neural network. Journal of Clinical Microbiology. 2020;58(6):e02053-19. doi: 10.1128/jcm.02053-19 EDN: GWHHRT
- Wallace MB, Sharma P, Bhandari P, et al. Impact of artificial intelligence on miss rate of colorectal neoplasia. Gastroenterology. 2022;163(1):295–304.e5. doi: 10.1053/j.gastro.2022.03.007 EDN: CVAOAF
- Liang Y, Wang Z, Huang D, et al. A study on quality control using delta data with machine learning technique. Heliyon. 2022;8(8):e09935. doi: 10.1016/j.heliyon.2022.e09935 EDN: XNSZKR
- Zhou R, Liang Y, Cheng H, et al. A multi-model fusion algorithm as a real-time quality control tool for small shift detection. Computers in Biology and Medicine. 2022;148:105866. doi: 10.1016/j.compbiomed.2022.105866 EDN: OBKKZC
- Wang H, Wang H, Zhang J, et al. Using machine learning to develop an autoverification system in a clinical biochemistry laboratory. Clinical Chemistry and Laboratory Medicine (CCLM). 2020;59(5):883–891. doi: 10.1515/cclm-2020-0716 EDN: SVNLZY
- Lippi G, Mattiuzzi C, Favaloro E. Artificial intelligence in the pre-analytical phase: state-of-the art and future perspectives. Journal of Medical Biochemistry. 2024;43(1):1–10. doi: 10.5937/jomb0-45936 EDN: PVAVYI
- Blatter TU, Witte H, Nakas CT, Leichtle AB. Big data in laboratory medicine-FAIR quality for AI? Diagnostics. 2022;12(8):1923. doi: 10.3390/diagnostics12081923 EDN: MCJCST
- Ghassemi M, Oakden-Rayner L, Beam AL. The false hope of current approaches to explainable artificial intelligence in health care. The Lancet Digital Health. 2021;3(11):e745–e750. doi: 10.1016/s2589-7500(21)00208-9 EDN: EHUNYG
- Paranjape K, Schinkel M, Hammer RD, et al. The value of artificial intelligence in laboratory medicine. American Journal of Clinical Pathology. 2020;155(6):823–831. doi: 10.1093/ajcp/aqaa170 EDN: KUADLL
- Ghosh K, Bellinger C, Corizzo R, et al. The class imbalance problem in deep learning. Machine Learning. 2022;113(7):4845–4901. doi: 10.1007/s10994-022-06268-8 EDN: AQXQUP
- Certuficate of state registration of a computer program No. 2023665713/ 19.07.2023. Byul. No. 7. Vasilev YuA, Vladzymyrskyy AV, Omelyanskaya OV, et al. Web platform for technological and clinical monitoring of the results of digital medical image analysis algorithms. Available from: https://elibrary.ru/download/elibrary_54200632_17081735.PDF (In Russ.) EDN: JIEPJK
- Zinchenko VV, Arzamasov KM, Kremneva EI, et al. Technological defects in software based on artificial intelligence. Digital Diagnostics. 2023;4(4):593–604. doi: 10.17816/DD501759 EDN: ORUFMM
- Sharova DE, Garbuk SV, Vasilyev YuA. Artificial intelligence systems in clinical medicine: the world’s first series of national standards. Standards and Quality. 2023;(1):46–51. doi: 10.35400/0038-9692-2023-1-304-22 EDN: SNMGQA
- Laddi A, Goyal S, Savlania A. Vein segmentation and visualization of upper and lower extremities using convolution neural network. Biomedical Engineering. Biomedizinische Technik. 2024;69(5):455–464. doi: 10.1515/bmt-2023-0331 EDN: PRAAZI
- Macaskill P, Takwoingi Y, Deeks JJ, Gatsonis C. Chapter 9: Understanding meta-analysis. In: Deeks JJ, Bossuyt PM, Leeflang MM, Takwoingi Y, editors. Cochrane handbook for systematic reviews of diagnostic test accuracy. version 2.0 (updated July 2023). Cochrane; 2023 [cited 2024 Aug 17]. Available from: https://training.cochrane.org/handbook-diagnostic-test-accuracy/current
- Pennestrì F, Banfi G. Artificial intelligence in laboratory medicine: fundamental ethical issues and normative key-points. Clinical Chemistry and Laboratory Medicine (CCLM). 2022;60(12):1867–1874. doi: 10.1515/cclm-2022-0096 EDN: ZOALXU
- Muehlematter UJ, Daniore P, Vokinger KN. Approval of artificial intelligence and machine learning-based medical devices in the USA and Europe (2015–20): a comparative analysis. The Lancet Digital Health. 2021;3(3):e195–e203. doi: 10.1016/s2589-7500(20)30292-2 EDN: UWEZGN
补充文件