Категория PI-RADS 3: возможности текстурного анализа в стратификации риска рака предстательной железы (систематический обзор)
- Авторы: Тян А.С.1, Кармазановский Г.Г.1,2, Карельская Н.А.1, Кондратьев Е.В.1, Грицкевич А.А.1, Калинин Д.В.1, Ковалев А.Д.1, Баева А.И.1
-
Учреждения:
- Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
- Российский национальный исследовательский медицинский университет имени Н.И. Пирогова
- Выпуск: Том 6, № 1 (2025)
- Страницы: 33-45
- Раздел: Систематические обзоры
- URL: https://ogarev-online.ru/DD/article/view/310050
- DOI: https://doi.org/10.17816/DD633500
- ID: 310050
Цитировать
Полный текст
Аннотация
Обоснование. Изменения предстательной железы категории PI RADS 3 — клиническая ситуация, требующая повышения точности диагностики и минимизации применения инвазивных методов. Изучение потенциальной ценности текстурного анализа изображений магнитно-резонансной томографии в стратификации риска рака предстательной железы является актуальной задачей современной медицинской диагностики.
Цель — систематизация и анализ современных данных о применении текстурного анализа для стратификации риска рака предстательной железы у пациентов с категорией PI-RADS 3, а также оценка его диагностической значимости в дифференциации клинически значимого и клинически незначимого рака предстательной железы.
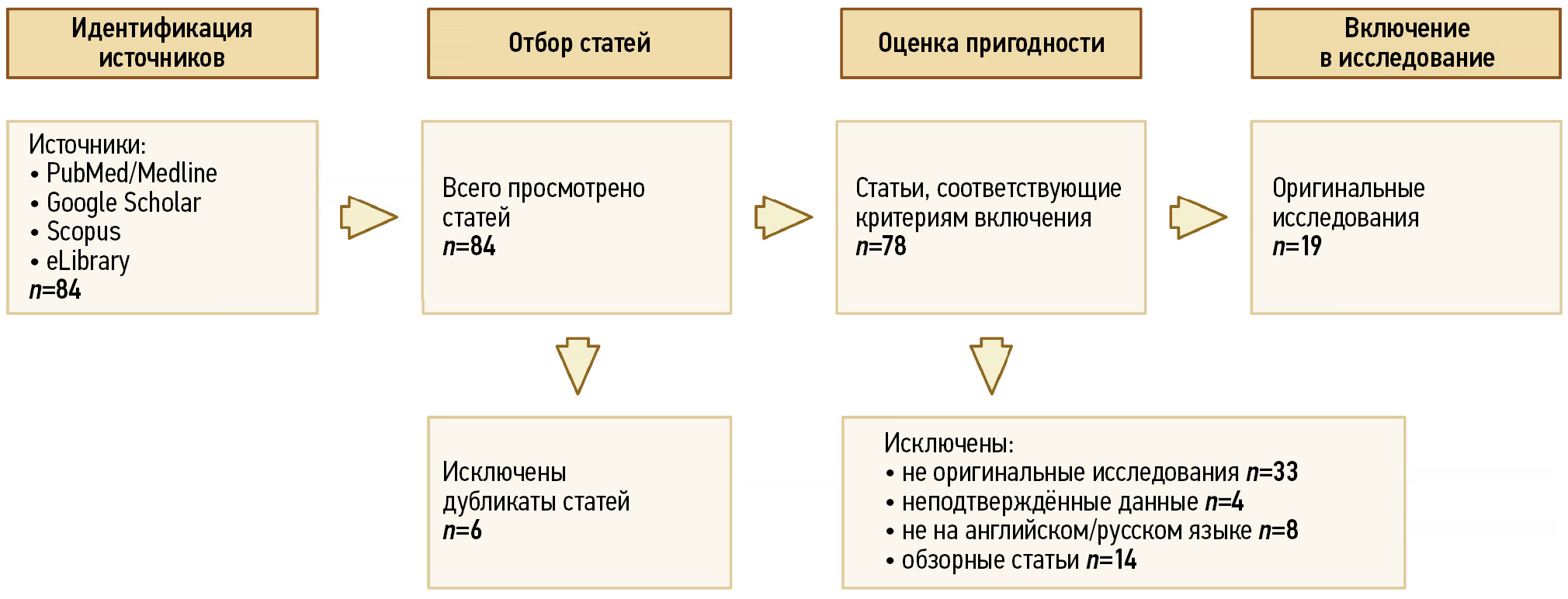
Материалы и методы. Отобраны и проанализированы статьи, опубликованные за последние 7 лет, найденные в базах данных реферативной и аналитической информации о научных исследованиях (Medline, Scopus) с использованием поисковых систем (PubMed, Google Scholar, eLibrary). Применяли ключевые слова, связанные с текстурным анализом и радиомикой в контексте диагностики и стратификации риска рака предстательной железы.
Результаты. Анализ отобранных публикаций показал, что применение машинного обучения и текстурного анализа значительно повышает точность диагностики рака предстательной железы. Эти методы позволяют более точно стратифицировать риски и определять реальную потребность в биопсии при раке предстательной железы, что потенциально ведёт к снижению количества ненужных инвазивных процедур.
Заключение. Текстурный анализ обладает возможностями для улучшения диагностической точности в случае изменений предстательной железы категории PI-RADS 3. Однако для его широкого клинического применения необходимо провести дополнительные исследования, направленные на стандартизацию методик, и мультицентровые клинические испытания.
Полный текст
Открыть статью на сайте журналаОб авторах
Александра Сергеевна Тян
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Автор, ответственный за переписку.
Email: tyan_a_s@staff.sechenov.ru
ORCID iD: 0009-0007-4193-7413
SPIN-код: 9110-9827
Россия, Москва
Григорий Григорьевич Кармазановский
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского; Российский национальный исследовательский медицинский университет имени Н.И. Пирогова
Email: karmazanovsky@ixv.ru
ORCID iD: 0000-0002-9357-0998
SPIN-код: 5964-2369
д-р мед. наук, профессор, академик РАН
Россия, Москва; МоскваНаталья Александровна Карельская
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Email: karelskaya.n@yandex.ru
ORCID iD: 0000-0001-8723-8916
SPIN-код: 9921-1430
канд. мед. наук
Россия, МоскваЕвгений Валерьевич Кондратьев
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Email: kondratev@ixv.ru
ORCID iD: 0000-0001-7070-3391
SPIN-код: 2702-6526
канд. мед. наук
Россия, МоскваАлександр Анатольевич Грицкевич
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Email: grekaa@mail.ru
ORCID iD: 0000-0002-5160-925X
SPIN-код: 2128-7536
д-р мед. наук, профессор
Россия, МоскваДмитрий Валерьевич Калинин
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Email: dmitry.v.kalinin@gmail.com
ORCID iD: 0000-0001-6247-9481
SPIN-код: 5563-5376
канд. мед. наук
Россия, МоскваАлександр Дмитриевич Ковалев
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Email: aledmikov@yandex.ru
ORCID iD: 0009-0001-9944-0473
Россия, Москва
Анастасия Игоревна Баева
Национальный медицинский исследовательский центр хирургии имени А.В. Вишневского
Email: nastya.baeva.2016@mail.ru
ORCID iD: 0000-0003-3747-7411
Россия, Москва
Список литературы
- Westphalen AC, McCulloch CE, Anaokar JM, et al. Variability of the Positive Predictive Value of PI-RADS for Prostate MRI across 26 Centers: Experience of the Society of Abdominal Radiology Prostate Cancer Disease-focused Panel. Radiology. 2020;296(1):76–84. doi: 10.1148/radiol.2020190646 EDN: FJNEKA
- Wadera A, Alabousi M, Pozdnyakov A, et al. Impact of PI-RADS Category 3 lesions on the diagnostic accuracy of MRI for detecting prostate cancer and the prevalence of prostate cancer within each PI-RADS category: a systematic review and meta-analysis. The British Journal of Radiology. 2020;94(1118):20191050. doi: 10.1259/bjr.20191050
- Satei AM, Kaur M, MacLean J, Hakim B. Review of clinically significant cancer in lesions labeled PI-RADS 3 on MRI using PI-RADS Version 2.1. Applied Radiology. 2023;(1 Suppl.):13–19.
- Barkovich EJ, Shankar PR, Westphalen AC. A systematic review of the existing prostate imaging reporting and data system version 2 (PI-RADSv2) literature and subset meta-analysis of PI-RADSv2 categories stratified by Gleason Scores. American Journal of Roentgenology. 2019;212(4):847–854. doi: 10.2214/AJR.18.20571
- Stanzione A, Gambardella M, Cuocolo R, et al. Prostate MRI radiomics: a systematic review and radiomic quality score assessment. European Journal of Radiology. 2020;129:109095. doi: 10.1016/j.ejrad.2020.109095 EDN: GUEKIR
- Bao J, Qiao X, Song Y, et al. Prediction of clinically significant prostate cancer using radiomics models in real-world clinical practice: a retrospective multicenter study. Insights into Imaging. 2024;15(1):68. doi: 10.1186/s13244-024-01631-w EDN: RFQGMA
- Gelezhe PB, Blokhin IA, Semenov SS, Caruso D. Magnetic resonance imaging radiomics in prostate cancer radiology: what is currently known? Digital Diagnostics. 2021;2(4):441–452. doi: 10.17816/DD70170 EDN: FFFGWI
- Min X, Li M, Dong D, et al. Multi-parametric MRI-based radiomics signature for discriminating between clinically significant and insignificant prostate cancer: cross-validation of a machine learning method. European Journal of Radiology. 2019;115:16–21. doi: 10.1016/j.ejrad.2019.03.010
- Woźnicki P, Westhoff N, Huber T, et al. Multiparametric MRI for prostate cancer characterization: combined use of radiomics model with PI-RADS and clinical parameters. Cancers (Basel). 2020;12(7):1767. doi: 10.3390/cancers12071767 EDN: DMCTFH
- Kwon D, Reis IM, Breto AL, et al. Classification of suspicious lesions on prostate multiparametric MRI using machine learning. Journal of Medical Imaging. 2018;5(3):034502. doi: 10.1117/1.JMI.5.3.034502
- He M, Cao Y, Chi C, et al. Research progress on deep learning in magnetic resonance imaging–based diagnosis and treatment of prostate cancer: a review on the current status and perspectives. Frontiers in Oncology. 2023;13:1189370. doi: 10.3389/fonc.2023.1189370 EDN: FEZIDE
- Chen Z, Li Z, Dou R, et al. Personalized optimization of systematic prostate biopsy core number based on mpMRI radiomics features: a large–sample retrospective analysis. BMC Cancer. 2025;25(1):116. doi: 10.1186/s12885-024-13391-3
- Hermie I, Van Besien J, De Visschere P, et al. Which clinical and radiological characteristics can predict clinically significant prostate cancer in PI-RADS 3 lesions? A retrospective study in a high-volume academic center. European Journal of Radiology. 2019;114:92–98. doi: 10.1016/j.ejrad.2019.02.031
- Epstein JI, Egevad L, Amin MB, et al. The 2014 International Society of Urological Pathology (ISUP) consensus conference on gleason grading of prostatic carcinoma. American Journal of Surgical Pathology. 2016;40(2):244–252. doi: 10.1097/PAS.0000000000000530
- Gulin GA, Zyryanov AV, Rubtsova NA, et al. Multiparametric magnetic resonance imaging and combined prostate biopsy: opportunities, advantages and pitfalls. Medical Visualization. 2021;25(2):138–152. doi: 10.24835/1607-0763-1023 EDN: MSXQIH
- Jin P, Shen J, Yang L, et al. Machine learning-based radiomics model to predict benign and malignant PI-RADS v2.1 category 3 lesions: a retrospective multi-center study. BMC Medical Imaging. 2023;23(1):47. doi: 10.1186/s12880-023-01002-9 EDN: QFWKOM
- Sonmez G, Tombul ST, Demirtas T, Demirtas A. Clinical factors for predicting malignancy in patients with PSA <10 ng/mL and PI-RADS 3 lesions. Asia-Pacific Journal of Clinical Oncology. 2020;17(2):e94–e99. doi: 10.1111/ajco.13347 EDN: ZVXTXF
- Vasilev AV, Mishchenko AV, Kadyrleev RA, et al. Cognitive mpMRI/TRUS biopsy of the prostate with using strain elastography. Medical Visualization. 2019;23(2):100–108. doi: 10.24835/1607-0763-2019-2-100-108 EDN: UPQEEH
- Fang AM, Shumaker LA, Martin KD, et al. Multi-institutional analysis of clinical and imaging risk factors for detecting clinically significant prostate cancer in men with PI-RADS 3 lesions. Cancer. 2022;128(18):3287–3296. doi: 10.1002/cncr.34355 EDN: HHUNTK
- Park KJ, Choi SH, Lee JS, et al. Risk stratification of prostate cancer according to PI-RADS® version 2 categories: meta-analysis for prospective studies. The Journal of Urology. 2020;204(6):1141–1149. doi: 10.1097/ju.0000000000001306 EDN: ASHPIN
- Önder Ö, Ayva M, Yaraşır Y, et al. Long-term follow-up results of multiparametric prostate MRI and the prognostic value of PI-RADS: a single-center retrospective cohort study. Diagnostic and Interventional Radiology. 2024;30(3):139–151. doi: 10.4274/dir.2023.232414
- Gromov AI, Kapustin VV. Usage of PI-RADS v2.1 system for prostate MRI: a practical approach. Medical Visualization. 2019;23(3):107–125. doi: 10.24835/1607-0763-2019-3-107-125 EDN: AHBXBT
- Gupta RT, Mehta KA, Turkbey B, Verma S. PI-RADS: Past, present, and future. Journal of Magnetic Resonance Imaging. 2020;52(1):33–53. doi: 10.1002/jmri.26896
- Washino S, Okochi T, Saito K, et al. Combination of prostate imaging reporting and data system (PI-RADS) score and prostate-specific antigen (PSA) density predicts biopsy outcome in prostate biopsy naïve patients. BJU International. 2017;119(2):225–233. doi: 10.1111/bju.13465
- Giambelluca D, Cannella R, Vernuccio F, et al. PI-RADS 3 lesions: role of prostate MRI texture analysis in the identification of prostate cancer. Current Problems in Diagnostic Radiology. 2021;50(2):175–185. doi: 10.1067/j.cpradiol.2019.10.009 EDN: KZLCMA
- Kаrmаzаnovsky GG, Shantarevich MY, Stashkiv VI, Revishvili AS. Reproducibility of CT and MRI texture features of hepatocellular carcinoma. Medical Visualization. 2023;27(3):84–93. doi: 10.24835/1607-0763-1372 EDN: BVBFNX
- Ferro M, de Cobelli O, Musi G, et al. Radiomics in prostate cancer: an up-to-date review. Therapeutic Advances in Urology. 2022;14:17562872221109020. doi: 10.1177/17562872221109020
- Fischer S, Tahoun M, Klaan B, et al. A Radiogenomic approach for decoding molecular mechanisms underlying tumor progression in prostate cancer. Cancers. 2019;11(9):1293. doi: 10.3390/cancers11091293
- Hectors SJ, Chen C, Chen J, et al. Magnetic resonance imaging radiomics-based machine learning prediction of clinically significant prostate cancer in equivocal PI-RADS 3 lesions. Journal of Magnetic Resonance Imaging. 2021;54(5):1466–1473. doi: 10.1002/jmri.27692 EDN: SGBOWM
- Magoulianitis V, Yang J, Yang Y, et al. PCa-RadHop: a transparent and lightweight feed-forward method for clinically significant prostate cancer segmentation. Computerized Medical Imaging and Graphics. 2024;116:102408. doi: 10.1016/j.compmedimag.2024.102408 EDN: XEOIXK
- Penzias G, Singanamalli A, Elliott R, et al. Identifying the morphologic basis for radiomic features in distinguishing different Gleason grades of prostate cancer on MRI: preliminary findings. PLOS ONE. 2018;13(8):e0200730. doi: 10.1371/journal.pone.0200730
- Qi Y, Zhang S, Wei J, et al. Multiparametric MRI-Based radiomics for prostate cancer screening with PSA in 4–10 ng/mL to reduce unnecessary biopsies. Journal of Magnetic Resonance Imaging. 2019;51(6):1890–1899. doi: 10.1002/jmri.27008 EDN: EINUYX
- Gong L, Xu M, Fang M, et al. Noninvasive prediction of high-grade prostate cancer via biparametric MRI radiomics. Journal of Magnetic Resonance Imaging. 2020;52(4):1102–1109. doi: 10.1002/jmri.27132 EDN: EMVNVC
- Wang Y, Liu W, Chen Z, et al. A noninvasive method for predicting clinically significant prostate cancer using magnetic resonance imaging combined with PRKY promoter methylation level: a machine learning study. BMC Medical Imaging. 2024;24(1):60. doi: 10.1186/s12880-024-01236-1 EDN: RASMDD
- Corsi A, De Bernardi E, Bonaffini PA, et al. Radiomics in PI-RADS 3 multiparametric MRI for prostate cancer identification: literature models re-Implementation and proposal of a clinical–radiological model. Journal of Clinical Medicine. 2022;11(21):6304. doi: 10.3390/jcm11216304 EDN: GGECVG
- Brancato V, Aiello M, Basso L, et al. Evaluation of a multiparametric MRI radiomic-based approach for stratification of equivocal PI-RADS 3 and upgraded PI-RADS 4 prostatic lesions. Scientific Reports. 2021;11(1):643. doi: 10.1038/s41598-020-80749-5 EDN: HAJXQY
- Hou Y, Bao ML, Wu CJ, et al. A radiomics machine learning-based redefining score robustly identifies clinically significant prostate cancer in equivocal PI-RADS score 3 lesions. Abdominal Radiology. 2020;45(12):4223–4234. doi: 10.1007/s00261-020-02678-1 EDN: JHWPET
- Zhang Y, Chen W, Yue X, et al. Development of a novel, multi-parametric, MRI-based radiomic nomogram for differentiating between clinically significant and insignificant prostate cancer. Frontiers in Oncology. 2020;10(FEB):888. doi: 10.3389/fonc.2020.00888 EDN: QTDXSC
- Gresser E, Schachtner B, Stüber AT, et al. Performance variability of radiomics machine learning models for the detection of clinically significant prostate cancer in heterogeneous MRI datasets. Quantitative Imaging in Medicine and Surgery. 2022;12(11):4990–5003. doi: 10.21037/qims-22-265 EDN: JIFGJJ
- Krauss W, Frey J, Heydorn Lagerlöf J, et al. Radiomics from multisite MRI and clinical data to predict clinically significant prostate cancer. Acta Radiologica. 2023;65(3):307–317. doi: 10.1177/02841851231216555 EDN: VVMHZC
- Huang EP, O’Connor JPB, McShane LM, et al. Criteria for the translation of radiomics into clinically useful tests. Nature Reviews Clinical Oncology. 2022;20(2):69–82. doi: 10.1038/s41571-022-00707-0 EDN: XNGIOB
Дополнительные файлы