Source of the COVID-19 pandemic: ecology and genetics of coronaviruses (Betacoronavirus: Coronaviridae) SARS-CoV, SARS-CoV-2 (subgenus Sarbecovirus), and MERS-CoV (subgenus Merbecovirus)
- 作者: Lvov D.K.1, Alkhovsky S.V.2
-
隶属关系:
- Doctor of Medical Sciences, Professor, Academician of RAS, Head of the Department of Ecology of Viruses, Head of Ecology and Epidemiology of Influenza Center, D.I. Ivanovsky Institute of Virology of National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya, Moscow, 123098, Russia
- D.I. Ivanovsky Institute of Virology of National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya
- 期: 卷 65, 编号 2 (2020)
- 页面: 62-70
- 栏目: REVIEWS
- URL: https://ogarev-online.ru/0507-4088/article/view/118020
- DOI: https://doi.org/10.36233/0507-4088-2020-65-2-62-70
- ID: 118020
如何引用文章
全文:
详细
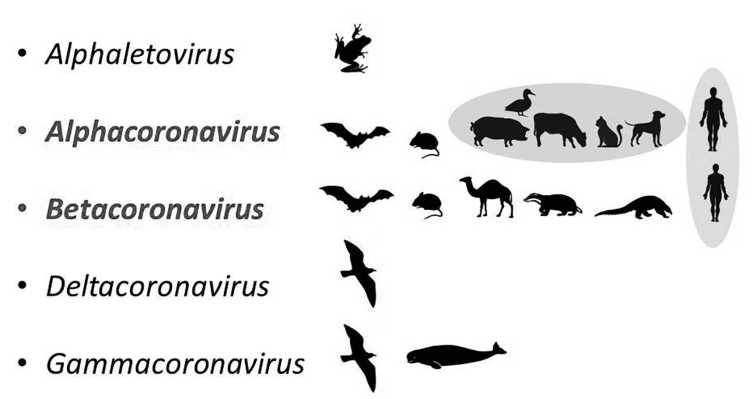
Since the early 2000s, three novel zooanthroponous coronaviruses (Betacoronavirus) have emerged. The first outbreak of infection (SARS) caused by SARS-CoV virus occurred in the fall of 2002 in China (Guangdong Province). A second outbreak (MERS) associated with the new MERS-CoV virus appeared in Saudi Arabia in autumn 2012. The third epidemic, which turned into a COVID-19 pandemic caused by SARS-CoV-2 virus, emerged in China (Hubei Province) in the autumn 2019. This review focuses on ecological and genetic aspects that lead to the emergence of new human zoanthroponous coronaviruses. The main mechanism of adaptation of zoonotic betacoronaviruses to humans is to changes in the receptor-binding domain of surface protein (S), as a result of which it gains the ability to bind human cellular receptors of epithelial cells in respiratory and gastrointestinal tract. This process is caused by the high genetic diversity and variability combined with frequent recombination, during virus circulation in their natural reservoir - bats (Microchiroptera, Chiroptera). Appearance of SARS-CoV, SARS-CoV-2 (subgenus Sarbecovirus), and MERS (subgenus Merbecovirus) viruses is a result of evolutionary events occurring in bat populations with further transfer of viruses to the human directly or through the intermediate vertebrate hosts, ecologically connected with bats.
This review is based on the report at the meeting «Coronavirus - a global challenge to science» of the Scientific Council «Life Science» of the Russian Academy of Science: Lvov D.K., Alkhovsky S.V., Burtseva E.I. COVID-19 pandemic sources: origin, biology and genetics of coronaviruses of SARS-CoV, SARS-CoV-2, MERS-CoV (Conference hall of Presidium of RAS, 14 Leninsky Prospect, Moscow, Russia. April 16, 2020)
作者简介
Dmitry Lvov
Doctor of Medical Sciences, Professor, Academician of RAS, Head of the Department of Ecology of Viruses, Head of Ecology and Epidemiology of Influenza Center, D.I. Ivanovsky Institute of Virology of National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya, Moscow, 123098, Russia
编辑信件的主要联系方式.
Email: dk_lvov@mail.ru
ORCID iD: 0000-0001-8176-6582
Sergey Alkhovsky
D.I. Ivanovsky Institute of Virology of National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya
Email: noemail@neicon.ru
ORCID iD: 0000-0001-6913-5841
俄罗斯联邦
参考
- de Groot R.J., Baker S.C., Baric R., Enjuanes L., Gorbalenya A.E., Holmes K.V., et al. Family Coronaviridae. In: King A.M., Adams M.J., Carstens E.B., Lefkowitz E.J., eds. Virus Taxonomy: Classification and Nomenclature of Viruses. Ninth Report of the International Committee on Taxonomy of Viruses. London: Elsevier; 2012: 806-28.
- Львов Д.К., Альховский С.В., Колобухина Л.В., Бурцева Е.И. Этиология эпидемической вспышки COVID-19 в г. Ухань (провинция Хубэй, Китайская Народная Республика), ассоциированной с вирусом 2019-nCoV (Nidovirales, Coronaviridae, Coronavirinae, Betacoronavirus, подрод Sarbecovirus): уроки эпидемии SARS-CoV. Вопросы вирусологии. 2020; 65(1): 6-16. DOI: http://doi.org/10.36233/0507-4088-2020-65-1-6-15
- Hamre D., Procknow J.J. A new virus isolated from the human respiratory tract. Proc. Soc. Exp. Biol. Med. 1966; 121(1): 190-3. DOI: http://doi.org/10.3181/00379727-121-30734
- McIntosh K., Dees J.H., Becker W.B., Kapikian A.Z., Chanock R.M. Recovery in tracheal organ cultures of novel viruses from patients with respiratory disease. Proc. Natl. Acad. Sci. USA. 1967; 57(4): 933-40. DOI: https://doi.org/10.1073/pnas.57.4.933
- Drosten C., Gunther S., Preiser W., van der Werf S., Brodt H.R., Becker S., et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N. Engl. J. Med. 2003; 348(20): 1967-76. DOI: http://doi.org/10.1056/NEJMoa030747
- Zaki A.M., van Boheemen S., Bestebroer T.M., Osterhaus A.D., Fouchier R.A., et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N. Engl. J. Med. 2012; 367(19): 1814-20. DOI: https://doi.org/10.1056/NEJMoa1211721
- Zhu N., Zhang D., Wang W., Li X., Yang B., Song J., et al. A novel coronavirus from patients with pneumonia in China, 2019. N. Engl. J. Med. 2020; 382(8): 727-33. DOI: http://doi.org/10.1056/NEJMoa2001017
- Zhou P., Yang X.L., Wang X.G., Hu B., Zhang L., Zhang W., et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020; 579(7798): 270-3. DOI: https://doi.org/10.1038/s41586-020-2012-7
- Lu R., Zhao X., Li J., Niu P., Yang B., Wu H., et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020; 395(10224): 565-74. DOI: https://doi.org/10.1016/S0140-6736(20)30251-8
- Lau S.K., Woo P.C., Li K.S., Huang Y., Tsoi H.W., Wong B.H., et al. Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. Proc. Natl. Acad. Sci. USA. 2005; 102(39): 14040-5. DOI: http://doi.org/10.1073/pnas.0506735102
- Li W., Shi Z., Yu M., Ren W., Smith C., Epstein J.H., et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005; 310(5748): 676-9. DOI: http://doi.org/10.1126/science.1118391
- Fan Y., Zhao K., Shi Z.L., Zhou P. Bat coronaviruses in China. Viruses. 2019; 11(3): pii E210. DOI: http://doi.org/10.3390/v11030210
- Rihtaric D., Hostnik P., Steyer A., Grom J., Toplak I. Identification of SARS-like coronaviruses in horseshoe bats (Rhinolophus hipposideros) in Slovenia. Arch. Virol. 2010; 155(4): 507-14. DOI: https://doi.org/10.1007/s00705-010-0612-5
- Ar Gouilh M., Puechmaille S.J., Diancourt L., Vandenbogaert M., Serra-Cobo J., Lopez Roïg M., et al. SARS-CoV related Betacoronavirus and diverse Alphacoronavirus members found in western old-world. Virology. 2018; 517: 88-97. DOI: http://doi.org/10.1016/j.virol.2018.01.014
- Balboni A., Palladini A., Bogliani G., Battilani M. Detection of a virus related to betacoronaviruses in Italian greater horseshoe bats. Epidemiol. Infect. 2011; 139(2): 216-9. DOI: http://doi.org/10.1017/S0950268810001147
- Drexler J.F., Gloza-Rausch F., Glende J., Corman V.M., Muth D., Goettsche M., et al. Genomic characterization of severe acute respiratory syndrome-related coronavirus in European bats and classification of coronaviruses based on partial RNA-dependent RNA polymerase gene sequences. J. Virol. 2010; 84(21): 11336-49. DOI: https://doi.org/10.1128/jvi.00650-10
- Donaldson E.F., Haskew A.N., Gates J.E., Huynh J., Moore C.J., Frieman M.B. Metagenomic analysis of the viromes of three North American bat species: viral diversity among different bat species that share a common habitat. J. Virol. 2010; 84(24): 13004-18. DOI: http://doi.org/10.1128/JVI.01255-10
- Wang L.F., Shi Z., Zhang S., Field H., Daszak P., Eaton B.T. Review of bats and SARS. Emerg. Infect. Dis. 2006; 12(12): 1834-40. DOI: http://doi.org/10.3201/eid1212.060401
- Tong S., Conrardy C., Ruone S., Kuzmin I.V., Guo X., Tao Y., et al. Detection of novel SARS-like and other coronaviruses in bats from Kenya. Emerg. Infect. Dis. 2009; 15(3): 482-5. DOI: http://doi.org/10.3201/eid1503.081013
- Drexler J.F., Corman V.M., Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral Res. 2014; 101: 45-56. DOI: http://doi.org/10.1016/J.ANTIVIRAL.2013.10.013
- Haagmans B.L., Al Dhahiry S.H.S., Reusken C.B.E.M., Raj V.S., Galiano M., Myers R., et al. Middle East respiratory syndrome coronavirus in dromedary camels: An outbreak investigation. Lancet Infect. Dis. 2014; 14(2): 140-5. DOI: https://doi.org/10.1016/S1473-3099(13)70690-X
- Sabir J.S., Lam T.T., Ahmed M.M., Li L., Shen Y., Abo-Aba S.E., et al. Co-circulation of three camel coronavirus species and recombination of MERS-CoVs in Saudi Arabia. Science. 2016; 351(6268): 81-4. DOI: https://doi.org/10.1126/science.aac8608
- Raj V.S., Farag E.A., Reusken C.B., Lamers M.M., Pas S.D., Voermans J., et al. Isolation of MERS coronavirus from dromedary camel, Qatar, 2014. Emerg. Infect. Dis. 2014; 20(8): 1339-42. DOI: http://doi.org/10.3201/eid2008.140663
- Paden C.R., Yusof M.F.B.M., Al Hammadi Z.M., Queen K., Tao Y., Eltahir Y.M., et al. Zoonotic origin and transmission of Middle East respiratory syndrome coronavirus in the UAE. Zoonoses Public Health. 2018; 65(3): 322-33. DOI: http://doi.org/10.1111/zph.12435
- Müller M.A., Corman V.M., Jores J., Meyer B., Younan M., Liljander A., et al. MERS coronavirus neutralizing antibodies in camels, eastern Africa, 1983-1997. Emerg. Infect. Dis. 2014; 20(12): 2093-5. DOI: https://doi.org/10.3201/eid2012.141026
- Calisher C.H., Childs J.E., Field H.E., Holmes K.V., Schountz T. Bats: important reservoir hosts of emerging viruses. Clin. Microbiol. Rev. 2006; 19(3): 531-45. DOI: https://doi.org/10.1128/CMR.00017-06
- Lvov D.K., Shchelkanov M.Y., Alkhovsky S.V., Deryabin P.G. Zoonotic Viruses of Northern Eurasia. Taxonomy and Ecology. London: Academic Press, Elsevier; 2015.
- Львов Д.К. Экология вирусов. В кн.: Львов Д.К., ред. Руководство по вирусологии. Вирусы и вирусные инфекции человека и животных. М.: МИА; 2013: 66-86.
- Li W., Moore M.J., Vasllieva N., Sui J., Wong S.K., Berne M.A., et al. Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus. Nature. 2003; 426(6965): 450-4. DOI: https://doi.org/10.1038/nature02145
- Ren W., Qu X., Li W., Han Z., Yu M., Zhou P., et al. Difference in receptor usage between severe acute respiratory syndrome (SARS) coronavirus and SARS-like coronavirus of bat origin. J. Virol. 2008; 82(4): 1899-907. DOI: https://doi.org/10.1128/jvi.01085-07
- Wan Y., Shang J., Graham R., Baric R.S., Li F. Receptor recognition by the novel coronavirus from Wuhan: an analysis based on decade-long structural studies of SARS coronavirus. J. Virol. 2020; 94(7): pii e00127-20. DOI: https://doi.org/10.1128/jvi.00127-20
- Ge X.Y., Li J.L., Yang X.L., Chmura A.A., Zhu G., Epstein J.H., et al. Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor. Nature. 2013; 503(7477): 535-8. DOI: https://doi.org/10.1038/nature12711
- Lau S.K., Feng Y., Chen H., Luk H.K., Yang W.H., Li K.S., et al. Severe acute respiratory syndrome (SARS) coronavirus ORF8 protein is acquired from SARS-related coronavirus from greater horseshoe bats through recombination. J. Virol. 2015; 89(20): 10532-47. DOI: https://doi.org/10.1128/jvi.01048-15
- Hu B., Zeng L.P., Yang X.L., Ge X.Y., Zhang W., Li B., et al. Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. PLoS Pathog. 2017; 13(11): e1006698. DOI: https://doi.org/10.1371/journal.ppat.1006698
- Ge X.Y., Wang N., Zhang W., Hu B., Li B., Zhang Y.Z., et al. Coexistence of multiple coronaviruses in several bat colonies in an abandoned mineshaft. Virol. Sin. 2016; 31(1): 31-40. DOI: https://doi.org/10.1007/s12250-016-3713-9
- Wang N., Li S.Y., Yang X.L., Huang H.M., Zhang Y.J., Guo H., et al. Serological evidence of bat SARS-related coronavirus infection in humans, China. Virol. Sin. 2018; 33(1): 104-7. DOI: http://doi.org/10.1007/s12250-018-0012-7
- Lam T.T., Shum M.H., Zhu H.C., Tong Y.G., Ni X.B., Liao Y.S., et al. Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins. Nature. 2020. DOI: https://doi.org/10.1038/s41586-020-2169-0
- Zhang T., Wu Q., Zhang Z. Probable pangolin origin of SARSCoV-2 associated with the COVID-19 outbreak. Curr. Biol. 2020; 30(7): 1346-51.e2. DOI: https://doi.org/10.1016/j.cub.2020.03.022
- Coutard B., Valle C., de Lamballerie X., Canard B., Seidah N.G., Decroly E. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. 2020; 176: 104742. DOI: https://doi.org/10.1016/j.antiviral.2020.104742
- Simmons G., Zmora P., Gierer S., Heurich A., Pöhlmann S. Proteolytic activation of the SARS-coronavirus spike protein: Cutting enzymes at the cutting edge of antiviral research. Antiviral Res. 2013; 100(3): 605-14. DOI: https://doi.org/10.1016/j.antiviral.2013.09.028
- Claas E.C.J., Osterhaus A.D., van Beek R., De Jong J.C., Rimmelzwaan G.F., Senne D.A., et al. Human influenza A H5N1 virus related to a highly pathogenic avian influenza virus. Lancet. 1998; 351(9101): 472-7. DOI: https://doi.org/10.1016/S0140-6736(97)11212-0
- Corman V.M., Ithete N.L., Richards L.R., Schoeman M.C., Preiser W., Drosten C., et al. Rooting the phylogenetic tree of Middle East respiratory syndrome coronavirus by characterization of a conspecific virus from an African bat. J. Virol. 2014; 88(19): 11297-303. DOI: https://doi.org/10.1128/jvi.01498-14
- Yang L., Wu Z., Ren X., Yang F., Zhang J., He G., et al. MERS-related betacoronavirus in Vespertilio superans bats, China. Emerg. Infect. Dis. 2014; 20(7): 1260-2. DOI: https://doi.org/10.3201/eid2007.140318
- Raj V.S., Mou H., Smits S.L., Dekkers D.H., Müller M.A., Dijkman R., et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC. Nature. 2003; 495(7440): 251-4. DOI: https://doi.org/10.1038/nature12005
- van Doremalen N., Miazgowicz K.L., Milne-Price S., Bushmaker T., Robertson S., Scott D., et al. Host species restriction of middle East respiratory syndrome coronavirus through its receptor, dipeptidyl peptidase 4. J. Virol. 2014; 88(16): 9220-32. DOI: https://doi.org/10.1128/JVI.00676-14
- Львов Д.К. Рождение и развитие вирусологии – история изучения новых и возвращающихся инфекций. Вопросы вирусологии. 2012; (S1): 5-20.
- Львов Д.К., ред. Методические рекомендации. Организация эколого-эпидемиологического мониторинга территорий Российской Федерации с целью противоэпидемической защиты населения и войск. М.; 1993.
- Львов Д.К., Борисевич С.В., Альховский С.В., Бурцева Е.И. Актуальные подходы к анализу вирусных геномов в интересах биобезопасности. Инфекционные болезни: новости, мнения, обучение. 2019; 8(2): 96-101. DOI: https://doi.org/10.24411/2305-3496-2019-12012
补充文件