Детекция Liman tick virus (неклассифицированный представитель Chuviridae) в культуре клеток клещей HAE/CTVM8
- Авторы: Литов А.Г.1,2, Щетинин А.М.3, Холодилов И.С.1, Белова О.А.1, Калянова А.С.1, Гущин В.А.3,4, Карганова Г.Г.1,2
-
Учреждения:
- ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
- Институт трансляционной медицины и биотехнологии ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
- ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
- ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
- Выпуск: Том 70, № 2 (2025)
- Страницы: 147-153
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://ogarev-online.ru/0507-4088/article/view/310653
- DOI: https://doi.org/10.36233/0507-4088-283
- EDN: https://elibrary.ru/hrnscv
- ID: 310653
Цитировать
Аннотация
Введение. Культуры клеток клещей широко используются для изучения биологии этих членистоногих и переносимых ими патогенов, в особенности вирусов. Большинство имеющихся в настоящее время культур клеток были получены из эмбриональных клеток клещей и могут быть инфицированы вирусами. Клеточная линия HAE/CTVM8, полученная из клещей Hyalomma anatolicum, часто используется для выделения переносимых клещами внутриклеточных инфекционных агентов.
Цель работы – изучение клеточной линии HAE/CTVM8 с помощью высокопроизводительного секвенирования с целью поиска вирусов в ней.
Материалы и методы. Культуральную жидкость клеток HAE/CTVM8 ультрацентрифугировали. Полученный осадок использовали для высокопроизводительного секвенирования после выделения РНК, реакции обратной транскрипции и синтеза второй цепи. Полученные прочтения фильтровали по длине и качеству в программе Trimmomatic, после чего собирали контиги с помощью программы SPAdes и анализировали их на присутствие вирусных последовательностей. Финальная сборка генома вируса осуществлялась в программе Ugene. Выравнивание последовательностей производили с использованием программы MAFFT. Построение филогенетических деревьев производилось с применением программы IQ-TREE.
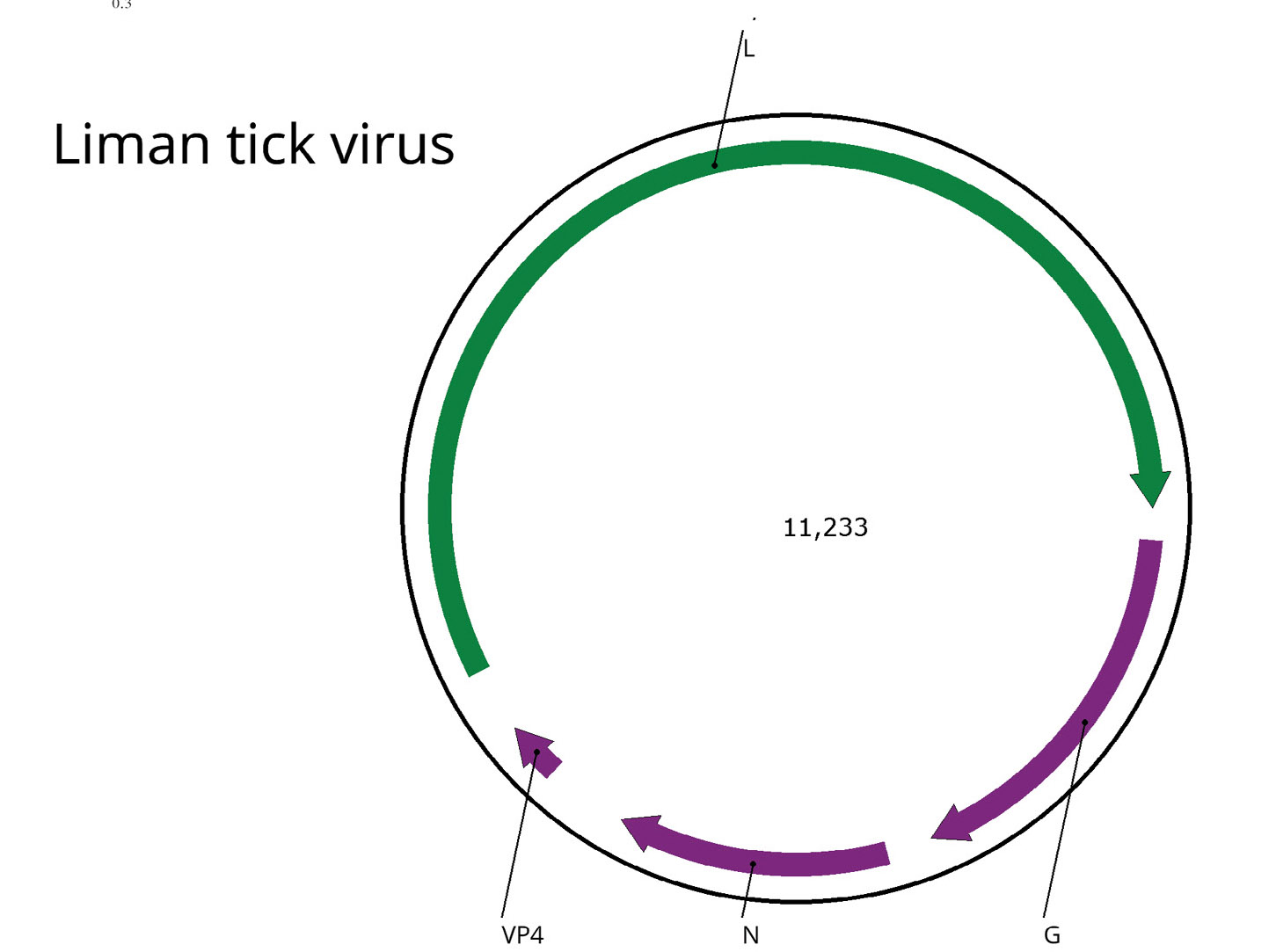
Результаты. Выявлена персистенция одного вируса – Liman tick virus (LMTV) – в культуре клеток HAE/CTVM8. Филогенетически LMTV принадлежит новому семейству Chuviridae, состоящему из вирусов, обнаруженных с помощью высокопроизводительного секвенирования, вирусологическая характеристика которых отсутствует.
Заключение. Полученная в настоящем исследовании информация крайне важна для использования культуры клеток HAE/CTVM8 в научных исследованиях и изоляции новых вирусов. Наше исследование показывает, что клеточная линия HAE/CTVM8 с персистирующим в ней LMTV представляет собой готовую систему для изучения репродукции представителей семейства Chuviridae.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Александр Геннадьевич Литов
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита); Институт трансляционной медицины и биотехнологии ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
Автор, ответственный за переписку.
Email: novosti-wxo@yandex.ru
ORCID iD: 0000-0002-6086-3655
канд. биол. наук, ведущий научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. Москва; 119991, г. МоскваАлексей Михайлович Щетинин
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России
Email: shchetinin.alexey@yandex.ru
ORCID iD: 0000-0003-1842-3899
научный сотрудник лаборатории механизмов популяционной изменчивости патогенных микроорганизмов
Россия, 123098, г. МоскваИван Сергеевич Холодилов
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
Email: ivan-kholodilov@bk.ru
ORCID iD: 0000-0002-3764-7081
канд. мед. наук, ведущий научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. МоскваОксана Андреевна Белова
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
Email: mikasusha@bk.ru
ORCID iD: 0000-0002-9040-0774
канд. биол. наук, ведущий научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. МоскваАнна Сергеевна Калянова
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита)
Email: annakalyanova@bk.ru
ORCID iD: 0009-0003-1154-3852
младший научный сотрудник лаборатории биологии арбовирусов
Россия, 108819, г. МоскваВладимир Алексеевич Гущин
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи» Минздрава России; ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: wowaniada@gmail.com
ORCID iD: 0000-0002-9397-3762
д-р биол. наук, ведущий научный сотрудник, заведующий лабораторией механизмов популяционной изменчивости патогенных микроорганизмов
Россия, 123098, г. Москва; 119991, г. МоскваГалина Григорьевна Карганова
ФГАНУ «Федеральный научный центр исследований и разработки иммунобиологических препаратов имени М.П. Чумакова РАН» (Институт полиомиелита); Институт трансляционной медицины и биотехнологии ФГАОУ ВО «Первый Московский государственный медицинский университет имени И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: karganova@bk.ru
ORCID iD: 0000-0002-8901-6206
д-р биол. наук, профессор, ведущий научный сотрудник, заведующая лабораторией биологии арбовирусов
Россия, 108819, г. Москва; 119991, г. МоскваСписок литературы
- Marques A.R., Strle F., Wormser G.P. Comparison of Lyme disease in the United States and Europe. Emerg. Infect. Dis. 2021; 27(8): 2017–24. https://doi.org/10.3201/eid2708.204763
- Ruzek D., Avšič Županc T., Borde J., Chrdle A., Eyer L., Karganova G., et al. Tick-borne encephalitis in Europe and Russia: Review of pathogenesis, clinical features, therapy, and vaccines. Antiviral. Res. 2019; 164: 23–51. https://doi.org/10.1016/j.antiviral.2019.01.014
- Bell-Sakyi L., Zweygarth E., Blouin E.F., Gould E.A., Jongejan F. Tick cell lines: tools for tick and tick-borne disease research. Trends. Parasitol. 2007; 23(9): 450–7. https://doi.org/10.1016/j.pt.2007.07.009
- Mangia C., Vismarra A., Kramer L., Bell-Sakyi L., Porretta D., Otranto D., et al. Evaluation of the in vitro expression of ATP binding-cassette (ABC) proteins in an Ixodes ricinus cell line exposed to ivermectin. Parasit. Vectors. 2016; 9: 215. https://doi.org/10.1186/s13071-016-1497-2
- Kholodilov I.S., Litov A.G., Klimentov A.S., Belova O.A., Polienko A.E., Nikitin N.A., et al. Isolation and characterisation of Alongshan virus in Russia. Viruses. 2020; 12(4): 362. https://doi.org/10.3390/v12040362
- Palomar A.M., Premchand-Branker S., Alberdi P., Belova O.A., Moniuszko-Malinowska A., Kahl O., et al. Isolation of known and potentially pathogenic tick-borne microorganisms from European ixodid ticks using tick cell lines. Ticks Tick Borne Dis. 2019; 10(3): 628–38. https://doi.org/10.1016/j.ttbdis.2019.02.008
- Husin N.A., Khoo J.J., Zulkifli M.M.S., Bell-Sakyi L., AbuBakar S. Replication kinetics of Rickettsia raoultii in tick cell lines. Microorganisms. 2021; 9(7): 1370. https://doi.org/10.3390/microorganisms9071370
- Salata C., Moutailler S., Attoui H., Zweygarth E., Decker L., Bell-Sakyi L. How relevant are in vitro culture models for study of tick-pathogen interactions? Pathog. Glob. Health. 2021; 115(7-8): 437–55. https://doi.org/10.1080/20477724.2021.1944539
- Bell-Sakyi L. Continuous cell lines from the tick Hyalomma anatolicum anatolicum. J. Parasitol. 1991; 77(6): 1006–8.
- Alberdi M.P., Dalby M.J., Rodriguez-Andres J., Fazakerley J.K., Kohl A., Bell-Sakyi L. Detection and identification of putative bacterial endosymbionts and endogenous viruses in tick cell lines. Ticks Tick Borne. Dis. 2012; 3(3): 137–46. https://doi.org/10.1016/j.ttbdis.2012.05.002
- Attoui H., Stirling J.M., Munderloh U.G., Billoir F., Brookes S.M., Burroughs J.N., et al. Complete sequence characterization of the genome of the St Croix River virus, a new orbivirus isolated from cells of Ixodes scapularis. J. Gen. Virol. 2001; 82(Pt. 4): 795–804. https://doi.org/10.1099/0022-1317-82-4-795
- Litov A.G., Shchetinin A.M., Kholodilov I.S., Belova O.A., Gadzhikurbanov M.N., Ivannikova A.Y., et al. High-throughput sequencing reveals three rhabdoviruses persisting in the IRE/CTVM19 cell line. Viruses. 2024; 16(4): 576. https://doi.org/10.3390/v16040576
- Harvey E., Rose K., Eden J.S., Lo N., Abeyasuriya T., Shi M., et al. Extensive diversity of RNA viruses in Australian ticks. J. Virol. 2019; 93(3): e01358–18. https://doi.org/10.1128/JVI.01358-18
- Li C.X., Shi M., Tian J.H., Lin X.D., Kang Y.J., Chen L.J., et al. Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses. Elife. 2015; 4: e05378. https://doi.org/10.7554/eLife.05378
- Ni X.B., Cui X.M., Liu J.Y., Ye R.Z., Wu Y.Q., Jiang J.F., et al. Metavirome of 31 tick species provides a compendium of 1,801 RNA virus genomes. Nat. Microbiol. 2023; 8(1): 162–73. https://doi.org/10.1038/s41564-022-01275-w
- Shi M., Lin X.D., Tian J.H., Chen L.J., Chen X., Li C.X., et al. Redefining the invertebrate RNA virosphere. Nature. 2016; 540(7634): 539–43. https://doi.org/10.1038/nature20167
- Kuhn J.H., Dheilly N.M., Junglen S., Paraskevopoulou S., Shi M., Di Paola N. ICTV virus taxonomy profile: Jingchuvirales 2023. J. Gen. Virol. 2023; 104(12): 001924. https://doi.org/10.1099/jgv.0.001924
- Kholodilov I.S., Belova O.A., Ivannikova A.Y., Gadzhikurbanov M.N., Makenov M.T., Yakovlev A.S., et al. Distribution and characterisation of tick-borne flavi-, flavi-like, and phenuiviruses in the Chelyabinsk region of Russia. Viruses. 2022; 14(12): 2699. https://doi.org/10.3390/v14122699
- Salata C., Monteil V., Karlberg H., Celestino M., Devignot S., Leijon M., et al. The DEVD motif of Crimean-Congo hemorrhagic fever virus nucleoprotein is essential for viral replication in tick cells. Emerg. Microbes Infect. 2018; 7(1): 190. https://doi.org/10.1038/s41426-018-0192-0
- Salvati M.V., Salaris C., Monteil V., Del Vecchio C., Palù G., Parolin C., et al. Virus-derived DNA forms mediate the persistent infection of tick cells by Hazara virus and Crimean-Congo hemorrhagic fever virus. J. Virol. 2021; 95(24): e0163821. https://doi.org/10.1128/JVI.01638-21
- Kholodilov I.S., Belova O.A., Morozkin E.S., Litov A.G., Ivannikova A.Y., Makenov M.T., et al. Geographical and tick-dependent distribution of flavi-like Alongshan and Yanggou tick viruses in Russia. Viruses. 2021; 13(3): 458. https://doi.org/10.3390/v13030458
- Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014; 30(15): 2114–20. https://doi.org/10.1093/bioinformatics/btu170
- Lo C.C., Chain P.S. Rapid evaluation and quality control of next generation sequencing data with FaQCs. BMC Bioinformatics. 2014; 15(1): 366. https://doi.org/10.1186/s12859-014-0366-2
- Litov A.G., Semenyuk I.I., Belova O.A., Polienko A.E., Thinh N.V., Karganova G.G., et al. Extensive diversity of viruses in millipedes collected in the Dong Nai biosphere reserve (Vietnam). Viruses. 2024; 16(9): 1486. https://doi.org/10.3390/v16091486
- Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: a unified bioinformatics toolkit. Bioinformatics. 2012; 28(8): 1166–7. https://doi.org/10.1093/bioinformatics/bts091
- Katoh K., Standley D.M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Mol. Biol. Evol. 2013; 30(4): 772–80. https://doi.org/10.1093/molbev/mst010
- Capella-Gutiérrez S., Silla-Martínez J.M., Gabaldón T. trimAl: a tool for automated alignment trimming in large-scale phylogenetic analyses. Bioinformatics. 2009; 25(15): 1972–3. https://doi.org/10.1093/bioinformatics/btp348
- Nguyen L.T., Schmidt H.A., von Haeseler A., Minh B.Q. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol. Biol. Evol. 2015; 32(1): 268–74. https://doi.org/10.1093/molbev/msu300
- Kalyaanamoorthy S., Minh B.Q., Wong T.K.F., von Haeseler A., Jermiin L.S. ModelFinder: fast model selection for accurate phylogenetic estimates. Nat. Methods. 2017; 14(6): 587–9. https://doi.org/10.1038/nmeth.4285
- Zhang Y., Hu B., Agwanda B., Fang Y., Wang J., Kuria S., et al. Viromes and surveys of RNA viruses in camel-derived ticks revealing transmission patterns of novel tick-borne viral pathogens in Kenya. Emerg. Microbes Infect. 2021; 10(1): 1975–87. https://doi.org/10.1080/22221751.2021.1986428
- Abrao E.P., da Fonseca B.A. Infection of mosquito cells (C6/36) by Dengue-2 virus interferes with subsequent infection by yellow fever virus. Vector. Borne Zoonotic Dis. 2016; 16(2): 124–30. https://doi.org/10.1089/vbz.2015.1804
- Kuwata R., Isawa H., Hoshino K., Sasaki T., Kobayashi M., Maeda K., et al. Analysis of mosquito-borne flavivirus superinfection in Culex tritaeniorhynchus (Diptera: Culicidae) cells persistently infected with Culex flavivirus (Flaviviridae). J. Med. Entomol. 2015; 52(2): 222–9. https://doi.org/10.1093/jme/tju059
- Patterson E.I., Kautz T.F., Contreras-Gutierrez M.A., Guzman H., Tesh R.B., Hughes G.L., et al. Negeviruses reduce replication of alphaviruses during coinfection. J. Virol. 2021; 95(14): e0043321. https://doi.org/10.1128/JVI.00433-21
Дополнительные файлы