Оценка профилактического эффекта нокдауна клеточных генов NXF1, PRPS1 и NAA10 при гриппозной инфекции на модели in vitro
- Авторы: Пашков Е.А.1,2, Шиквин Д.А.3, Пашков Г.А.1,2, Нагиева Ф.Г.1, Богданова Е.А.2, Быков А.С.2, Пашков Е.П.2, Свитич О.А.1,2, Зверев В.В.1,2
-
Учреждения:
- ФГБУН «Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова» Минздрава России
- ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
- ФГБОУ ВО «МИРЭА – Российский технологический университет»
- Выпуск: Том 70, № 1 (2025)
- Страницы: 66-77
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://ogarev-online.ru/0507-4088/article/view/287911
- DOI: https://doi.org/10.36233/0507-4088-289
- EDN: https://elibrary.ru/oqonmm
- ID: 287911
Цитировать
Аннотация
Введение. Грипп – острое респираторное вирусное инфекционное заболевание, индуцируемое одноименными вирусами. Существующие на сегодняшний день профилактические и терапевтические подходы имеют важное противоэпидемическое значение, однако имеется ряд проблем, таких как быстрое возникновение резистентных штаммов, отсутствие формирования перекрестного иммунитета и эффективность вакцин. Одним из подходов в создании противогриппозных средств является использование механизма РНК-интерференции и малых интерферирующих РНК (миРНК), комплементарных к матричной РНК мишени вирусных и клеточных генов.
Цель – оценка профилактического противогриппозного эффекта миРНК, направленных к клеточным генам NXF1, PRPS1 и NAA10, на модели in vitro.
Материалы и методы. Исследовали антигенные варианты вируса гриппа типа А: A/California/7/09 (H1N1), А/WSN/33 (H1N1) и A/Brisbane/59/07 (H1N1); клеточные культуры A549 и MDCK. Исследование выполняли посредством молекулярно-генетических (трансфекции, выделение нуклеиновых кислот, полимеразная цепная реакция с обратной транскрипцией в реальном времени) и вирусологических методов (заражение клеточных культур, титрование по визуальному цитопатическому действию, оценка вирусного титра посредством метода Рамакришнана).
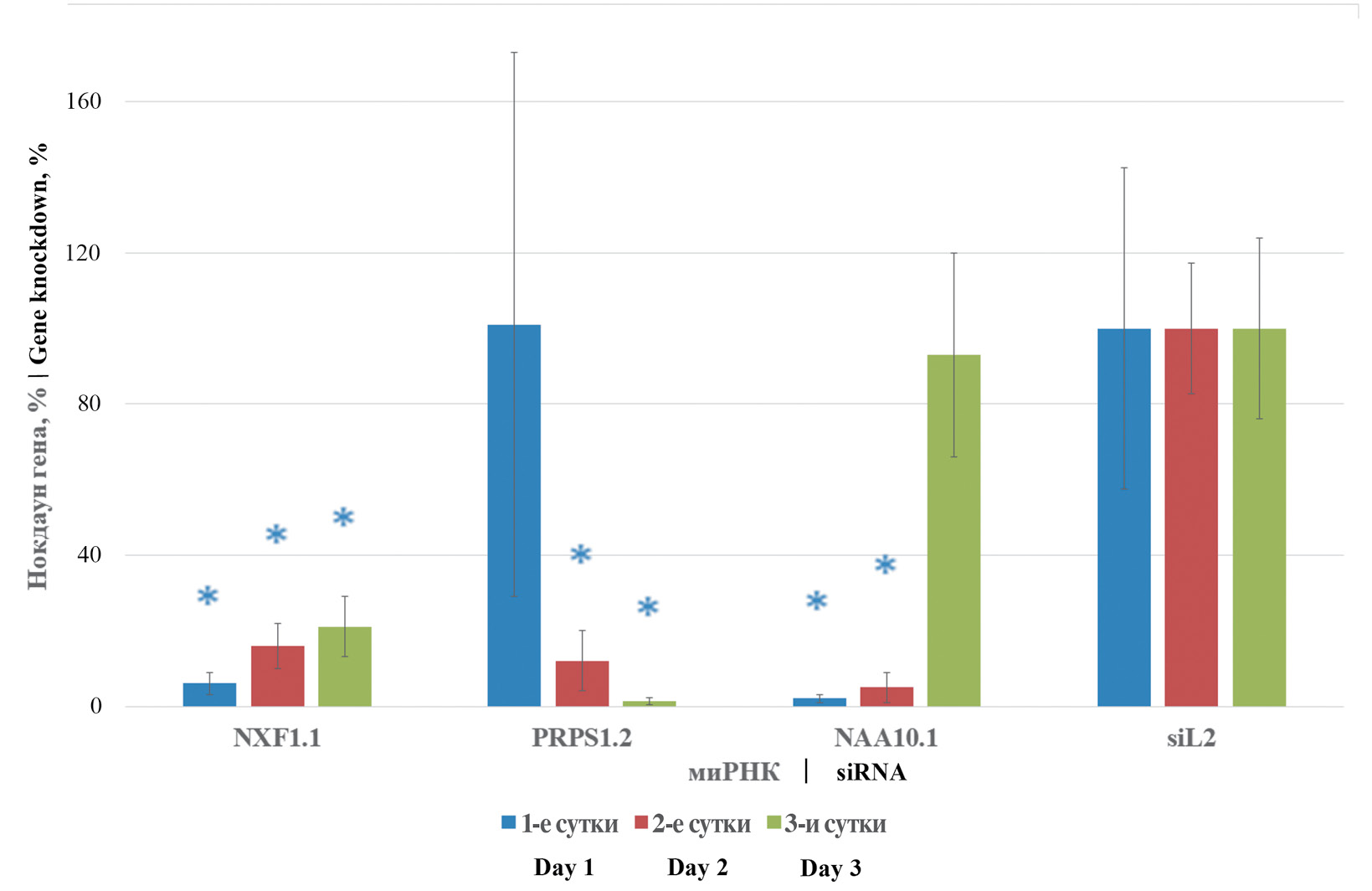
Результаты. Показано, что миРНК, таргетированные к клеточным генам NXF1, PRPS1 и NAA10 при профилактическом применении в клеточной культуре в концентрации 0,25 мкг на лунку, при инфицировании штаммами вируса гриппа A/California/7/09 (H1N1), А/WSN/33 (H1N1) и A/Brisbane/59/07 (H1N1) при множественности инфекции 0,01, снижают вирусную репликацию до уровня 220 ТЦД50 на 1 мл клеточной среды, тогда как в контрольных необработанных клетках вирусный урожай составил ~106 ТЦД50 на 1 мл среды.
Выводы. Снижение экспрессии указанных генов NXF1, PRPS1 и NAA10 приводит к нарушению жизненного цикла и активности вирусов гриппа. Подобный подход может быть потенциально исследован и использован для близко- и дальнородственных представителей иных семейств вирусов.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Евгений Алексеевич Пашков
ФГБУН «Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова» Минздрава России; ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Автор, ответственный за переписку.
Email: pashckov.j@yandex.ru
ORCID iD: 0000-0002-5682-4581
канд. мед. наук, ассистент кафедры микробиологии, вирусологии и иммунологии ФГАОУ ВО Первый МГМУ им. И.М. Сеченова Минздрава России (Сеченовский Университет); младший научный сотрудник лаборатории прикладной вирусологии ФГБНУ НИИВС им. И.И. Мечникова
Россия, 105064, г. Москва; 119991, г. МоскваДмитрий Андреевич Шиквин
ФГБОУ ВО «МИРЭА – Российский технологический университет»
Email: carrypool@gmail.com
ORCID iD: 0009-0005-9874-2081
студент кафедры биотехнологии и промышленной фармации института тонких химических технологий им. М.В. Ломоносова
Россия, 119454, г. МоскваГеоргий Алексеевич Пашков
ФГБУН «Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова» Минздрава России; ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: georgp2004@mail.ru
ORCID iD: 0000-0003-0392-9969
студент клинического института детского здоровья им. Н.Ф. Филатова
Россия, 105064, г. Москва; 119991, г. МоскваФирая Галиевна Нагиева
ФГБУН «Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова» Минздрава России
Email: fgn42@yandex.ru
ORCID iD: 0000-0001-8204-4899
д-р мед. наук, доцент, заведующая лабораторией гибридных клеточных культур
Россия, 105064, г. МоскваЕкатерина Александровна Богданова
ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: bogdekaterin@yandex.ru
ORCID iD: 0000-0002-5620-1843
канд. мед. наук, доцент кафедры микробиологии, вирусологии и иммунологии
Россия, 119991, г. МоскваАнатолий Сергеевич Быков
ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: drbykov@bk.ru
ORCID iD: 0000-0002-8099-6201
д-р мед. наук, профессор кафедры микробиологии, вирусологии и иммунологии
Россия, 119991, г. МоскваЕвгений Петрович Пашков
ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: 9153183256@mail.ru
ORCID iD: 0000-0002-4963-5053
д-р мед. наук, профессор кафедры микробиологии, вирусологии и иммунологии
Россия, 119991, г. МоскваОксана Анатольевна Свитич
ФГБУН «Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова» Минздрава России; ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: svitichoa@yandex.ru
ORCID iD: 0000-0003-1757-8389
чл.-корр. РАН, д-р мед. наук, директор ФГБНУ НИИВС им И.И. Мечникова, заведующая лабораторией молекулярной иммунологии ФГБНУ НИИВС им. И.И. Мечникова, профессор кафедры микробиологии, вирусологии и иммунологии ФГАОУ ВО Первый МГМУ им. И.М. Сеченова Минздрава России (Сеченовский Университет)
Россия, 105064, г. Москва; 119991, г. МоскваВиталий Васильевич Зверев
ФГБУН «Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова» Минздрава России; ФГАОУ ВО «Первый Московский государственный медицинский университет им. И.М. Сеченова» Минздрава России (Сеченовский Университет)
Email: vitalyzverev@outlook.com
ORCID iD: 0000-0002-0017-1892
академик РАН, д-р биол. наук, научный руководитель ФГБНУ НИИВС им. И.И. Мечникова, профессор, заведующий кафедрой микробиологии, вирусологии и иммунологии ФГАОУ ВО Первый МГМУ им. И.М. Сеченова Минздрава России (Сеченовский Университет)
Россия, 105064, г. Москва; 119991, г. МоскваСписок литературы
- Purcell R., Giles M.L., Crawford N.W., Buttery J. Systematic review of avian influenza virus infection and outcomes during pregnancy. Emerg. Infect. Dis. 2025; 31(1): 50–6. https://doi.org/10.3201/eid3101.241343
- Bin N.R., Prescott S.L., Horio N., Wang Y., Chiu I.M., Liberles S.D. An airway-to-brain sensory pathway mediates influenza-induced sickness. Nature. 2023; 615(7953): 660–7. https://doi.org/10.1038/s41586-023-05796-0
- Kenney A.D., Aron S.L., Gilbert C., Kumar N., Chen P., Eddy A., et al. Influenza virus replication in cardiomyocytes drives heart dysfunction and fibrosis. Sci. Adv. 2022; 8(19): eabm5371. https://doi.org/10.1126/sciadv.abm5371
- Conrad A., Valour F., Vanhems P. Burden of influenza in the elderly: a narrative review. Curr. Opin. Infect. Dis. 2023; 36(4): 296–302. https://doi.org/10.1097/QCO.0000000000000931
- Watanabe T. Renal complications of seasonal and pandemic influenza A virus infections. Eur. J. Pediatr. 2013; 172(1): 15–22. https://doi.org/10.1007/s00431-012-1854-x
- van de Veerdonk F.L., Wauters J., Verweij P.E. Invasive aspergillus tracheobronchitis emerging as a highly lethal complication of severe influenza. Am. J. Respir. Crit. Care Med. 2020; 202(5): 646–8. https://doi.org/10.1164/rccm.202005-1883ED
- Feys S., Cardinali-Benigni M., Lauwers H.M., Jacobs C., Stevaert A., Gonçalves S.M., et al. Profiling bacteria in the lungs of patients with severe influenza versus COVID-19 with or without aspergillosis. Am. J. Respir. Crit. Care Med. 2024; 210(10): 1230–42. https://doi.org/10.1164/rccm.202401-0145OC
- Białka S., Zieliński M., Latos M., Skurzyńska M., Żak M., Palaczyński P., et al. Severe bacterial superinfection of influenza pneumonia in immunocompetent young patients: case reports. J. Clin. Med. 2024; 13(19): 5665. https://doi.org/10.3390/jcm13195665
- Pleguezuelos O., James E., Fernandez A., Lopes V., Rosas L.A., Cervantes-Medina A., et al. Efficacy of FLU-v, a broad-spectrum influenza vaccine, in a randomized phase IIb human influenza challenge study. NPJ Vaccines. 2020; 5(1): 22. https://doi.org/10.1038/s41541-020-0174-9
- Isakova-Sivak I., Rudenko L. Next-generation influenza vaccines based on mRNA technology. Lancet Infect. Dis. 2025; 25(1): 2–3. https://doi.org/10.1016/S1473-3099(24)00562-0
- Hodgson D., Sánchez-Ovando S., Carolan L., Liu Y., Hadiprodjo A.J., Fox A., et al. Quantifying the impact of pre-vaccination titre and vaccination history on influenza vaccine immunogenicity. Vaccine. 2025; 44: 126579. https://doi.org/10.1016/j.vaccine.2024.126579
- Gaitonde D.Y., Moore F.C., Morgan M.K. Influenza: diagnosis and treatment. Am. Fam. Physician. 2019; 100(12): 751–8.
- Li Y., Huo S., Yin Z., Tian Z., Huang F., Liu P., et al. The current state of research on influenza antiviral drug development: drugs in clinical trial and licensed drugs. mBio. 2023; 14(5): e0127323. https://doi.org/10.1128/mbio.01273-23
- Wang J., Li Y. Current advances in antiviral RNA interference in mammals. FEBS J. 2024; 291(2): 208–16. https://doi.org/10.1111/febs.16728
- Traber G.M., Yu A.M. RNAi-based therapeutics and novel RNA bioengineering technologies. J. Pharmacol. Exp. Ther. 2023; 384(1): 133–54. https://doi.org/10.1124/jpet.122.001234
- Qureshi A., Tantray V.G., Kirmani A.R., Ahangar A.G. A review on current status of antiviral siRNA. Rev. Med. Virol. 2018; 28(4): e1976. https://doi.org/10.1002/rmv.1976
- Chokwassanasakulkit T., Oti V.B., Idris A., McMillan N.A. SiRNAs as antiviral drugs – Current status, therapeutic potential and challenges. Antiviral. Res. 2024; 232: 106024. https://doi.org/10.1016/j.antiviral.2024.106024
- Wang L., Dai X., Song H., Yuan P., Yang Z., Dong W., et al. Inhibition of porcine transmissible gastroenteritis virus infection in porcine kidney cells using short hairpin RNAs targeting the membrane gene. Virus Genes. 2017; 53(2): 226–32. https://doi.org/10.1007/s11262-016-1409-8
- Lambeth L.S., Zhao Y., Smith L.P., Kgosana L., Nair V. Targeting Marek’s disease virus by RNA interference delivered from a herpesvirus vaccine. Vaccine. 2009; 27(2): 298–306. https://doi.org/10.1016/j.vaccine.2008.10.023
- Chen W., Liu M., Jiao Y., Yan W., Wei X., Chen J., et al. Adenovirus-mediated RNA interference against foot-and-mouth disease virus infection both in vitro and in vivo. J. Virol. 2006; 80(7): 3559–66. https://doi.org/10.1128/JVI.80.7.3559-3566.2006
- Keene K.M., Foy B.D., Sanchez-Vargas I., Beaty B.J., Blair C.D., Olson K.E. RNA interference acts as a natural antiviral response to O’nyong-nyong virus (Alphavirus; Togaviridae) infection of Anopheles gambiae. Proc. Natl. Acad. Sci. USA. 2004; 101(49): 17240–5. https://doi.org/10.1073/pnas.0406983101
- Ahmed F., Kleffmann T., Husain M. Acetylation, methylation and allysine modification profile of viral and host proteins during influenza A virus infection. Viruses. 2021; 13(7): 1415. https://doi.org/10.3390/v13071415
- Izumi H. Conformational variability prediction of influenza virus hemagglutinins with amino acid mutations using supersecondary structure code. Methods. Mol. Biol. 2025; 2870: 63–78. https://doi.org/10.1007/978-1-0716-4213-9_5
- Lesch M., Luckner M., Meyer M., Weege F., Gravenstein I., Raftery M., et al. RNAi-based small molecule repositioning reveals clinically approved urea-based kinase inhibitors as broadly active antivirals. PLoS Pathog. 2019; 15(3): e1007601. https://doi.org/101371/journal.ppat.1007601
- Li X., Berg N.K., Mills T., Zhang K., Eltzschig H.K., Yuan X. Adenosine at the interphase of hypoxia and inflammation in lung injury. Front. Immunol. 2021; 11: 604944. https://doi.org/10.3389/fimmu.2020.604944
- Read E.K., Digard P. Individual influenza A virus mRNAs show differential dependence on cellular NXF1/TAP for their nuclear export. J. Gen. Virol. 2010; 91(Pt. 5): 1290–301. https://doi.org/10.1099/vir.0.018564-0
- Ramakrishnan M.A. Determination of 50% endpoint titer using a simple formula. World J. Virol. 2016; 5(2): 85–6. https://doi.org/10.5501/wjv.v5.i2.85
- Пашков Е.А., Самойликов Р.В., Пряников Г.А., Быков А.С., Пашков Е.П., Поддубиков А.В. и др. Иммуномодулирующий эффект комплексов миРНК in vitro при гриппозной инфекции. Российский иммунологический журнал. 2023; 26(4): 457–62. https://doi.org/10.46235/1028-7221-13984-IVI httpss://elibrary.ru/byxobk
- Estrin M.A., Hussein I.T.M., Puryear W.B., Kuan A.C., Artim S.C., Runstadler J.A. Host-directed combinatorial RNAi improves inhibition of diverse strains of influenza A virus in human respiratory epithelial cells. PLoS One. 2018; 13(5): e0197246. https://doi.org/10.1371/journal.pone.0197246
- Файзулоев Е.Б. Изучение противовирусной активности антисмысловых РНК и рибозимов в отношении инфекции, вызываемой вирусом алеутской болезни норок: Автореф. диcс. ... канд. биол. наук. М.; 2002.
- Lee H.K., Loh T.P., Lee C.K., Tang J.W., Chiu L., Koay E.S. A universal influenza A and B duplex real-time RT-PCR assay. J. Med. Virol. 2012; 84(10): 1646–51. https://doi.org/10.1002/jmv.23375
- Bustin S.A., Benes V., Nolan T., Pfaffl M.W. Quantitative real-time RT-PCR – a perspective. J. Mol. Endocrinol. 2005; 34(3): 597–601. https://doi.org/10.1677/jme.1.01755
- Howard C.W., Zou G., Morrow S.A., Fridman S., Racosta J.M. Wilcoxon-Mann-Whitney odds ratio: A statistical measure for ordinal outcomes such as EDSS. Mult. Scler. Relat. Disord. 2022; 59: 103516. https://doi.org/10.1016/j.msard.2022.103516
- Ge Q., Filip L., Bai A., Nguyen T., Eisen H.N., Chen J. Inhibition of influenza virus production in virus-infected mice by RNA interference. Proc. Natl. Acad. Sci. USA. 2004; 101(23): 8676–81. https://doi.org/10.1073/pnas.0402486101
- Sui H.Y., Zhao G.Y., Huang J.D., Jin D.Y., Yuen K.Y., Zheng B.J. Small interfering RNA targeting M2 gene induces effective and long-term inhibition of influenza A virus replication. PLoS One. 2009; 4(5): 5671. https://doi.org/10.1371/journal.pone.0005671
- Piasecka J., Lenartowicz E., Soszynska-Jozwiak M., Szutkowska B., Kierzek R., Kierzek E. RNA secondary structure motifs of the influenza A virus as targets for siRNA-mediated RNA interference. Mol. Ther. Nucleic. Acids. 2020; 19: 627–42. https://doi.org/10.1016/j/omtn.2019.12.018
- Zhou Y., Liu Y., Gupta S., Paramo M.I., Hou Y., Mao C., et al. A comprehensive SARS-CoV-2-human protein-protein interactome reveals COVID-19 pathobiology and potential host therapeutic targets. Nat. Biotechnol. 2023; 41(1): 128–39. https://doi.org/10.1038/s41587-022-01474-0
- Hu J., Zhang L., Liu X. Role of post-translational modifications in influenza A virus life cycle and host innate immune response. Front. Microbiol. 2020; 11: 517461. https://doi.org/10.3389/fmicb.2020.517461
- Zhang K., Cagatay T., Xie D., Angelos A.E., Cornelius S., Aksenova V., et al. Cellular NS1-BP protein interacts with the mRNA export receptor NXF1 to mediate nuclear export of influenza virus M mRNAs. J. Biol. Chem. 2024; 300(11): 107871. https://doi.org/10.1016/j.jbc.2024.107871
- Esparza M., Bhat P., Fontoura B.M. Viral-host interactions during splicing and nuclear export of influenza virus mRNAs. Curr. Opin. Virol. 2022; 55: 101254. https://doi.org/10.1016/j.coviro.2022.101254
- Dawson A.R., Wilson G.M., Coon J.J., Mehle A. Post-Translation Regulation of Influenza Virus Replication. Annu. Rev. Virol. 2020; 7(1): 167–87. https://doi.org/10.1146/annurev-virology-010320-070410
- Husain M. Influenza A virus and acetylation: the picture is becoming clearer. Viruses. 2024; 16(1): 131. https://doi.org/10.3390/v16010131
- Mei M., Cupic A., Miorin L., Ye C., Cagatay T., Zhang K., et al. Inhibition of mRNA nuclear export promotes SARS-CoV-2 pathogenesis. Proc. Natl. Acad. Sci. USA. 2024; 121(22): e2314166121. https://doi.org/10.1073/pnas.2314166121
- Zhang K., Xie Y., Muñoz-Moreno R., Wang J., Zhang L., Esparza M., et al. Structural basis for influenza virus NS1 protein block of mRNA nuclear export. Nat. Microbiol. 2019; 4(10): 1671–9. https://doi.org/10.1038/s41564-019-0482-x
- Wendt L., Brandt J., Bodmer B.S., Reiche S., Schmidt M.L., Traeger S., et al. The Ebola virus nucleoprotein recruits the nuclear RNA export factor NXF1 into inclusion bodies to facilitate viral protein expression. Cells. 2020; 9(1): 187. https://doi.org/10.3390/cells9010187
- Chen J., Umunnakwe C., Sun D.Q., Nikolaitchik O.A., Pathak V.K., Berkhout B., et al. Impact of nuclear export pathway on cytoplasmic HIV-1 RNA transport mechanism and distribution. mBio. 2020; 11(6): e01578–20. https://doi.org/10.1128/mBio.01578-20
- Guo J., Zhu Y., Ma X., Shang G., Liu B., Zhang K. Virus infection and mRNA nuclear export. Int. J. Mol. Sci. 2023; 24(16): 12593. https://doi.org/10.3390/ijms241612593
- Пак А.В., Пашков Е.А., Абрамова Н.Д., Поддубиков А.В., Нагиева Ф.Г., Богданова Е.А. и др. Действие противовирусных миРНК на выработку цитокинов in vitro. Тонкие химические технологии. 2022; 17(5): 384–93. httpss://doi.org/10.32362/2410-6593-2022-17-5-384-393 httpss://elibrary.ru/meflst
- Banerjee A., Mukherjee S., Maji B.K. Manipulation of genes could inhibit SARS-CoV-2 infection that causes COVID-19 pandemics. Exp. Biol. Med. (Maywood). 2021; 246(14): 1643–9. https://doi.org/10.1177/15353702211008106
Дополнительные файлы