Моноклональные антитела к гемагглютинину вируса гриппа А/Н7N3 (Orthomyxoviridae: Alphainfluenzavirus: Influenza A virus)
- Авторы: Сорокин Е.В.1, Царёва Т.Р.1, Руднева И.А.2, Тимофеев Б.И.2, Ляшко А.В.2, Баланова М.А.2, Артёмов Е.К.2, Гребенникова Т.В.2, Тимофеева Т.А.2
-
Учреждения:
- ФГБУ «Научно-исследовательский институт гриппа им. А.А. Смородинцева» Минздрава России
- Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
- Выпуск: Том 66, № 3 (2021)
- Страницы: 189-197
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://ogarev-online.ru/0507-4088/article/view/118161
- DOI: https://doi.org/10.36233/0507-4088-45
- ID: 118161
Цитировать
Полный текст
Аннотация
Введение. Варианты вируса гриппа (ВГ) А подтипа Н7, как и Н5, обладают высоким пандемическим потенциалом. Однако имеющиеся сведения об антигенной структуре гемагглютинина (HA) Н7 значительно уступают по объёму аналогичным данным в отношении НА подтипа Н5.
Цели исследования – разработка и характеристика панели моноклональных антител (МКАТ), направленных к НА подтипа Н7 возбудителя гриппа А.
Материал и методы. Культуру вируса накапливали в 10-дневных куриных эмбрионах. Очистку и концентрацию вирусных частиц, определение концентрации белка, получение МКАТ и асцитных жидкостей, реакцию гемагглютинации (РГА) и реакцию торможения гемагглютинации (РТГА), оценку активности антител в непрямом иммуноферментном анализе (ИФА), а также определение изотипов МКАТ и реакцию нейтрализации (РН) проводили стандартными способами.
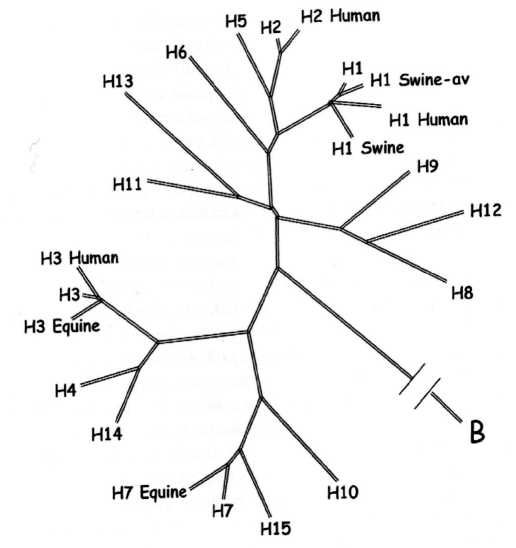
Результаты. Полученные МКАТ к штамму А/mallard/Netherlands/12/2000 (H7N3) исследованы в РТГА с набором штаммов разных лет выделения, относящихся к различным эволюционным группам. Во всех случаях антитела обладали сниженной реакционной способностью по сравнению с вирусом-иммуногеном. Выявлено перекрёстное взаимодействие МКАТ 9E11 и 9G12 в РТГА с ВГ А/H15.
Обсуждение. Возбудитель гриппа А с НА подтипа Н7 может послужить потенциальным агентом будущей пандемии. Разработка панели МКАТ к НА этого подтипа представляется актуальной задачей как для ветеринарии, так и для общественного здравоохранения.
Заключение. Полученные нами антитела могут найти применение не только для эпитопного картирования НA подтипа вируса Н7 (которое к настоящему времени недостаточно разработано) и в качестве реагентов тест-систем, но и с целью определения общих («универсальных») эпитопов в данной молекуле у разных штаммов Н7.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Е. В. Сорокин
ФГБУ «Научно-исследовательский институт гриппа им. А.А. Смородинцева» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0003-1732-1727
197376, Санкт-Петербург
РоссияТ. Р. Царёва
ФГБУ «Научно-исследовательский институт гриппа им. А.А. Смородинцева» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0003-4757-0521
197376, Санкт-Петербург
РоссияИ. А. Руднева
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0001-5000-2547
123098, Москва
РоссияБ. И. Тимофеев
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0001-7425-0457
123098, Москва
РоссияА. В. Ляшко
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0001-5714-9461
123098, Москва
РоссияМ. А. Баланова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0003-4151-4123
123098, Москва
РоссияЕ. К. Артёмов
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0002-6786-6357
123098, Москва
РоссияТ. В. Гребенникова
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Email: fake@neicon.ru
ORCID iD: 0000-0002-6141-9361
123098, Москва
РоссияТ. А. Тимофеева
Институт вирусологии им. Д.И. Ивановского ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени почётного академика Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: timofeeva.tatyana@inbox.ru
ORCID iD: 0000-0002-8991-8525
Тимофеева Татьяна Анатольевна, заведующая лабораторией физиологии вирусов
123098, Москва
РоссияСписок литературы
- Львов Д.К., ред. Вирусы и вирусные инфекции человека и животных. М.: МИА; 2013. 2. Elbers A.R., Fabri T.H., de Vries T.S., de Wit J.J., Pijpers A., Koch G. The highly pathogenic avian influenza A (H7N7) virus epidemic in the Netherlands in 2003 – lessons learned from the first five outbreaks. Avian Dis. 2004; 48(3): 691–705. https://doi.org/10.1637/7149
- Kemink S.A., Fouchier R.A., Rozendaal F.W., Broekman J.M., Koopmans M., Osterhaus A.D., et al. A fatal infection due to avian influenza-A (H7N7) virus and adjustment of the preventive measures. Ned. Tijdschr. Geneeskd. 2004; 148(44): 2190–4.
- WHO. Overview of the emergence and characteristics of the avian influenza A(H7N9) virus. Available at: http://www.who.int/influenza/human_animal-interface/influenza-h7n9/WHO_H7N9_review_ 31May13.pdf (accessed May 14, 2021).
- Schmeiser F., Vasudevan A., Verma S., Wang W., Alvarad E., Weiss C., et al. Antibodies to antigenic site A of influenza H7 hemagglutinin provide protection against H7N9 challenge. PLoS One. 2015; 10(1): e0117108. https://doi.org/10.1371/journal.pone.0117108
- Thornburg N.J., Zhang H., Bangaru S., Sapparapu G., Kose N., Lampley R.M., et al. H7N9 influenza virus neutralizing antibodies that possess few somatic mutations. J. Clin. Invest. 2016; 126(4): 1482–94. https://doi.org/10.1172/jci85317
- Yao L., Chen Y., Wang X., Bi Z., Xiao Q., Lei J., et al. Identification of antigenic epitopes in the haemagglutinin of H7 avian influenza virus. Avian. Pathol. 2020; 49(1): 62–73. https://doi.org/10.1080/0 3079457.2019.1666971
- Седова Е.С., Верховская Л.В., Артёмова Э.А., Щербинин Д.Н., Лысенко А.А., Руднева И.А., и др. Защита мышей от заражения вирусом гриппа птиц субтипа Н7 с помощью иммунизации рекомбинантным аденовирусом, кодирующим консервативные антигены вируса гриппа А. БИОпрепараты. Профилактика, диагностика, лечение. 2020; 20(1): 60–7. https://doi. org/10.30895/2221-996X-2020-20-1-60-67
- Суховецкая В.Ф., Дондурей Е.А., Дриневский В.П., Соминина А.А., Майорова В.Г., Писарева М.М., и др. Методические рекомендации. Выделение вирусов гриппа в клеточных культурах и куриных эмбрионах и их идентификация. Санкт-Петербург; 2006.
- Сорокин Е.В., Царёва Т.Р., Желтухина А.И. Моноклональные антитела к гемагглютинину вирусов гриппа В викторианской эволюционной линии. Вопросы вирусологии. 2018; 63(6): 275– 80. https://doi.org/10.18821/0507-4088-2018-63-6-275-280
- Price M.N., Dehal P.S., Arkin A.P. FastTree 2-approximately maximum-likelihood trees for large alignments. PLoS One. 2010; 5(3): e9490. https://doi.org/10.1371/journal.pone.0009490
- Le S.Q., Gascuel O. An improved general amino acid replacement matrix. Mol. Biol. Evol. 2008; 25(7): 1307–20. https://doi. org/10.1093/molbev/msn067
- Stamatakis A. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies. Bioinformatics. 2014; 30(9): 1312–3. https://doi.org/10.1093/bioinformatics/btu033
- Dang C.C., Le Q.S., Gascuel O., Le V.S. FLU, an amino acid substitution model for influenza proteins. BMC Evol. Biol. 2010; 10: 99. https://doi.org/10.1186/1471-2148-10-99
- Кущ А.А., Климова Р.Р., Масалова О.В., Фёдорова Н.Е., Ботиков А.Г., Федякина И.Т., и др. Получение и свойства моноклональных антител к высокопатогенному штамму вируса гриппа птиц A(H5N1), выделенного на территории Российской Федерации. Вопросы вирусологии. 2008; 53(5): 9–14.
- Климова Р.Р., Масалова О.В., Бурцева Е.И., Чичев Е.В., Леснова Е.И., Оскерко Т.А., и др. Моноклональные антитела к пандемическому вирусу гриппа A/IIV-Moscow/01/2009 (H1N1)swl, обладающие высокой вируснейтрализующей активностью. Вопросы вирусологии. 2011; 56(3): 15–20.
- Matrosovich M.N., Klenk H.-D., Kawaoka Y. Receptor specificity, host-range, and pathogenicity of influenza viruses. In: Kawaoka Y., ed. Influenza Virology: Current Topics. Wymondham, UK: Caister Academic Press; 2006: 95–138.
- Sievers F., Wilm A., Dineen D.G., Gibson T.J., Karplus K., Li W., et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Mol. Syst. Biol. 2011; 7: 539. https://doi.org/10.1038/msb.2011.75
- Banks J., Speidel E.C., McCauley J.W., Alexander D.J. Phylogenetic analysis of H7 haemagglutinin subtype influenza A viruses. Arch. Virol. 2000; 145(5): 1047–58. https://doi.org/10.1007/s007050050695
- Robinson D.F., Foulds L.R. Comparison of phylogenetic trees. Math. Biosci. 1981; 53(1): 131–47. https://doi.org/10.1016/0025- 5564(81)90043-2
- Webster R.G., Govorkova E.A. Continuing challenges in influenza. Ann. NY Acad. Sci. 2014; 1323(1): 115–39. https://doi.org/10.1111/ nyas.12462
- Chen L., Ruan F., Sun J., Chen H., Liu M., Zhou J., et al. Establishment of sandwich ELISA for detecting the H7 subtype influenza A virus. J. Med Virol. 2019; 91(6): 1168–71. https://doi.org/10.1002/jmv.25408
- Dong J., Fan J., Wang J., Zhang Q., Yang Y., Jia Y., et al. Development and evaluation of a C-ELISA for rapid detection of antibody to AIV-H7. Anal. Biochem. 2019; 572: 52–7. https://doi.org/10.1016/j. ab.2019.02.024
- Jadhao S.J., Achenbach J., Swayne D.E., Donis R., Cox N., Matsuoka Y. Development of Eurasian H7N7/PR8 high growth reassortant virus for clinical evaluation as an inactivated pandemic influenza vaccine. Vaccine. 2008; 26(14): 742–50. https://doi.org/10.1016/j. vaccine.2008.01.036
- Meseda C.A., Atukorale V., Soto J., Eichelberger M.C., Gao J., Wang W., et al. Immunogenicity and protection against influenza H7N3 in mice by modified vaccinia virus Ankara vectors expressing influenza virus hemagglutinin or neuraminidase. Sci. Rep. 2018; 8(1): 5364. https://doi.org/10.1038/s41598-018-23712-9
- Боголюбов А.С., Жданова О.В., Кравченко М.В. Справочник по орнитологии. Миграции птиц. М.: Экосистема; 2006.
Дополнительные файлы